單分子拴球實驗在生醫研究的應用
Single-Molecule Tethered Particle Motion
in Biomedical Research
單分子拴球實驗為一簡單量測蛋白質與 DNA 作用情形的單分子技術。利用高解析的光學顯微技術,我們 得以準確地觀測接連在 DNA 及玻片表面的乳膠小球之布朗運動情形,並藉由其布朗運動的大小直接對應 到在生化反應中 DNA 長度的變化,因此單分子拴球實驗已被廣泛地應用於研究單一蛋白質與單一 DNA 分 子作用之反應機制。我們更利用在乳膠小球上施加一微小伸展力來抑制其布朗運動大小,並藉此改良出施 力式拴球實驗。此施力設計不僅改善實驗之解析度,擴展可應用之 DNA 長度範圍,並得到一理論可預測 之 DNA 長度與布朗運動的線性關係。此施力式拴球實驗已被用來研究 RecBCD 解螺旋與 DNA 分子作用 情形,發現此解螺旋分子沿 DNA 移動時,藉由辨識 DNA 特殊序列來調控其性質,並表現出移動速率由 快轉慢之改變。陳晏誠、范秀芳、李弘文
Yen-Cheng Chen, Hsiu-Fang Fan, Hung-Wen Li
Tethered particle motion (TPM) method is a simple technique to study the protein-DNA interactions at the single molecule level. By directly imaging the Brownian motion of the polystyrene bead, which is tethered to the coverslip through a DNA molecule, TPM allows the real-time measurement of DNA length change during enzymatic processes. We developed a force TPM experiment to further improve the resolution of the measurement, to extend the DNA application range and to achieve a linear and predictable relation between DNA length and bead Brownian motion. This FTPM method has been used to study the motion of a DNA helicase, RecBCD, along individual duplex DNA molecules and provided insights on its protein-DNA interaction.
一、前言
近數十年的生物化學研究都致力於了解酵素的 反應機制,科學家希望藉由了解這些反應機制,提 供人類疾病治療的分子線索。一般生化實驗是一種 平均的實驗,即使在高靈敏度的偵測極限範圍,科 學家們所觀測到的現象還是大於 1010 個分子所平 均呈現的結果。然而每個生物分子並不是處在完全 相同的分子環境下,而且每個生化分子進行生化反 應的過程也不是完全同步,因此一般的生化實驗只 能提供一個平均行為的觀測,對於一些半生期短及 濃度低的反應中間物,無法提供靈敏的觀測資料。 舉例來說,如果一個酵素分子在同個條件下可以有 很多種狀態存在,那平均的生化實驗便無法精確地 說明其真實的狀態分布。如果我們可以一次只觀察 到一個酵素分子和其反應物的作用,那麼我們就有 單分子檢測 專題可能更加準確地知道同個條件下的多種狀態,或是 隨著時間推移,酵素的每一個反應步驟,進而提供 該酵素詳盡的反應分子機制。
二、單分子實驗
近年來顯微鏡技術的進步讓我們能夠以超高解 析度、靈敏度的方法進行測量。但若要直接觀測單 一生物分子,仍需要在目標物上面做進一步的標 定,將分子運動的訊號放大以便於觀測。這些常用 的標定方法,包括在生化分子上接一個螢光染料, 藉由螢光分子的高訊噪比,直接觀察單一螢光分子 的放光現象 (single molecule fluorescence) 來研究分 子間的結合 (圖 1(a)),或是利用一對螢光分子進行 分子間的能量轉移 (螢光共振能量轉換顯微術, fluorescence resonance energy transfer, FRET) (圖 1(b)),來得知生物分子在反應過程中的構形變化。 另外,還可以在生物分子上綁一個光學顯微鏡下可 觀測的大目標物,如微米大小的乳膠小球或是奈米 粒子,藉由觀察球的位置變化 (單分子軌跡,single particle tracking),來觀察生物分子的運動軌跡 (圖 1(c)), 或 是 單 分 子 拴 球 實 驗 (tethered particle motion, TPM),藉由觀察乳膠小球的布朗運動,而 得知生物分子在 DNA 上的位置。科學家們也可用 特定的力鑷作用,減少乳膠小球的布朗運動,達成 奈米級的解析度,來量測生化分子的運動,例如光 學鑷子 (optical tweezers) (圖 1(d)),或是施加一個 磁場對磁球施力的磁鑷子 (magnetic tweezers)。三、拴球實驗:量測單一 DNA 分子
長度
單分子拴球實驗是一個利用和 DNA 相連的乳 膠小球布朗運動大小,來測量 DNA 長度變化的技 術(1) 。在實驗上我們將單一個 DNA 分子,一端利 用抗原/抗體作用力固定在表面上,而另一端則和 圖 1. (a) 單分子螢光,(b) 螢光共振 能量轉換顯微術,(c) 單分子 軌跡,(d) 光學鑷子。 ड डЍ ၹݭតೱ ਕณ็ᅍ ਕณ็ᅍ ӣ̬ኳ۞̈ ֯ቱ̈ ̶̄ொજ ࡪቯ ྫ ඕЪ ᏈЍߖफ़ DNA ᅔ৵ (a) (c) (d) (b)一顆大小約為數百奈米大小的乳膠小球相連。在室 溫下及溶液狀態中,乳膠小球會受到周遭水分子的 不斷撞擊,而表現出布朗運動。因為乳膠小球相連 在 DNA 一端,布朗運動的大小就會受限於 DNA 的長度,就像是被拴在柵欄上的牛所能吃草的範圍 和拴牛的繩子長度有關一樣,DNA 越長,則球的 布朗運動越大 (如圖 2(a))。 在實驗過程中,利用光學顯微鏡的影像技術即 時的紀錄,並利用數位化影像系統來擷取球中心點 位置隨時間變化的情形,進而描述其布朗運動大 小。舉例來說,若擷取的每張畫面時間為 30 毫 秒,1 秒鐘內就可以得到 33 個畫面,而求得球中 心的位置會隨著時間而變化 (圖 2(b))。利用在不同 DNA 長度下所得到球布朗運動位置變化的標準 差,可以得到在短的 DNA 長度區間內 (< 2 kb) 布 朗 運 動 的 大 小 和 DNA 長 度 有 一 線 性 關 係 (圖 2(c))。因此我們可藉由校正圖 (圖 2(c)) 及實驗上所 觀察的拴球之布朗運動大小,來決定 DNA 長度隨 時間變化的情形。 與其他實驗方法相比較,拴球實驗的簡單方法 及直觀的量測方式已被廣泛地運用在研究酵素與 DNA 的作用上,如合成 RNA 的合成 (RNA polymerase, RNAP)、DNA 解螺旋、DNA 構型的 改變、DNA 限制和 DNA 重組等。只要酵素 與 DNA 的反應過程中有伴隨著 DNA 長度的變 化,都可利用單分子拴球實驗來量測。除了單分子 拴球實驗,也可利用力鑷 (tweezers) 實驗來研究酵 素和 DNA 的作用,一般力鑷實驗為了降低 DNA 在溶液中會晃動所產生雜訊,通常利用施力的方式 (> 4 pN) 來減低布朗運動所產生的雜訊,以求得較 佳的精確度。 拴球實驗提供在無外加力的環境條件來觀察生 物分子,與一般酵素在生物體內的環境相仿,和其 他高解析度的單分子力鑷實驗相比時,更可以提供 酵素在不同施加外力情況下的反應機制,讓科學家 們在研究上有不同的選擇,以符合真實生物體內的 環境。無可諱言,如前所述,因為拴球實驗是利用 觀察雜訊的大小來作為實驗上的訊號,在精準度上 會比較難突破,造成一定程度的侷限性,是其在應 用上需解決的問題,而我們也設計了新的實驗方法 來增加它的解析度。
四、應用在 DNA 解螺旋
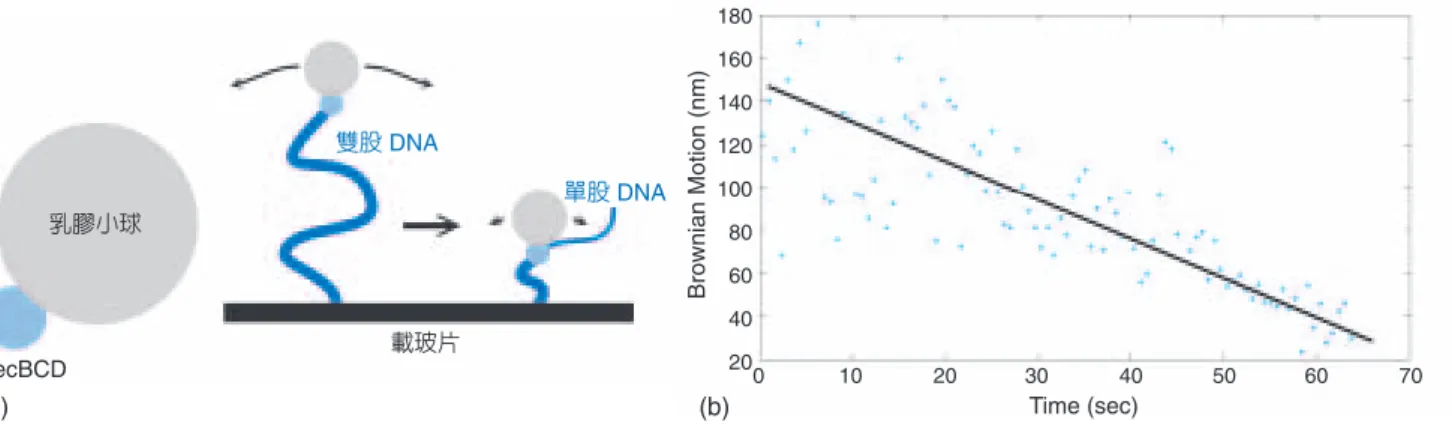
筆者的實驗室利用拴球實驗來直接觀測單一個 DNA 解螺旋 (helicase),將一個雙股 DNA 分子 解螺旋並在其 DNA 分子上移動。RecBCD 是在大 腸桿菌中的一個解螺旋,為 DNA 修復酵素組的 一員,當 DNA 雙股受到損壞而斷裂,RecBCD 會 辨認斷裂的 DNA 末端將其解螺旋,並啟動 DNA 重組 (DNA recombination) 修復的機制來修補受損 的 DNA。 RecBCD 同 時 具 有 解 螺 旋 和 分 解 (nuclease) 的功能,在修補的過程中,RecBCD 打 開 DNA 雙股往前移動,並配合其 C 端 (C-terminal) 的分解,將解開 DNA 的其中一條單股分解,再 配合其他重組酵素 (RecA) 的作用,重組 DNA 完 成修復。 在單分子拴球實驗中,我們在 RecBCD 上面 ྶࠟͯ ྶࠟͯ DNA DNA (a) (b) (c) 0 500 1000 1500 2000 150 120 90 60 30 0 DNA Length (bp) Brownian Motion (nm) 圖 2. (a) 拴球實驗示意圖,左上小圖中每一點代表球在每一畫面中的中心位置。(b) 各個時間點的影像,經 由數位影像處理找到球的中心位置 (紅色小點)。(c) DNA 長度和球位置變化的標準差實驗上呈一線性 關係。最近的研究指出 DNA 長度與乳膠小球之布朗 運動的關係,並非如實驗所觀測簡單的線性關係(2)
。 為了改善這個現象,並增加拴球實驗的解析度,我 們設計了施力式拴球實驗 (force tethered particle motion, FTPM),欲利用些微的施力來減少乳膠小 球的布朗運動,並增加拴球實驗的解析度。施力的 方式有好幾種,第一種是利用固定流速的水流加注 在乳膠小球所造成的伸展力 (stretching force)。由 熱力學上的能量均分定律 (equipartition theorem), 可知乳膠小球在垂直於施力方向的均方位移 (mean square displacement, MSD, ·∆x2Ò) 是和 DNA 的伸長 長度,l,成絕對的線性正比。 其中 F 是所受的力,T 是溫度(3) 。這個能量均分定 律清楚地說明乳膠小球的均方位移和 DNA 伸長長 度 (l) 及施力大小 (F) 的關係,因此在這個施力式 的拴球實驗中,不需經由實驗去求得 DNA 的長度 和布朗運動實驗關係式 (如圖 3(c)),只需知道受力 大小及溫度,便可決定其關係。由於拴球實驗的長 度判別解析度受到小球的布朗運動影響,當施加外 力時可減低其布朗運動程度,應該能夠藉此提高解 析度。 和橡皮筋一樣,DNA 的長度會受到施加外力 的影響,此現象可以用 worm-like chain 的實驗式描 述(4) : Dx k T F l B 2 = 利用抗原/抗體作用接上一顆乳膠小球 (圖 3(a)), 並在玻片表面固定一段雙股 DNA 分子。在溶液中 RecBCD 會擴散,並辨認出受損的 DNA 雙股末端 進行結合,在能量的供給 (ATP) 下,RecBCD 即會 進行解螺旋的反應,解開雙股 DNA 向前移動。而 我們便可經由 RecBCD 上所接的乳膠小球之布朗 運動的變化,得知單一個 RecBCD 分子在 DNA 上 的位置隨時間變化的情形。如圖 3(b) 所示,觀察 球的布朗運動隨著時間的推移而有所減少,表示 RecBCD 在 DNA 的位置是由上向下移動,使得 DNA 越來越短,而圖 3 中的斜率即是 RecBCD 在 DNA 上的移動速率。在給予相同能量時 (相同 ATP 濃度),單分子拴球實驗所量測的解螺旋速度和一 般生化平均實驗所量測的速率相似,這表示在拴球 實驗的設計中,乳膠小球及玻片表面不會影響 RecBCD 的工作效率,也指出單分子的確可用來真 實地描述酵素的工作機制。
五、施力式拴球實驗
如前所述拴球實驗中 DNA 長度的換算,是利 用乳膠小球之布朗運動程度在有限範圍內與 DNA 長 度成正比的實驗關係式。然而當 DNA 的長度過長 (> 2 kb),導致乳膠小球的運動範圍過大,拴球實驗 便無法精準地運用球的晃動大小來換算成相對應的 DNA 長度。除此之外,目前並無完整理論可以解 釋 DNA 長度與乳膠小球布朗運動之線性關係。(a) (b) Time (sec)
Brownian Motion (nm)
圖 3. (a) 左圖,RecBCD 上面接一顆乳膠小球。右圖,進行實驗時,RecBCD 會尋找雙股 DNA 末端,找到 之後在 ATP 供給下便會開始向前移動。(b) 以拴球實驗觀察 RecBCD 的酵素作用,可以看見球的布朗 運動隨著時間變小,也就是 RecBCD 有向前移動,使 DNA 長度變短。 ֯ቱ̈ ྶࠟͯ ᗕ۵ DNA RecBCD ಏ۵ DNA 0 10 20 30 40 50 60 70 180 160 140 120 100 80 60 40 20
其中 LP 為抗彎長度 (persistence length),L0為輪廓
總長 (contour length),l 為 DNA 之伸長長度 (extension length)。由於 DNA 的輪廓總長正比於 DNA 內含之鹼基對數目 (已知雙股 DNA 10 bp,螺 距為 3.4 nm),所以在同一施力條件下,DNA 之伸 長長度與其所含之鹼基對數目成正比。所以在施力 式的拴球實驗中,乳膠小球在垂直施力方向的均方 位移與 DNA 內含鹼基對數目呈線性正比。如圖 4 所示,在同一 DNA 長度下所加的外力越大,乳膠 小球的布朗運動越小,而所量測的均方位移之準確 度越好 (誤差範圍小)。然而當所受外力越大時,其 斜率就如理論所預測的變小 (正比於 kBT/F)。該斜 率變小的趨勢指出在不同 DNA 長度下,其均方位 移的變化量很小,亦即在不同 DNA 長度下,均方 位移的值都差不多。 由於拴球實驗的解析度同時決定於均方位移量 測的精確度及用均方位移值區分不同長度 DNA 的 分辨力 (斜率),所以拴球實驗的最佳解析度不會是 在施加外力很大 (沒有分辨力) 或很小 (沒有精確 度) 的情況下。利用圖的斜率和誤差範圍的比例, F k T L l L l L B P =
-(
)
+ -È Î Í Í Í ˘ ˚ ˙ ˙ ˙ 1 4 1 1 4 0 2 0 可決定出實驗條件下最佳的解析度是在 0.2 pN (10–12牛頓) 施力下所達成。 如果比較 0.2 pN 微小施加外力的情況下與一 般無施加外力的拴球實驗的解析度,發現施力式拴 球實驗可提高 2 至 4 倍的解析度 (如表 1 所示),並 可擴展 DNA 長度的範圍至 4 kb 以上(5) 。六、RecBCD 解螺旋
水流方式的施力不僅增加偵測的解析度,並擴 展應用範圍,利用此施力式拴球實驗,我們能夠更 仔細地研究 RecBCD 酵素的作用機制。RecBCD 負 責在大腸桿菌內的 DNA 損壞之修補,是由三個蛋 白質單體所組成的三質體複合蛋白,其中 RecB 具 有 3¢ → 5¢ 到解螺旋特性,而 RecD 具有 5¢ → 3¢ 端 的解螺旋特性,而 RecC 則被認為是負責擔任辨識 特定序列 chi (5¢-GCTGGTGG-3¢) 及調控蛋白之功 能。 目前研究發現 RecBCD 成功地辨識特定序列 chi 之後,可進而調控其解螺旋之特性。早期利用 非施力式的拴球實驗研究在 RecBCD 及少量的 ATP 濃度下,發現 RecBCD 可以成功的辨識特定 序列 chi,但並無任何速率變化情形(6) 。現在利用 施力式拴球實驗,在增高解析度的情況下,再次檢 測 RecBCD 於低濃度 ATP 下的運動情形。如圖 5 所 示 , 可 看 到 在 特 殊 的 DNA 序 列 chi 之 前 RecBCD 移動的速度比較快,在 chi 序列稍作停留 後,RecBCD 的速度就會明顯變緩。這個現象可以 在三十幾個 RecBCD 分子解螺旋的過程中,被我 們 的 施 力 式 拴 球 實 驗 所 觀 測 到 , 成 功 地 印 證 RecBCD 對於 chi 序列的辨別是不受 ATP 調控的。0 1000 2000 3000 4000 0 5000 10000 15000 20000 25000 30000 35000
Mean Square Displacement (n
m 2 ) Tether Length (bp) 圖 4. 均方位移對 DNA 鹼基對數目做圖,在不同施 力情況下呈現線性關係,其斜率反比於施力 大小。■= 0.06 ± 0.02 pN;□= 0.21 ± 0.06 pN; ▽= 0.86 ± 0.21 pN;◇= 14.03 ± 4.55 pN。 DNA 長度 解析度 (base pair) F = 0.2 pN F = 0 pN (FTPM) TPM 1893 330 681 4190 422 1771 表 1. 比較在 0.2 pN 微小施加外力的情況下與一般 無施加外力的拴球實驗的解析度。
水流式的施力拴球實驗已成功地應用於生化研 究上,不過提供一個穩定水流會需要大量的生物樣 品,面對比較難取得的昂貴生物分子就會有困難。 我們正在利用具磁性的乳膠小球取代目前的乳膠小 球,並利用強力磁鐵在側邊對磁球施力,造成和水 流式施力拴球實驗一樣的延展力 (如圖 5(c))。該實 驗設計不但可維持和水流式一樣的精確度,也可去 除因固定流速不穩定而造成的訊號擾動,更不需大 量生物樣品,可以應用在研究更多種酵素分子的機 制上。
七、結論
單分子實驗提供科學家們更詳細地由分子層面 去觀察生物的現象。藉由這些實驗可偵測生物分子 的異質性和其快速的構型變化。拴球實驗是個方便 操作的單分子實驗技術,其運用球的布朗運動大小 換算成 DNA 的長度,提供不需施力的選擇。經由 施加一個微弱的力 (< 0.2 pN),便可提升精確度, 讓拴球實驗有更好的應用範圍。未來的研究中,除 了闡明未知的生物作用外,提高單分子實驗的精確 度不光是拴球實驗,對於所有的實驗技術都是一個 可向前邁進的目標。參考文獻
1. H. Yin, R. Landick, and J. Gelles, Biophys. J., 67, 2468 (1994). 2. P. C. Nelson, C. Zurla, D. Brogioli, J. F. Beausang, L. Finzi,
and D. Dunlap, J. Phys. Chem., 110, 17260 (2006)
3. T. R. Strick, J. F. Allemand, D. Bensimon, and V. Croquette,
Biophys. J., 74, 2016 (1998).
4. C. G. Baumann, S. B. Smith, V. A. Bloomfield, and C. Bustamante, PNAS, 94, 6185 (1997).
5. H. F. Fan and H. W. Li, Biophys. J., 96, 1875 (2009) 6. K. M. Dohoney and J. Gelles, Nature, 409, 370 (2001).
‧陳晏誠先生目前就讀於國立台灣大學化學系。 ‧范秀芳小姐為國立台灣大學化學博士,現任國立台灣
大學化學研究所博士後研究員。
‧李弘文先生為美國加州大學柏克萊分校化學博士,現 任國立台灣大學化學系副教授。
‧Yen-Cheng Chen is currently a student in the Department of Chemistry at National Taiwan University.
‧Hsiu-Fang Fan received her Ph.D. in chemistry from National Taiwan University. She is currently a postdoctoral fellow in the Department of Chemistry at National Taiwan University. ‧Hung-Wen Li received his Ph.D. in Chemistry from the University of California at Berkeley, USA. He is currently an associate professor in the Department of Chemistry at National Taiwan University.
圖 5. (a) 水流式拴球實驗。(b) 在水流式拴球實驗所看到的 RecBCD 在 DNA 上移動的速率變化,可以分辨 在有 chi 序列的情況下,會有速度快-停頓-速度慢的變化趨勢。(c) 磁力式拴球實驗。 (a) (c) (b) z y x F N S Dx z y x Dx 0 20 40 60 80 100 120 140 150 80 70 60 50 40 30 20 10 Time (sec) Brownian Motion (nm)