行政院國家科學委員會專題研究計畫 期中進度報告
利用基因體技術研發特殊用途性質稻米品系(1/3)
計畫類別: 個別型計畫 計畫編號: NSC93-2317-B-002-008- 執行期間: 93 年 08 月 01 日至 94 年 07 月 31 日 執行單位: 國立臺灣大學農藝學系暨研究所 計畫主持人: 盧虎生 計畫參與人員: 老嘉玲 報告類型: 精簡報告 報告附件: 出席國際會議研究心得報告及發表論文 處理方式: 本計畫可公開查詢中 華 民 國 94 年 5 月 31 日
(一)中文摘要 稻作目前為我國生產面積與產值最大之農作物,自2002年加入WTO後面臨國際市場的競 爭壓力下,我國良質米的市場發展受到相當的限制。解決策略之一是研發具特殊用途品質的 品種,以開發多樣化、多用途品質的市場,增進我國稻作產業的競爭力。農業試驗所嘉義分 所近年來利用台梗9號及台農67號發展出數個高世代、已具遺傳穩定性的誘變族群,初步分析 顯示這些族群品在蛋白質、澱粉等分子上具有相當變異性,具有開發特殊性質品種的潛力。 本計畫擬以NIR方法初步篩選不同粗蛋白質與直鏈澱粉含量之品系,利用proteomics進一步分 析選拔具特殊蛋白質之品系,同時進行農藝性狀評估、田間栽培適應性評估,與利用價值評 估,期望能選出具特殊性狀且具利用潛力之新品系。本研究計畫目前已建立有效之水稻蛋白 質特性篩選方法與流程,以此方法篩選所收集之水稻品系。經篩選近330個不同性質之誘變族 群、野生種及傳統雜交組合後代高世代品系,已篩選出約30個高蛋白及30個低蛋白品系,並 由野生稻品系中發現多型性表現,及栽培稻與野生稻間的差異表現。由此些材料族群篩選出 之品系已進入今年一期作栽培試驗,將持續進行後代蛋白質表現穩定性測定及農藝性狀評 估 。 對 於 其 中 特 殊 之 品 系 , 目 前 正 進 行 2D 電 泳 及 蛋 白 質 identification 分 析 (Q-TOF 或 LC/MS/MS), 以進一步瞭解其稻米蛋白質分子功能特性。 關鍵詞:水稻,品質育種,蛋白質體學,特殊品質 Abstrate
Rice is the largest cultivated crop on Taiwan. After entering WTO, breeding for quality improvement has been facing a bitter challenge due to importation of high quality rice from international rice market. A promising solution for the challenge is to create new varieties with novel characteristics and added market value. In recent years Agriculture Research Institute has developed several chemical induced mutation populations. These mutation populations are genetically stable and contain considerable variation in macro biomolecules content, such as proteins and amylase. In this project, we will use NIR spectroscopy to screen mutation lines having significant variation in protein and amylase contents. And then determine protein specificity and identity for selected genetic lines by proteomics. Agronomic traits of selected lines and their adaptability for organic culture system are further evaluated in the field. We have set up the effective SDS-PAGE procedure to screen mutation lines. And have screened 30 high protein and 30 low protein content lines from 330 different genetic lines, including mutation population, wild type and traditional cross-over breeding lines. In addition, there are a certain polymorphism in protein profiles between wild type lines and cultivars. The selected lines have planted for the following trials of agronomic traits and protein expression stability. Further more, we will analyze the protein function of some special lines by 2D electrophoresis and protein identification analysis.
(二)前言 稻作目前為我國生產面積與產值最大之農作物,自 1980 年代以來台灣的稻作發展已由 產量的改良轉型為品質的提升,因而發產良質米育種計畫。自 2002 年加入 WTO 後面臨國際 市場競爭下,我國良質米的市場發展受到相當的限制,解決的策略之一為研發具特殊用途品 質的品種,以開發多樣化的市場,增進稻米的市場領域與價值。相較於國際之品質育種,日 本在食味品質育種上居世界領先,以越光品種最具代表性。但為增加其稻米之利用性與市場 價值,早自 1980 年代起,日本已進行”新形質米”研發計畫,目前已育成包括直鏈澱粉含量品 種、低過敏蛋白質品種、低 glutelin 蛋白質品種,有色米、大胚米、香米等等具經濟市場價值 之品種,已在田間試種甚至在市場推出,此些品種稻米價格顯著高於一般良質米品種,具有 相當之市場競爭力。遺憾的是,此些特殊品種之種子在智財權保護下外界難以獲得,無法將 其特殊性狀導入我國育種計畫中。 已知稻米蛋白質與食味品質有關,高蛋白質品種可應用於營養食品加工,低蛋白質品種 適用於釀酒(Shizukawa et al., 2002),特殊品種適用於特別的消費族群,而誘變育種可提供特 殊的基因功能。國內農業試驗所歷年來嘗試以誘變創造新的性狀來源,嘉義分所之吳永培博 士近年來以化學誘變法,以產生數組高世代遺傳性狀穩定之品系族群,包括低蛋白質含量、 香米、糯性及低直鏈澱粉含量等具有相當變異性之族群。配合近遠紅外光分析儀(NIR)快 速分析技術,可對蛋白質進行實際育種上的分析和操作,並提高選拔效率(Villareal et al., 1994;Delwiche et al., 1996),唯對此些蛋白質的生理功能所知則相當有限。 近年來植物功能性基因組 functional genomics 的研究中,蛋白質體研究上也發展出 proteomics 的觀念,及利用更精細之 2D 雙向電泳及 LC/MS/MS 或 Q-TOF 等定序技術,可篩 選出在某發育生理作用或對環境反應過程中差異表現(differential expressed)的蛋白質,並依 其序列推論它們的功能。本研究室目前已建立各種儲藏性蛋白質的分析方法,包括電泳及逆 向色層分析(RP-HPLC),亦建立多個品種稻米蛋白質圖譜,並確認此圖譜上 50 點以上之蛋 白質種類,包括合成直鏈澱粉之主要酵素及主要儲存性蛋白質 prolamin 及 glutelin 群分子(Lin
et al., 2003)。本研究室擬利用此已建立的蛋白質研究技術,篩選前述之誘變族群,期望能篩 選出具特殊蛋白質性質之品系,開發出新的基因型(育種材料)或品種。 (三)研究目的 本計畫即利用本研究室已建立之蛋白質體分析方法,篩選高世代已具遺傳穩定性的誘變 品系(非轉殖)或傳統優良雜交組合後代,並配合田間農藝性狀的評估,期望開發具特殊性 狀之基因型,研發具特殊成分之優質稻米品系,以提昇我國稻作育種與產業之競爭力。基於 本研究室現有的技術與人力有限,本計畫將以蛋白質性狀篩選為主。本年計畫目的為收集不 同來源及特性之水稻品系,建立有效之水稻蛋白質特性篩選方法與流程,以此方法分析所收 集之水稻品系之蛋白質變異性,篩選出具顯著蛋白質差異之品系。
(四)研究方法
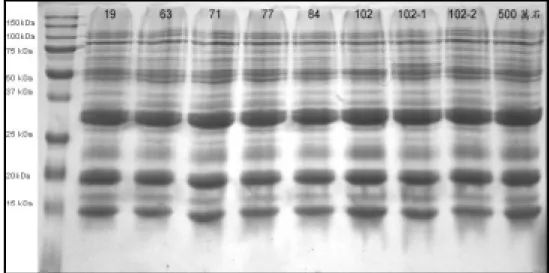
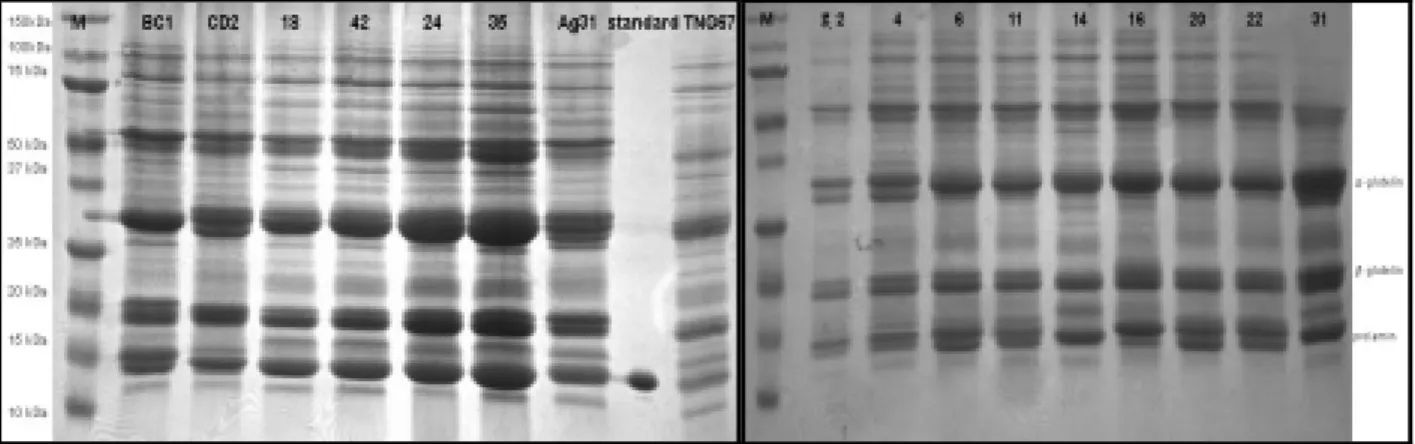
本研究室已建立基因體技術中之稻米 Proteomics 分析技術、配合 NIR(Near Infared Spectroscopy)非破壞性稻米成分分析技術,可有效篩選含特殊蛋白質基因表現之產物或成分 的遺傳材料。此外,我國農試單位如農業試驗所嘉義試驗分所及各改良場近年來利用化學誘 變或特殊親本雜交、已獲得不少具遺傳穩定性的品系族群(非轉殖),初步分析顯示這些族群 品系在稻米外表型、蛋白質、澱粉等分子上具有相當變異性,具有開發特殊性質品種的潛力。 本計畫擬先以稻米外表型與 NIR 方法初步篩選不同外表型、粗蛋白質與直鏈澱粉含量之品 系,再利用 proteomics(SDS-PAGE 單向及雙向電泳分析)進一步分析選拔具特殊蛋白質表現 之品系,同時進行後代蛋白質表現穩定性、農藝性狀評估、及田間栽培適應性評估、與利用 價值評估等,期望能選出具特殊性狀且具利用潛力之新品系。 (五)結果與討論 本年度(第一年)計畫以篩選各農業試驗單位之水稻遺傳育種族群品系為主要目標,目前執 行情形與進度如下: 1.以酵素 lysozyme 建立蛋白質含量標準曲線,結果如圖 1 所示,蛋白質濃度與面積呈線性相 關,R-square 值為 0.98,因此確定此標準曲線可應用於檢定稻米儲藏性蛋白質( glutelins 及 prolamins )之品質,評估育種或遺傳材料之研發利用性(如營養或製酒適性)。 2.利用農試所嘉義分所之疊氮化鈉誘變族群及低蛋白雜交組合後代(F5 以上之高世代),先經 NIR 方法初步篩選出具蛋白質變異、糯性及半糯性共 152 個品系,再經 SDS-PAGE 電泳分 析。以台粳九號為對照組,共選出 8 個高蛋白質品系及 18 個低蛋白質品系。如圖 2 所示, 編號 67,72,105,106 及 160 為低蛋白質品系,編號 156,171 及 190 為高蛋白質品系。 3.利用花蓮農業改良場之紅糯(光香純)、及進入高級試驗及初級試驗共 89 個雜交育種後代 之品系,再經由 SDS-PAGE 電泳選出 10 個高蛋白質品系及 8 個低蛋白質品系。結果如圖 3 所示,並經由軟體計算 PBII/PBI 比值,得知日本知名酒米品種五百萬石的比值為 3.17,而 紅糯品系多為 3.45∼5.13,略高於五百萬石。 4. 利 用 苗 栗 農 業 改 良 場 經 外 觀 初 步 篩 選 穀 粒 具 心 腹 白 之 50 個 雜 交 育 種 品 系 , 再 經 由 SDS-PAGE 電泳選出 7 個高蛋白質品系及 6 個低蛋白質品系。結果如圖 4 所示,編號 6,7 及 8 為高蛋白質品系,編號 4,11 及 12 為低蛋白質品系。 5.利用高雄農業改良場初步篩選 44 個野生稻品系,再經由 SDS-PAGE 電泳分析,以台農 67 號(TNG67)為對照組,並以 2ug 標準蛋白質(濃度 2 ug/ul 之酵素 lysozyme 注入 10 ul), 進行蛋白質定量。共選出 30 個高蛋白質品系,另有 13 個品系在 glutelins 及 prolamins 有顯 著之多型性表現,結果如圖 5 所示。
系,開發出新的基因型。本研究內容與原訂計畫目標一致,本試驗為三年期研究計畫之第一 年期,目前已建立有效之水稻蛋白質特性篩選方法與流程,以此方法篩選所收集之水稻品系。 篩選近 330 個不同性質之誘變族群、野生種及傳統雜交組合後代高世代品系,已篩選出約 30 個高蛋白及 30 個低蛋白品系,並由野生稻品系發現多型性表現,及栽培稻與野生稻間的差異 表現。由此些材料族群篩選出之品系已進入今年一期作栽培試驗,將持續進行後代蛋白質表 現穩定性測定及農藝性狀評估。對於其中特殊之品系,目前正進行 2D 電泳及蛋白質 identification 分析(Q-TOF 或 LC/MS/MS), 以進一步瞭解其稻米蛋白質分子功能特性。 本計畫預計在第二年建立水稻糊粉層蛋白質單向電泳之蛋白質圖譜,配合本研究室已建立 之水稻穀粒蛋白質雙向電泳,建立稻米蛋白質品質之蛋白質體(proteomic)檢定系統。並將 進入栽培試驗的水稻品系進行蛋白質表現之再次確認,同時篩選更多的水稻品系,以期獲得 具特殊成分與市場經濟潛力之新品系。 (七)參考文獻
Delwiche, S.R., K.S. Mckenzie and B.D. Webb. 1996. Quality characteristics in rice by near-infrared reflectance analysis of whole-grain milled samples. Cereal Chem. 73: 257-263. Lin, S.K., M.C. Chang, Y.G. Tsai and H.S. Lur. 2003. Proteomic analysis of the expression of
quality related proteins during rice caryopsis development and its response to high temperature. 2003 International Symposium on Plant Functional Genomics.
Shizukawa, Y., Y. Oohashi., T. Masumura. and K. Tanaka. 2002. The contents and distribution of prolamin, glutelin and globulin that constitute storage protein ( protein body ) in brown rice on some rice cultivars for brewery or meal. Jpn. J. Crop. Sci., 71: 224-225 ( in Japanese )
Villareal, C.P., D.L. Cruz, N.M., and B. O. Juliano. 1994. Rice amylase analysis by near-infrared transmittance spectroscopy. Cereal Chem. 71: 292-296.
圖 1. 以酵素 lysozyme 建立稻米儲藏性蛋白質含量檢量標準曲線 圖 2. 農試所嘉義分所以疊氮化鈉誘變族群及低蛋白雜交組合後代水稻品系之蛋白質 單向電泳分析結果。 抽取不同水稻品種之穀粒蛋白質,進行單向電泳分析。膠體濃度為 13.5 %,每個齒槽加入 20 μl 總蛋白質,並以台粳九號(TK9)為對照組。 y = 0.1655x + 0.052 R2 = 0.9815 0 0.05 0.1 0.15 0.2 0.25 0 0.2 0.4 0.6 0.8 1 1.2 protein concentration (mg/ml) ar ea
圖 3. 花蓮農業改良場紅糯品系與五百萬石之蛋白質單向電泳分析結果 抽取不同水稻品種之穀粒蛋白質,進行單向電泳分析。膠體濃度為 13.5 %,每個齒槽加入 20 μl 總蛋白質,並以五百萬石為對照組。 圖 4. 苗栗農業改良場具心腹白之雜交育種品系之蛋白質單向電泳分析結果 抽取不同水稻品種之穀粒蛋白質,進行單向電泳分析。膠體濃度為 13.5 %,每個齒槽加入 20 μl 總蛋白質。
圖 3. 高雄農業改良場野生稻品系之蛋白質單向電泳分析結果
抽取不同水稻品種之穀粒蛋白質,進行單向電泳分析。膠體濃度為 13.5 %,每個齒槽加入 10 μl 總蛋白質,以台農 67 號(TNG67)為對照組,並以 2ug (濃度 2 ug/ul 之酵素 lysozyme 注入 10 ul)為標準,進行蛋白質定量。