台灣地區溪狡蛛(蜘蛛目:跑蛛科)族群遺傳結構及親緣地理闕係之
探討 羅英元陳世煌* 國立臺灣師範大學生命科學系 (收稿日期: 2011.9.15 '接受日期: 2012.2.1 ) 摘要 台灣的中央山脈縱貫南北、地形高聳複雜,為台灣島內物種分布的天然地理屏障,一般認為節 肢動物如昆蟲和蜘蛛等具有良好的擴散能力,族群間的交流不易被地形所阻隔。然而徘徊性的溪狡 蛛(Dolomedes raptor) 生活棲所侷限於池塘或溪潤等有水的潮濕、環境,若蛛未見遷絲、空飄的行為,擴 散能力不佳,其族群分化模式是否與結網性的蜘蛛不同,有待進一步探討。本研究利用粒線體 cytochrome oxidase subunit I(CO1) 基因部分序列探討溪狡蛛之族群遺傳結構,總共分析來自臺灣地區 21 個採樣點、共 153 隻個體。獲得 CO I 基因部分序列長度 617坤,包括 34 個變異位點、 23 個基因型。由 AMOVA 分析得知台灣地區溪狡蛛的遺傳結構在族群間有顯著的差異,且
neighbor-joining 以及 maximum parsimony 親緣關條樹型圖皆顯示台灣地區溪狡蛛可分為北部、中南
部和東部三大~群。族群問遺傳分化拈數則顯示台灣地區溪狡蛛族群間基因交流會受到山脈和水車 的阻隔,具有「區域性分化」的現象,其分化模式和具有良好擴散能力的人面蜘蛛截然不同。而中 部和南部族群間的基因交流則較為順暢,由於中部和南部是台灣最主要的農耕區,地勢平坦,推測
溪狡蛛可利用人工開鑿的渠道,在各大水~間進出有關。;美狡蛛在台灣東部較為罕見,大多分布在
中央山脈以西的山區,且和生活習性相似的褐腹狡蛛 (Dolomedes mizhoanus) 少有共域棲息的現象, minimum spanningnetwork 也顯示東部溪狡蛛族群是由北部擴散而來,且遺傳多樣性較低,推測:美
狡蛛較不適應東部鴻急的溪流,其族群可能曾受到強烈瓶頸效應的影響;或可能是族群不易擴散到 東部,東部族群受到創始者效應、強烈還汰的結果,仍需更進一步的調查研究。 開鐘個: ~:美狡蛛、跑蛛料、基因交流、族群遺傳結構、親緣地理學 緒言 ;真技蛛 (Dolomedes raptor) 在分類上屬於跑 蛛料(Pisauridae)、狡蛛屬(Dolomedes) , 為一 中大型蜘蛛'雌蛛體長約23 ~27mm 、雄蛛約 12 ~ 15 mm' 全身一位為黑褐色,雌蛛第一、二對 步足蹄、節通常有白色斑紋,頭胸部兩側部分個體 帶有橘黃色、縱帶;雄蛛身體兩側有白色、縱帶,特 別是頭胸部兩側最為發達 (Chen ,2001 ) ,而若蛛 身上常有多個白色斑點,身體顏色較成蛛鮮豔。 廣泛分布於台灣海拔約 1000 公尺以下的中低海 拔山區,以及中國大陸、日本和韓國等地區。溪 狡蛛為徘徊型夜行性蜘蛛'棲息地侷限在池塘和 溪洞等有水的環境,喜好停棲於石頭上或岸邊草 叢下,常將第一對步足搭在水面上捕食落水的昆 色,雌蛛產下卵囊後會以聲肢( chelicera) 攜帶 卵囊,直到卵孵化、若蛛全部離開卵囊為止,展 現強烈的護卵行為,離開卵囊的若蛛也未發現有 牽 J是在空飄( ballooning) 的行為。 ;美狡蛛之棲息環境侷限、無邊絲、空飄行為 等,都顯示其擴散能力可能不如金蛛科 ( Araneidae)、長腳蛛科(Te甘agnathidae)或姐 蛛料( Theridiidae) 等結網性蜘蛛。由於物種本 身的擴散能力,為其能否越過地理屏障、影響族 群間基因交流的重要因子之一。一位認為,昆蟲 和蜘蛛可藉由自由飛行或風力進行長距離移 動,其有較優秀的擴散能力,因此較不容易受到 地理阻隔的干擾,使得族群間的基因交流較為順
暢 (Slatkin, 1985 ; Roderick,1996 ; Nibouche et叫, 1998 ; Ramirez&Haakonsen, 1999) 。在台灣,由
貫南北、地勢高聳複雜、加上東西走向為主的河 流切割,已有許多報告指出如此複雜的地形對於 台灣許多物種而言都是項天然的地理屏障。相反
的,白粉蝶 (Pierisrapae crucivora) 則因具有良
好的飛行能力使其族群間交流順暢(Lee, 2004) ,人面蜘蛛 (Nephi!a pi!抄es) 亦可能藉由 風力以遷絲空飄方式來進行長距離移動,較不易 受到中央山脈等地理阻隔的干擾,而未有顯著族 群分化的現象 (Lee et al., 2004) 。但除了人面蜘 蛛之外,關於台灣蜘蛛的行為、生態、族群遺傳 等研究仍相當缺乏。台灣島內複雜的地形對於一 些擴散能力較差的蜘蛛會受到何種程度的阻 隔、以及呈現何種分化模式等問題,都亟待進一 步研究。 另外,由於;美狡蛛一生都棲息在水邊,對環 境的選擇較侷限,推測族群之間需要有連接的水 系作為其交流的管道,而不易靠著直接越過陸地 來進行擴散,因此除了中央山脈之外,流域和流 域之間的陸地或許也是溪狡蛛重要的隔離因 子,和一般陸生節肢動物有所不同。綜合以上溪 狡蛛的特性,可作為探討台灣地形對擴散能力不 佳的蜘蛛在族群遺傳結構上有何影響之良好材 料。 由於蜘蛛的外部形態特徵變異非常細微,在 探討種內族群變異時,常有許多問題存在,但近 年來分子生物技術越趨成熟,能夠對許多傳統外 部形態分析上所無法解決的問題,提供另一有力 的解答,在國外已有許多利用分子生物學技術來 探討蜘蛛類親緣關條、族群遺傳結構、生物地理
學等的研究(例如 Gillespieet al.,1994; Fanget瓜, 2000 ; Colganet al.,2002) 。其中粒線體 DNA 中
的細胞色素氧化晦 I (COl) 基因在大部份無脊 椎動物中具有相當保守的引子,且演化速度快、 具有高變異度,在許多進行無脊椎動物種內變異 的研究時可提供足夠的解析度,是一個很適合的 分子標記(Simonet al., 1994) 。目前已有許多關 於蜘蛛族群遺傳變異和親緣關餘的研究皆指出
COl 基因是一個合適的標記(Hedin,2001 ; Hedin
& Maddison, 2001 ; Garb et al., 2004 ; Vink &
Paterson,2003)。故本研究擬選用 COl 基因,當 作探討溪狡蛛族群遺傳變異的分子遺傳標記。 材料與方法 樣本採集 於 2004~2007 年間,根據台灣地區的河川流
22bk叫阿
Tsengwen River Erhjen River 'Q, 圖一、台灣地區溪狡蛛採樣點之位置,其樣點編號如 表一所示。Figure1.Sample locations ofDolomedes raptorin this study. Location numbers 1-21 refer table 1.
域分佈,分別在淡水河流域、竹安溪流域、後龍 溪流域、濁水溪流域、急水漢、曾文溪流域、高 屏溪(楠梓仙溪)流域以及秀姑巒溪流域內各選 取 1--7 個採樣點,總共 21 個樣點(圖一)分別 進行採樣,每個樣點採集 1--20 隻不等的;其狡蛛 (Dolomedes raptor) 個體(表一) ,並採集同屬 之褐腹狡蛛 (D. mizhoanus) 和赤條狡蛛 (D. saganus) 做為多個外群進行分析。獲得之蜘蛛樣 本帶回實驗室,取下 1--2 隻步足進行 DNA 革取 (但每對步足至少保留一隻腳),其餘部分則浸 泡於 70% 酒精內保存,以供日後形質測量之用。 DNA 萃取、 peR 及定序
DNA 萃取方法參考 modified LiCI method
( Gemme1 & Akiyama, 1996) 並加以適當修改。
PCR 反應所使用之引子像參考 Folmer et al.
( 1994) 的 C1-J-1490 力口以修飾 (C1-J-1477 : 5'-ATAAA GATAT TGGAA CTTTA TATTT-3'),以
及 Simonet al. (1994)的 C1-N-2191 (5'-CCCGG
TAAAA TTAAA ATATA AACTT C-3') , 來擴增
COl 基因部分片段。 PCR 的反應程序為:(l)
以 95°C 、 4 分鐘,使雙股 DNA 完全變性解開 ( denature) , (2) 進行的次的增幅循環,每一
表-、溪狡蛛之採樣點、樣點編號、個體數(N)、基因型數(Nhap) 、核苦酸歧異度(π)及基因型歧異度 (h) 。 Table1.List of sample locations,location number,sample size(N),number of haplotype(Nhap),nucleotide diversity (π)and haplotype diversity(的 ofDolomedes raptorin Taiwan.
Loca位on N Nhapπh 1.PaomaTrail 跑馬古道 2 1 0 0 2.Wutuku 烏塗窟 10 5 0.00155 0.66667 3.Pinghsi 平溪 6 1 0 0 4.Tsaonan 草浦 4 1 0 0 5.Yinghotung 銀河洞 5 2 0.00065 0.4 6.Szukanshui 四炭水 7 2 0.00093 0.28571 7.Sanming 三民 2 2 0.00162 1 8.Kueihui 奎輝 9 2 0.00036 0.22222 --型orth PI哩些血旦1!::.~巨型io且.~-_!.::~---一--一一一一一-一一一-一一一一-一一-一一--一一一一一一-一一一一一-一一一P一一一-一一-一-一一一-一4笠哇 9. Gon邵伊l組公館 15 3 0.00960 O.后64盯76位2 10.Dahu 大湖 4 3 0.00972 0.83333 一位型哩!三型~_~!!!X~.p-哩latio且-(~虫"PEP-9三!_91___一一一一一一一一些一一一一 4 一一一一 o.空空空'31 一一一一一一一峭壁哩 2一一一一一一 11.Dakeng 大坑 6 2 0.00108 0.33333 12.Jongsingsintsun 中興新村 10 4 0.00447 0.64444 13.Chiuchiufeng 九九峰 6 4 0.00443 0.8 14.Taomikeng 桃米坑 7 4 0.00617 0.71429 一£EE1rHB且1哩io且一些空空空tio且已去的一一一一一一一一-一一一些一一一一旦一一一一一 -mppzp一一---一些 77哩哇,一一一-15.Kua刮目ling 關子嶺 20 7 0.00452 0.85789 16.Shuangsi 雙溪 12 4 0.00508 0.65152 17.Gueidan 龜丹 6 3 0.00540 0.6 18.Muguakeng 木瓜坑 12 5 0.00526 0.72727 19.Hean 和安 3 2 0.00648 0.66667 S'哩血 Po血!妞"姓些型!哩哇 1空出!_~---一一一一一一一一一豆豆 -EE---IPE--一一一些企堅;90 一一一一一些79681一一-20.Loshan 羅山 1 1 21.Antung 安通 6 2 0.00054 0.33333 East Population (Locations 20-21) 7 2 0.00046 0.28571 開;但℃、 30 秒使 DNA 與引子鍊和( annealing) ;
nOe 、 1 分鐘進行 DNA 延伸反應(extension) ( 3 ) 進行的個循環之後,再進行 nOe 、 7 分鐘的最 後延伸反應(final Extension),即得到最終的 PRC 增幅產物。 PCR 產物於1.2%瓊脂膠片上以 100V 進行電泳 30 分鐘,梭測增幅的PCR 產物的品質 和片段長度,確定此片段無誤後,進行DNA 定 序。 資料處理和分析 將定序後之序列以 Sequencher 軟體 (SequencherTM3.0 版)進行比對以及排序。以 DnaSP4.l0軟體(Rozas& Rozas,1999 ; Rozas et
al.,2003) 進行 Tajima'sD test (Tajima, 1989) 之
中性假說梭測 (Kimura, 1983),梭測存在於族群 間的變異是否只受到隨機基因漂變的影響,而不 受天擇選汰的影響。 計算族群內核苦酸歧異度( nucleotide diversi旬, π) 以及基因型歧異度(haplotype diversity,的 (Nei, 1987)來評估族群內的遺傳變 異程度。並計算分化指數(FST,fixationindex) 以 及基因交流指數(Nm,gene flow index)(Hudson et
a!., 1992)°FST表示族群間的遺傳分化情形,其 值越高表示族群間的分化程度越高。
利用 MEGA(Molecular Evolutionary Genetics Analysis,version 3.1)軟體(Kumar et al.,2004 )
以 Kimura-2-parameter model (Kimura, 1980.) 進
行遺傳距離的估算,並以 PAUP 4.0 軟體
( Swofford,2002)建立 Neighbor-joining(Saitou
& Nei, 1987)以及 Maximum Parsimony 親緣關條
樹型圖;分析使用故發式搜尋 (heuristicsearch) ,
(consensus tree) 。以 bootstrap 重複取樣 1000 次,評估所得親緣關峰、樹型圖的支持度
(Felsenstein, 1985) 。
將各基因型序列資料整合,利用 Ar1equin2.0 ( Schneideret叫, 2000 )軟體進行最小關聯網狀
圖 (minimumspanning network, Excoffier et al., 1992 ; Excoffier et al., 1994) 分析,以探討各基 因型之間的親緣關條、以及族群的過去歷史。 結果 序列及遺傳變異分析 本研究共成功定序 153 隻溪狡蛛粒線體 COl 基因部分片段之 DNA 序列資料。序列長度 共 617旬,平均鹼基頻率為 :T 佔 24.9% 、 C 佔 18 .2%、 A 佔 44.9% 、 G 佔 12% '以 AfT 所佔比 例較高。所有序列資料中包含 34 個變異位點、 23 個基因型,平均核苦酸歧異度 (π) 為 0.0132 卜 平均基因型歧異度 (h) 為 0.857 。經 Tajima'sD test 之中性假說檢測,結果顯示本研究中溪狡蛛COl 部分片段為中性(P>O.l),其變異不受到天擇選 汰的影響。 為了比較各個流域內溪狡蛛的遺傳變異程 度,我們將;其狡蛛分為北部、中北部、中部、南 部和東部五個族群(表一)。北部族群(採樣點 1~8 )包括平漢、烏塗窟、銀河洞、四泉水、草 浦、三氏、奎輝和跑馬古道;中北部族群(採樣 點 9~10 )包括大湖和公館;中部族群(採樣點 11~14) 包括大坑、九九峰、中興新村和桃未坑; 南部族群(採樣點 15~19 )包括關子嶺、灣丘、 龜丹、木瓜坑及和安;東部族群(採樣點20~21) 包括安通和羅山。 各族群間基因型數以南部族群(n= 10) 為 最高的,東部族群 (n = 2) 為最低(表一)。各 族群的核苦酸歧異度介於 0.00046 ,..., 0.00931 之 間,以中北部族群最高、東部族群為最低;基因 型歧異度介於 0.28571 ,..., 0.79681 之間,最高的為 南部族群、最低的為東部族群。 各樣點之溪狡蛛基因型分布如表二所示,結 果顯示基因型的分布其有地域性差異的趨勢。除 了中部和南部地區有共同的基因型分布外,北 部、東部與(中+南)部之間皆沒有共同的基因 型存在,顯示台灣各地區溪狡蛛的遺傳結構已有 明顯地理上分化的現象。而中北部地區則同時兼 其有北部和中部地區的基因型,顯示此區是北部 和中部地區溪狡蛛族群基因交流的過渡地帶。 表二、;美狡蛛各採樣點之基因型分布和頻率。樣點編號如表一所示。
Table 2. Haplotype distribution and frequency ofDolomed,臼 raptorfor each sample location. Location numbers 1-21 refer table 1. SiiIIlJl1e location Haplotype 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 A 2 6 6 4 4 6 l 8 6 B 7 2 5 6 2 C 3 4 6 2 6 D 7 4 3 2 E 5 F 3 G 2 2 H 2 I 2 J 4 K L 扎f 2 N O P Q R S T U V
w
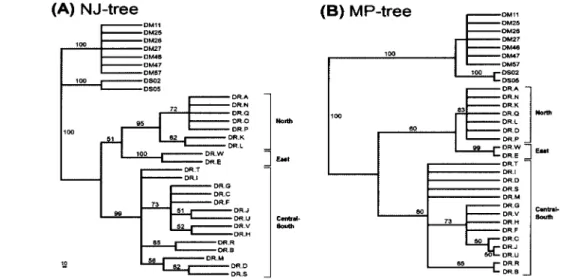
(A) NJ-tree 10。 -Eaae787725 州闖闖闖關關岫甜甜 DDODODDDD 10。 句。。 hJ 叫 酬 岫" E 「1||||| 」 1 」 DR.G DR.C DR.F
..
DR.J DR.U DR.v DR.H OR.R OR.B DR.M 理 DR.O DR.S (B) MP-tree OM刊 OM25 OM26 DM27 OM46 0M47 OM!57 100 _0502 ‘..0505 DR.A .., DR.N I DR.K I DR.Q I North DRιI DR.D I DR.P I CR.w-1Eut DR,E I DR.T DR.! DR.D DR.S DR.M DR.G IICen甘﹒. DR.v I Sou.. DR.H DR.F DR.C DR.J CR.U DR,R DR.B 10。 10。 80 eo.鴨rei· . . .呦, 2且 73 車里圖二、利用(A) neighbor-joining以及(B) maximumparsimony 方法重建之台灣地區溪狡蛛親緣關條樹型圖,其中
DR 為溪狡株 'DM 和 DS 為外群分別為褐腹狡蛛和赤條狡蛛。二者可將溪狡蛛區分為北部、東部和中南部三大
靠群。
Figure 2. Phylogenetic relationships ofDolomedesrap的rconstructed by (A)neighbor-joini 月(NJ)and (B) maximum parsimony (MP) methods in Taiwan. Both trees show thatD. raptorcan be subdivided into three major lineages-the North,East and Central-south lineages. Abbreviations: DR,D. raptor,DM,D. mizhoanus,and DS,D. saganus. Both DM and DS are outgroups.
族群間基因交流與分化 ;美狡蛛族群間的基因交流指數 (Nm) 與遺傳 分化指數 (Fsr) 如表三所示。結果顯示,中部和 南部族群的遺傳分化指數最低 (Fsr=0.103) , 其 族群間的交流較為順暢,而其餘族群間的遺傳分 化指數都偏高,表示族群間基因交流並不順暢, 尤其是東部族群與其他族群問之遺傳分化指數 特別高 (Fsr>0.79 )。由 AMOVA 分析來檢測不 同階層的遺傳分化程度,結果顯示族群問 (among population) 變其佔所有變其的 70.51% 、族群內 樣點問(among locations withinpopulation) 的變
異佔 4.51% 、而樣點內(within location) 的變其 則佔 24.98%' 顯示台灣地區溪狡蛛大部分的變其 存在於族群間與樣點內,尤其族群問佔了大多數 的變異,表示各地區溪狡蛛之間的遺傳結構差異 顯著。 親緣關係樹型圖 將溪狡蛛以基因型為單位,利用 Kimura-2-paremetermodel 計算遺傳距離,並利用 bootstrap 方法進行 1000 次重複取樣,得到一個 Neighbor-joining 親緣關條樹型圖(圖二A) ,其 結果顯示台灣溪狡蛛大致上可分為北部、東部和 中南部三大*群。中部與南部地區的基因型無法 各自成為單車群,反而呈現交錯混雜的狀態,顯 示兩地區的基因型組成近似且有部分完全相 同,因此可合併為中南部*群。中北部地區同時 其有分布於北部的基因型和分布於中南部的基 因型,除了基因型A 被歸在北部群之外,其他的 基因型都被歸在中南部的分群,因此無法獨自被 歸為一群。由 Maximumparsimony 親緣關條樹型 圖(圖二 B) 也得到相似的結果,但兩樹型圖之 北部和東部車群分支的支持度都不高,其 bootstrap 值最高僅 60 。 網狀親緣關係圖
以 minimumspanning network 方法重建台灣 地區溪狡蛛最小關聯網狀圖(圖三)也可區分為 北部、東部和中南部三大*群。基因型A 和基因 型 B 位在網狀圖的中央位置,為相對古老的基因 型,在所有個體中所佔的數量也較多,基因型A 為北部族群主要分布的基因型、而基因型B 則是 中部族群主要分布的基因型之一;其餘位在網狀 圖末端位置的基因型,則為相對年輕的基因型; 基因型 E 和基因型 W 為東部族群特有的基因 型,是從北部的基因型A 衍生而來,因此顯示東 部族群可能是北部族群由宜蘭越過立霧漢、花蓮 j柔而來。
表三、;其技蛛各族群間之遺傳分化指數 (Fsr) 及基因交流指數 (Nm) • 對角線以土之數值 Ii!, Nm 、對角線以下之
數值1', F.\T
Table 3. Degree of gene flow expressed byFsr(lower-left) andNm(upper-right) values among the five populations
orth Central-North Centrol South East
North 。 19 0.05 0.05 0.01 Cenlnll-North 0.573 。 71 。.37 0.07 Cenlnll 0.827 。 260 2.18 0.03 South 0.845 0 .4ω 。.103 0.03 East 0.969 。 790 0.878 0.887 North
s
Central-South East 團三、台灣地區溪狡蛛基因型之最小闕,時網狀圈,可區分為北部、東部和中南部三大~群A-W 代表各基因型, 數值為總苦種改變的步數Figure 3. Haplotype minimum spanning network ofDolomedes rapror in Taiwan. The enUre c1adogram could be divided into thr'自 m句or lineages the North. East and Cenlnll-soutb Ijneages. A W ,"dieme haplotypes and numbers indicate steps of nucleotide changes.
討論 台灣溪狡蜍的基因交流與族群分化 由親緣關條樹型國的結呆,台灣溪狡蛛大致 土可分為北部、中南部以及東部三大*-群﹒東部 族群具有獨立的基因型,是其他族群所沒有的; 而中央山脈以面的部分,南部和中部族群具有許 多相同的基因型,其遺傳分化指數偏低 (F;訂= 0.103) ,顯示溪狡妹中都與南部族群間仍有相當 程度的交流﹒這種南北方向的基因交流以中北部 地區(笛果縣)為其界線, ~r 笛祟的大潮和公館兩 個樣點所代表的中北部族群,這兩個樣點都同時 具有北部族群以及中南部族群的基因型,而北部 族群和中南部族群開貝'J 完全沒有相同的基因 型,表示苗桑地區1'"蟲狡抹在中央山脈西側南北 基因交流的過渡地帶﹒雖然中北部族群同時具有 北部和中南部族群的基因型﹒但只有基因型A 典 北部族群相同,其餘的太多和中南部族群相同, 而其獨特的基因型R 在統緣關條街型函中也銬在 中南部*-群﹒由遺傳分化指數來看,北部和中北 .g~族群間的分化指數 (FIT 一 0.573) ,也比中北部 和中.g~族群問 (
F
IT~ 0.260) 、以及中北部和南部 族群間 (Fsr=
0 .404) 稍微來的高,顯示苗祟的 大潮和公館兩個樣點的,真狡蛛﹒其親緣關你是和 中南部族群比較接近的﹒ 上述結果,顯示台灣地區,其狡妹的族群分化 模式與人面蜘蛛的完全不一樣 Lceel 01. (2004) 研究台灣全島各地的人面蜘蛛遺傳結構,幾乎都 為 EA 基因型分布,僅有在西北部和東北部地區 有少數其他基因型分布,顯示台灣地區的人面蜘 蛛並沒有顯著的遺傳分化現象﹒一般認為許多昆 蟲手,蜘蛛因為具有飛行或空飄能力,較不受地形 阻隔、能進行長距離的遷移,而人面蜘蛛的這絲 室厲風 (ballooning) 行為正可幫助其長距離遷移 Gertsch ( 1979) 指出,給由室氣來移動的蜘蛛, 其移動距離可達 300 km 遠、移動高度可高速將 近 5km 因此強大的擴散能力應是人面蜘蛛未受 到中央山脈阻隔、族群間基因交流順暢的主要原 因﹒相反的, ;真 4主妹不但沒有空軍現行為,且棲地 選擇偽限於有水的環境、在台灣分布的最高海拔僅約 1000 m' 遠低於中央山脈的主稜。其他研究 者也指出,分布在台灣低海拔的物種可能會受到
中央山脈的阻隔,使他們在台灣東、西兩側分化
並產生遺傳結構上的差異 (Hsu et aI., 2000,
2001) ,例如其氏樹蛙 (Yeh, 1997) 、澤蛙 (Toda
et aI., 1998) 和赤尾青竹絲(Creer et aI.,2001 )
等。本研究亦證實中央山脈的阻隔,對於分布在 海拔 1000 m 以下的漢狡蛛而言,也是一道天然 的地理屏障,族群間的基因交流確有顯著的抑制 作用。 除了山脈阻隔之外,由於溪狡蛛多棲息在漢 洞、平緩的溪流和池塘等環境中,臨水而居,最 遠曾在距離水邊約 50 公尺的山坡上被發現(Chen, personal observation) ,推測溪狡蛛的遷徙和擴散 主要是靠著水車上下游之間的河道交流,橫越陸 地礦散的距離並不會太遠,因此除了山志之外, 各水在之間的寬闊陸地可能也是阻隔溪狡蛛族 群間交流的屏障。本研究結果顯示,台灣地區溪 狡蛛的族群分化模式和一些初級性淡水魚較為 類似,例如台灣石食賓(Acrossocheilusparadoxus)
( Wang et aI.,2000)和台灣鏟領魚(均ricorhinus barbatuIus) (Wang et叫, 2004 )等。初級性淡水 魚的遷徙或擴散主要是靠著水車上下游之間的 河道交流,除了河川襲奪之外,很少出現自然擴 散的情況,而台灣地區的淡水魚由於生活的水域 受到山祟的隔離,以及各水系之間具有不同的水 文形態,使各水系問聯繫不易,因此淡水魚會在 各自的溪流中形成獨立的族群,其遺傳變異就可 能會出現區威性分化的現象。台灣地區溪狡蛛在 中央山脈以西,依基因型分布可以後龍溪為界分 為北部和中南部兩大~群,北部~群包括了淡水 河和竹安溪流域,南部在群包括了大肚漠、曾文 ;柔和楠梓仙溪流域,而後龍溪流域為北部和中南 部兩大~群南北的過渡地帶,此一結果和同樣以 後龍溪為界分為南北兩大族群的台灣石魚賓非 常相似 (Wanget aI., 2000) ;而漢狡蛛以中央山 脈和後龍溪為界分為北部、中南部和東部三大~ 群,亦和 Tzeng (1986) 依照淡水魚分布以中央 山脈和濁水溪流域為界劃分為中北部、南部和東 部三個地理區相似。;美狡蛛許多特性不同於其他 陸生節肢動物,受到地理阻隔的影響反而和初級 性淡水魚較為類似,呈現類似的分化模式,說明 了生活習性、行為和生態環境選擇是造成漢狡蛛 和其他陸城節肢動物有不同分化模式的重要因 素。 而造成溪狡蛛以大肚溪流成為主的中部族 群和色括曾文 j柔和楠梓仙溪為主的南部族群之 間仍有一定程度交流的原因,推測可能和幾個因 素有關,包括中部和南部的地勢較為平坦、水在 較為複雜、以及農業發展的關餘,有許多人工開 鑿錯縱複雜的大圳、大排水溝和灌溉渠道來連結 各大溪流,造成流城間的水系隔離並不完全,使 漢狡蛛可以在豐水期或是大雨時、藉由暴漲的溪 水擴散到其他流域,促進族群間的交流。 溪狡蛛親緯地理關係
在 minimum spanning network 的分析方法 中,認為祖先族群中的古老基因型可經由突變或 重組而不斷衍生出新的基因型,使古老的基因型 有較大的機會位於最小關聯網狀圖內部,而新衍 生出的基因型則會位於網狀圖的邊緣(Crandall & Templeton, 1993)' 古老基因型通常分布較廣 泛,在族群中所佔的比例也較高。由本研究的分 析結果顯示其基因型亦大致可分為北部、中南部 和東部三大群(圖三) ,基因型 A 和基因型 B 位 在最小關聯網狀圈內部,是屬於相對古老的基因 型,而連接在基因型 A 末端的基因型 E 和基因型 W' 是離開祖先族群後所衍生出來的新分支,僅 出現在東部的安通和羅山,因此可以推論東部族 群是由北部族群越過立霧漢、花蓮溪遷移而來, 使東部族群和北部族群有較近的親緣關餘,在
Neighbor-joining 以及 Maximum Parsimony 親緣 關條樹型圖中也支持此種假說。但由於立霧;真的 消急和清水斷崖特殊的地形成為生物的天然地 理屏障, ~.美狡蛛要越過此障礙由北部擴散到東部 建立族群並不容易,由東部族群偏低的遺傳變異 (π=0.00046 ; h= 0.28571 ) ,說明勢必經歷嚴重 的瓶頸效應 (bottleneck effect) 或創始者效應
(founder event) (Avise,2000 ; Grant & Bowen 1998)。相反的中南部溪流相對較為平緩,開發 利用程度也不如北部嚴重,在中低海拔山區仍有 許多植被豐富、遮蔽度高且不受人為干擾之環境 過合溪狡蛛棲息,形成長期而穩定的族群,因而 擁有較高的遺傳變異 (π= 0.00590--0.00625 ;h= 0.77586--0.79681 ) 本研究結果顯示台灣地區溪狡蛛同時受到 地形及水車的隔離影響,已有顯著族群遺傳結構 上的分化,並呈現不同於一般節肢動物特殊的分 化模式。
聽謝 本研究蒙農委會林務局經費補助( 95 農料 -1 1.1. 3-務 -el(7)) ,國立台灣師範大學生命科學系 遺傳多樣性實驗室李壽先老師在實驗操作上的 協助,種子植物分類研究室王震哲老師提供資料 分析上的協助,以及蜘蛛研究室劉錫軒、黃文俊 和鄭天勻等協助採集樣本,特予誌謝。 參考文獻
Avise J
e.
2000. Phylogeography: The History and Fonnation of Species. Harvard University Press,Cambridge.Chen SH. 2001. A Guide to Common Spiders of Taiwan. Council of agricultu時, Executive Yuan,RO
e.
(in Chinese).Colgan DJ, Brown S, Major RE, Christie F, Gray MR and CassisG. 2002. Population genetics of wolf spiders of fragmented habitat in the wheat belt of New South Wales. Molecular Ecology 11: 2295-2305.
Crandall KA and Templeton AR. 1993. Empirical tests of some predictions from coalescent theory with applications to intraspecies phylogeny reconstructure. Genetics 134: 959-969.
Creer S, Malhotra A, Thorpe RS and Chou WHo 2001. Multiple causation of phylogeographical
防御m as revealed by nested clade analysis of the bamboo viper (Trimeresurus st可negeri) within Taiwan. Molecular Ecology 10:
1967-1981.
Excoffier L and Smouse PE. 1994. Using allele frequencies and geographic subdivision to reconstruct gene 甘ee within a species: molecular variance parsimony. Genetics 136: 343-359.
Excoffier L, Smouse PE and Quattro JM. 1992.
Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data. Genetics 131: 479-491.
Fang K,Yang CC,Lue BW, Chen SH & Lue KY. 2000. Phylogenetic corroboration of superfamily Lycosoidea spiders (Araneae) as inferred from partial mitochondrial 12S and 16S ribosomal DNA sequences. Zoological Studies 39(2): 107-113.
Felsenstein J. 1985. Confidence limits on
phylogenies: an approach using the bootstrap. Evolution 39(4): 783-791.
Folmer 0, Black M, Hoeh W, Lutz Rand Vrijenhoek R. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology 3: 294-299.
Garb JE,Gonza'lez A and Gillespie RG. 2004. The black widow spider genus Latrodectus
(Araneae: Theridiidae): phylogeny,
biogeography,and invasion history. Molecular Biology and Evolution 31: 1127-1142.
Gemmell NJ and Akiyama S. 1996. An efficient method for the extraction of DNA from vertebrate tissues. Trends in Genetics 12: 338-339.
Gertsch WJ. 1979. American Spiders. 2nd ed. New York: Van NostrandRei址lOld.
Gillespie RG, Croom HB and Palumbi SR. 1994. Multiple origins of a spider radiation in Hawaii. Proceedings National Academy of Sciences,
USA 91: 2290-2294.
Grant WAS and Bowen BW. 1998. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation. Heredity 89: 415-426.
Hedin M. 2001. Molecular insights into species phylogeny, biogeography, and morphological stasis in the relict spider genus Hypochilus (Araneae: Hypochilidae). Molecular Phylogenetics and Evolution 18(2):238咽25 1.
Hedin M and Maddison WP. 2001. A combined molecular approach to phylogeny of the jumping spider subfamily Dendryphantinae (Araneae: Salticidae). Molecular Phylogenetics and Evolution 18: 386-403. Hsu FH,Lin FJ and Lin YS. 2000. Phylogeographic
variation in mitochondrial DNA of Fonnosan white-bellied rat Niviventer culturatus.
Zoological Studies 39: 38-46.
Hsu FH,Lin FJ and Lin YS. 2001. Phylogeographic sturcture of the Fonnosan wood mouse,
Apodemus semotus Thomas. Zoological Studies 40: 91-102.
Hudson RR, Slatkin M and Maddison WP. 1992. Estimation of levels of gene flow from DNA sequence data. Genetics 132 : 583-589.
Kimura M, 1980. A simple model for estimating evolutionary rates of base substitutions
through comparative studies of nucleotide sequences. Journal of Molecular Evolution 16: 111-120.
Kimura M. 1983. The Neutral Theory of Molecular Evolution. Cambridge University Press,
Cambridge,MA.
Kumar S, Tamura K and Nei M. 2004. MEGA3: integrated software for molecular evolution genetics analysis and sequence alignment.
Briefings in Bioinformatics 5: 150-163.
Lee HY. 2004. Study on mtDNA variability of two species of Pieris butterflies (Lepidoptera,
Pieridae) in Taiwan. Master's thesis,National Taiwan Normal University, Taipei. (in Chinese).
Lee JW, Jiang L, Su YC and Tso 1M. 2004. Is Central Mountain Range a geographic barrier to the giant wood spider Nephila pilipes (Araneae: Tetragnathidae) in Taiwan
?
A population genetic approach. Zoological Studies 43(1): 112-122.Nei M. 1987. Molecular Evolution Genetics. Columbia University Press,New York.
Nibouche S, Bues R, Toubon JF and Poitout S. 1998. Allozyme polymorphism in the cotton bollworm Helicoverpa armigera (Lepidoptera: Noctuidae) : comparison of African and European populations. Heredity 80: 438-445. Ramirez MG and Haakonsen KE. 1999. Gene flow
among habitat patches on a fragmented landscape in the spider Al宮iope trifasciata
(Araneae: Araneidae). Heredity 83: 580-585. Roderick GK. 1996. Geographic structure of insect
populations: gene flow, phylogeography, and their uses. Annual Review of Entomology 41: 325-352.
Rozas I and Rozas R. 1999. DnaSP version 3: an integrated program for molecular population genetics and molecular evolution analysis. Bioformatics 15: 174-175.
Rozas J,Sanchez-DelBarrioJC,扎1essenguerX and Rozas R. 2003. DnaSP, DNA polymorphism analyses by the coalescent and other methods. Bioformatics 19: 2496-2497.
Saitou Nand Nei M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. 扎10lecular Biology and Evolution 4: 406-425.
Schneider S, Roessli D and Excoffier L. 2000. ARLEQUIN 2.0: A Software for Population Genetic Data Analysis. Genetics and Biometry Laboratory, University of Geneva,
Switzerland.
Simon C,Frati F,Beckenbach A, Crespi B,Liu H and Flook P. 1994. Evolution, weighting and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers. Annuals of the Entomological Society of America 87: 651- 701.
Slatkin M. 1985. Gene flow in natural populations. Annuals Review of Ecology and Systematics 16: 393-430.
Swofford DL. 2002. PAUP*. Phylogenetic Analysis Using Parsimony (*and other methods). Version 4. Sinauer Associates, Sunderland, 扎1assachusetts.
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis of DNA polymorphism. Genetics 123: 585-595.
Toda 孔1,孔1atsui 扎。'I, Lue KY and Ota H. 1998. Genetic variation in the Indian rice frog,Rana limnocharis (Amphibia: Anura) in Taiwan,as revealed by allozyme data. Herpetologica 54: 73-82.
Tzeng CS. 1986. Distribution of the freshwater fishes of Taiwan. Journal of Taiwan Museum 39: 127-146.
Vink CJ and Paterson AM. 2003. Combined molecular and morphological phylogenetic analyses ofthe New Zealand wolfspider genus
Anoteropsis (Araneae: Lycosidae). Molecular
Phylogenetics and Evolution 28:576-587. Wang JP, Hou KC and Chiang TY. 2000.
Mitochondrial DNA phylogeography of
Acrossocheilus paradoxus (Cyprinidae) in Taiwan. Molecular Ecology 9: 1483-1494. Wang JP,Lin HD,Huang S. Pan CH,Chen XL and
Chiang TY. 2004. Phylogeography of
均ricorhinus barbatulus (Cyprinidae) in Taiwan based on nucleotide variation of mtDNA and allozymes. Molecular Phylogenetics and Evolution 31: 1143-1156. Yeh WS. 1997. Phylogeographic structure of
Rhacophorus moltrechti. Master's thesis,
National Taiwan University, Taipei. (in Chinese).
Study on Population Genetic Structure and Phylogeography of
Dolomedes
rapωr
(Araneae
,
Pisauridae) in Taiwan
Ying-Yuan Luo,Shyh-Hwang Chen*Department ofLife Science,National Taiwan Normal University
Tai闕,Taiwan
(Received: 15 September20 日, accepted: 1 February 2012)
ABSTRACT
Dolomedes raptor may have weak dispersal ability because of their habitats restricted in water environment, mostly along the grassy bank of ponds, rocks along the streams or creeks,without the ballooning behavior,and distributed at elevations below 1000 m. Moreover,the Central Mountain Range (CMR) and rivers in Taiwan form a natural geographic barrier for many organisms in the island. Therefore, we chose D. raptor to investigate their population genetics and phylogeography in Taiwan.
We used the partial sequence of mitochondrial DNA cytochrome oxidase subunit I (CO1) as a genetic marker in this study. A total of 153individuals 企om 21 localities in Taiwan were obtained. The length of sequenced COI gene was 6l7bp,including 34 polymorphism sites and 23 haplotypes. Analysis of AMOVA indicates that genetic structures are different significantly among populations. Phylogenetic trees generated from both Neighbor-joining and Maximump訂simonymethods showed that D. rapωrin Taiwan could be grouped into three major lineages, namely northern, eastern and central-southern lineages. All 也e FST values are high between populations, except for those between the central and
southern,the central and central-northern populations(FST= 0.10299-0.26942).Itindicates populations of
D. raptorin Taiwan exhibited genetic differentiation is resulting from isolations by both the mountains and the lands between basins. The differentiation model of D. raptor is similar to those of primary freshwater fishes,but not Nephilapilip白,an orb-web spider,with strongly dispersal ability. Moreover,the gene flow between south and central populations was notisolatβd.Itmight be resulted from the southern and central parts of Taiwan that have a smooth topology,with complex drainage systems for agriculture and are frequently irrigated by the people.
Minimum spanning network showed 血at the eastern population was derived from the northern population. However,the genetic diversity in eastern population showed lowindicates 曲的 itmight have undergone the strong founder event or the bottleneck effect.