利用格網環境佈署兩種剪裁對齊工具
Deployment and Implementation of Two Spliced
Alignment Tools on Grid environment
許芳榮 逢甲大學資訊工程學系教授、生物資訊研究中心主任、 生醫資訊暨生醫工程碩士學位學程主任 Email:frhsu@fcu.edu.tw 薛偉成 逢甲大學資訊工程學系生物資訊實驗室 Email:m9490309@fcu.edu.tw 摘要―本實作以 globus 建構的格網環境為基礎,佈 署兩種不同的生物序列剪裁對齊工具,以解決大量的序列 比對以及資料儲存問題。針對兩種不同的剪裁工具提出不 同的服務流程,紀錄並監看工作狀態,參考節點效能為負 載平衡之依據。網頁式操作介面,取代使用者過去需記憶 指令的操作方式。
Abstract ― Spliced alignment is an important and time-consuming task in bioinformatics research. In this study, we show how to implement and deploy two spliced alignment tools on grid environment. In this grid environment, jobs status are recorded and monitored. Users could assign tasks and data partition methods according to performance of each site. A user friendly web service is also available.
關鍵詞―剪裁對齊工具,格網計算,序列比對
Keywords ― splice alignment tool , grid computing,sequence alignment
一、簡介
在生物資訊領域的研究如研究 SNP 事件、 RNA editing、AS……等事件研究都和 EST 比對 有關,每年都有大量的 EST 序列被發表,NCBI 每年也公佈相當多的基因體計畫擷取的基因體 序列,如此大量的序列需要被比對,使得高計 算能力以及高儲存能力的環境被需要。不同的 序列剪裁比對工具有不同的比對結果,單一比 對工具的結果顯得太過武斷,採用兩種或兩種 以上的比對工具較為客觀。然而選用的工具愈 多,所需的計算與儲存能力更加需要。格網環 境的高計算與高儲存能力被廣泛運用在各領域 [9],在生物資訊格網方面,早期的 BeoBLAST[6] 透過查詢資料的分割方式將多個查詢分散到多 個節點上;Aaron E.等作者所提的 mpiBLAST[1] 將 BLAST[2] database 做分割並分散的平行環境 上,透過分散式的查詢達成平行處理;Paulo C Carvalho 等作者發表的 Squid[7]實作出尋找閒置 節點才派送查詢工作的方式,並紀錄節點狀 態;Arun Krishnan 提出的 GridBLAST[3],進一 步提出評估格網效能方式;Chao-Tung Yang 等 作 者 提 出 的 G-BLAST[4] 更 結 合 叢 集 計 算 環 境,提出節點效能篩選策略,對於較高效能的 節 點 給 予 較 多 的 查 詢 數 。 這 些 環 境 都 使 用 BLAST 工具;然而除了 BLAST 外,常見的 ClustalW 的多序列比對工具也被廣泛運用到格 網環境上。但鮮少見剪裁比對工具被佈署到格 網環境上。
二、剪裁比對工具
剪裁比對設計用於剪裁的序列比對於對應 的基因體序列上。剪裁比對的問題,是為了找 出基因體序列候選的外顯子(exons)鏈,而這些 候選的外顯子鏈能最適合對應到目的序列上。指定輸入基因體序列 G,目的序列 T,以及候選 的外顯子集合 B。候選的外顯子鏈(Γ)使全域比 對分數 S(Γ*,Γ)在所有的候選外顯子鏈是最高 的。剪裁比對如圖 1 所示。 圖 1 序列剪裁比對說明 透過序列剪裁比對工具,利用剪裁形式找 出外顯子(exon)與內隱子(intron)找出序列最適 當的裁切位置,再將 EST 序列對回基因體序列 上。依先前的研究指出依不同的工具而言,內 隱子的裁切位置超過 92%落在 GT-AG 上,此外 有其他類型的裁切位置如 GC-AG。
GMAP[8]是 mRNA 及 EST 序列映射及比對 到基因體的工具軟體。可快速進行序列映射或 比對,無論映射或比對都可隨意切換基因體資 料庫,如硬體允許,可批次處理或多執行緒執 行。作法上以長度為 24bp 建立索引查找表,以 及索引偏移量建立表格,實際資料結構為兩個 12bp,映射時以兩個 12bp 長的序列為基準,往 EST 兩端找出最長的映射長度;比對時沿用映 射結果為基礎,以最長的映射序列片段兩端做 動態規劃,待使用者輸入待查詢序列找出待查 詢序列位於基因體序列哪個位置 以及相似程 度。
MUGUP(Multi-Layer Genome Unique
Markers Positioning) [5]這個序列比對工具,基礎 理論是 Multi-Layer Unique Marker (UM)多層單 標籤方法,在基因體中只出現一次的片段。例 如長度為 14 的 Unique Marker 代表 14 個連續的 bp 在基因體中只出現一次的序列當作比對的索 引,具有定位的功能,單標籤長度愈長,定位 率愈高,缺點花費時間愈長;短 UM 數量少而 定位快速,採用多層單標籤方法,於長 UM 中 插入短 UM 結合短 UM 的快速與長 UM 的高定 位成功率,定位後再向基因體序列上、下游兩 端序列以貪婪法做動態規劃,比對出更長的序 列片段,適合 ESTs 或 mRNAs 比對到全基因體 序列。MUGUP 當使用 UMs 來定位時,UMs 之 長度越長,唯一性愈高,定位的成功率越高, 但所需時間卻是急速成長。整個比對動作分為 兩個部份:預先將全基因體序列(Whole Genome) 或 接 合 序 列 (contig) 或 染 色 體 (chromosome) 掃 描,找出 UM 並紀錄這些 UM 位置使用長度為 7、14、21、28 做為索引長度,建立多層單標籤 表格。 標籤表格建立步驟如下所述: 步驟一:將長度為 14、21、28 個 bp 所轉化的 四進位值建表,並紀錄其所在位置,依四進位 值排序(bucket sort)後去除有重複的資料,得到 在基因組上只出現一次紀錄,即找到 UM14、 UM21、UM28。 步驟二:為建立 UM14、UM21、UM28之多 層單標籤的架構,需將 UM28的位置紀錄,依排 序後的結果,每 7 個 bp 分為一層索引,則分成 四層。並將 UM14、UM21插到所對應由 UM28產 生的索引層內,即完成多層單標籤的建置。 當多層單標籤表格建立完成後,可用來映 射 EST、mRNA 或 SNP,亦為比對必要步驟, 映射及比對步驟如下: 步驟一:算出在序列中每一個位置向尾取 7 個 bp 的四進位值。 步驟二:將第 1 個位置、第 8 個位置、第 15 個位置、第 22 個位置,四個 7 位四進位值, 配成一組,分別當作搜尋第 1 層、第 2 層、第 3 層、第 4 層索引的值,若在第二層就在序列上 找到 UM14,則將序列第 1 個到第 14 個 bp 在基 因體上的位置標示出來,再用第 15 個位置、第 22 個位置、第 29 個位置、第 36 個位置的 7 位 四進位值查多層單標籤;若是到第四層才查到 只出現一次的單標籤 UM28,則將此位置之後 28
個 bp 在基因體上的位置標示出來,並從此 28 個 bp 之後繼續循環使用步驟 2,若用所在位置 的那一組四個 7 位四進位值查多層單標籤卻找 不到 UM,則移動一個位置,由下一組四個 7 位 四進位值來查表。 步驟三:以序列上最多 bp 所標示的接合序 列為準,去除位置的矛盾 UM,以多數決定序列 在基因體上的起始位置,即完成序列的定位。 該結果亦為 EST 映射到某接合序列的結果。 圖 2 MUGUP 內部流程圖 找到定位片段後,隨即往兩端以貪婪法則 進行動態規劃,並計算分數,找出最相似片段, 即為比對結果。實作上 EST 依此相同步驟分比 對到基因體正股及反股作比對,將 EST 序列反 轉 後 亦 比 對 到 基 因 體 正 股 與 反 股 序 列 上 。 MUGUP 流程如圖 2;序列比對動作如圖 3。 EST 正股 EST 反轉 反股 圖 3 MUGUP 序列比對實際動作之四種組合 格網為平行分散式系統,屬於機器分散層 級、半開放式網路,整合分散各地的資源成為 更大的資源系統,這些資源包含計算資源、儲 存資源、資料集……等,透過網路互連在一起, 彼此共享這些資源。當這些資源被整合後,可 以減少整合的計算資源的在管理上的資源閒置 時間,提供資源管理、資源分配、資源利用、 格網使用者管理、工作排程、格網安全等服務。 Grid Middleware 為應用服務與節點資源間的中 間層,提供格網環境相關議題方面的服務。 格網計算的特徵包含彈性,安全,協調各 體、組織動態收集的資源間的分享,簡明,安 全,資源分享間的協調及跨站合作,虛擬組織 的協同能力,在開放的異質伺服環境分享應用 程式及資料共同解決問題,聚集大量異地分散 的計算資源能力以處理大問題如同所有的伺服 器及資源放在單一網點,軟、硬體基礎設施提 供獨立、一致、普及的且低成本可使用的計算 資源。格網中介層底層為實體層:實體機器透 過網路設備彼此連接在一起;上一層為實體硬 體資源及連接的通訊協定,為各機器的處理 器、記憶體、儲存設備……等資源;再上一層 為格網中間層,格網環境的虛擬作業系統,提 供格網相關服務;最上層則是格網應用服務 層,對於在格網環境使用者所使用的應用服 務,都由這層的服務提供,例如生物資訊之序 列比對應用程式。 Globus Project[10] 提供軟體工具使我們容 易建構格網計算系統及格網應用。這些工具集 合成 Globus Toolkit。Globus Toolkit 包含軟體安 全、資訊基礎設施、資源管理、資料管理、通 訊、失效偵測以及可攜性。
三、剪裁比對工具佈署與系統實作
為解決兩種不同的序列比對工具的龐大計 算需求以及序列資料儲存空間,我們提出格網 環 境 的 實 作 , 稱 為 BRCGrid (Bioinformatics Research Center Grid)。整合 GMAP 及 MUGUP 兩個序列剪裁比對工具並佈署於格網環境上, 設計使用者易於操作之介面,提供序列裁切比 對計算及儲存環境,包含系統各項資源資訊、 工作排程系統與工作派送系統、工作狀態監視 系統、序列分散比對後結果檔之合併處理以及 通知系統、兩工具不同的工作流程、序列資料 儲存系統、網頁介面,系統架構如圖 4。系統邏 輯架構如圖 5。節點資源規格如表 1。 建立三種全基因體標 籤表格 動態規劃比對 基因體序列 Sequence EST/mRNA 比對後結果 檔 result files “映射”序列最長片段User
Biolab01 Biolab02 Biolab03 Biolab04
BioGrid User Information Database Genome/Sequence Database: 1. Genome Database 2. ESTs 3. …...
Biolab01, Biolab02, Biolab03, Biolab04, Biolab06: Computing Grid
Job Monitor System Job Dispatch System Combination Results System Schedule System Information System Fetching Genome Database Return Results (HTTP)
Create a New Job
Preparing to Execute Execution sub-jobs Fetching Grid System Information Save/Update/fetching Grid System Information
Job Status Information: .Job run information .Execution time .Execution data number .Job status Query Status Quer y Stat us Biolab06 Update & Fetching Server Information Checking Genomic Database 圖 4 BRCGrid 系統架構圖 圖 5 BRCGrid 系統邏輯架構圖 表 1 各節點資源規格表
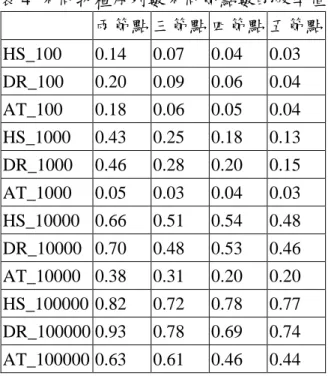
主機 host01 host02 host03 host04 host06 IP 140.134 .*.* 140.134 .*.* 140.134 .*.* 140.134 .*.* 140.128 .*.* CPU P4 1.5G P4 1.5G P4 1.5G P4 1.5G P4 2G 主 記 憶體 SDRAM 1G SDRAM 1G SDRAM 1G SDRAM 1G DDR 512M 硬碟 160GB 7200rpm 160GB 7200rpm 160GB 7200rpm 160GB 7200rpm 80G+ 250G 網路 10/100 10/100 10/100 10/100 10/100 Genome/Sequence Database 存放各物種各 版本的 GMAP 資料庫檔案及 MUGUP 各類型 標籤表格。GMAP 參照的資料庫檔案為物種的 全基因體建立的資料庫檔案。MUGUP 除有全基 因體標表格類型外,還包含染色體標籤表格及 接合序列標籤表格。當子節點在比對 ESTs 序列 找不到適合的標籤表格或資料庫檔案時,則系 統啟動 GridFTP 從 Genome/Sequence Database 複製所需標籤表格或資料庫檔案到需要的子節 點上,繼續執行序列比對工作。這些標籤表格 及資料庫檔案的管理由系統管理者以人工方 式,依照 NCBI 所公佈的各物種版本更新資訊做 不定期的資料庫檔案及標籤表格更新,並不定 期新增物種資料。Information Database 記錄各節 點硬體資訊以及資源使用狀態資料,由 Ganglia 搭配 RRDTool 為紀錄元件,提供圖形化顯示系 統資源。為了這兩種不同工具計算上的負載平 衡,我們定義了節點效能評量方式,依節點效 能將待比對的大量序列資料作不同數量的分 割,效能好的節點分到較多的序列,效能較低 的節點分到較少的序列數。效能的定義為單位 時間內節點能完成的序列比對數量。使節點工 作完成時,有最少的等待時間。效能評估並參 考物種與工具在過去的工作紀錄。以各節點為
單位,計算總比對 EST 數除以總比對時間為效 能,如公式(1)。 Trn Tdn Pn (1) Pn:節點 n 效能 Tdn:節點 n 符合條件的最近五筆歷史紀錄 總 EST 數 Trn:節點 n 符合條件的最近五筆歷史紀錄 總執行時間
BRCGrid 系統實作一個 FCFS(First Come First Serve)的工作排程系統。使用者由頁面選用 工具後所輸入的序列資料以及物種資訊和相關 動 作 類 別 與 參 數 後 , 會 被 記 錄 到 系 統 的”job_record”資料表,並給定工作識別碼。 BRCGrid 系統會定期到”job_record”資料表依照 時間由遠而近擷取未被執行的工作識別碼以及 相關工作資訊,並啟動系統服務。Job schedule algorithm 如圖 6。 Schedule(job serial_num) {
check system flag; if (system is busy)
{
return system busy; }
else {
get the job id and job information form job_record database and the job_id is the smallest;
call system service to run the job;
set system flag as busy;
} }
圖 6 工作排程系統演算法
Job dispatch system 擷取各節點效能作為分 派不同負載之依據,依這些節點效能資訊,將 待比對序列進行依節點效能不同給予不同的序 列數,以序列為單位分割為數個較小的檔案, 再派送到各節點上執行。各節點分割數如公式 (2)。示意圖如圖 7。 Te Pn Pn En m n
1 (2) Pn:節點 n 的效能 m:欲使用之節點數 Te:所有欲被對齊的序列數 En:節點 n 被分配到的序列數Job monitor system 序列比對之工作派送 後,BRCGrid 系統對於工作排程派送之工作進 行工作狀態偵測,即時更新狀態,並顯示於 result files 網頁。
Combination results system 為當各子節點工 作完成後,系統會由子節點透過 GridFTP 將執 行結果傳回主節點,主節點進行結果檔整併, 完成後於網頁通知使用者工作完成,提供使用 者下載結果檔。
Sequence 1 Sequence 2 … Sequence k Sequence 1 Sequence 2 … Sequence r Sequence n+1 Sequence n+2 … Sequence p Output file Node 1 Node 3 Master Node Combinate results Master Node Job Dispatcher …… subjob 1 subjob 3 Sequence k+1 Sequence k+2 … Sequence n Node 2 subjob 2 Master Node Sequence dispatch schema Sequence q+1 Sequence q+2 … Sequence r Node 5 …… subjob 5 Sequence p+1 Sequence p+2 … Sequence q Node 4 subjob 4 Master Node Information System 圖 7 子工作派送示意圖 我們針對 BRCGrid 環境提供的這兩種工具 提出不同的服務流程。MUGUP 更使用兩階段比 對方法。 GMAP 於 BRCGrid 之服務流程如圖 8 所 示。首先於頁面選擇欲使用的序列比對工具, EST 序列由使用者由網頁輸入,並指定欲比對 的物種(包含序列版本)。並取得工具動作型態, 是”剪裁比對”、”映射”或是”映射及剪裁比 對”。系統後端會紀錄上傳的檔案、物種類別、 工具動作類型,並透過 schedule system 編定排 程。經排程確定執行時,系統先行計算 EST 數 量,依照分散的節點數量以及節點效能,進行 分割。接著系統啟動格網服務,通過授權後以 GridFTP 傳遞待比對序列檔、工作命令檔到執行 節點上,並檢查子節點是否存在待比對物種的 GMAP 資 料 庫 檔 案 , 若 不 存 在 , 則 從 Genome/sequence database 複製該物種 GMAP 資料庫檔案到子節點上。隨後進行佈署子工 作,系統會自動取得子工作識別碼,並隨即檢 查各子工作狀態直到所有子工作狀態完成。檢 查子工作狀態過程同時檢查授權的證書時效, 如即將逾期,則再做一次取得授權動作延長授 權證書時效。當子工作全部完成後,系統利用 GridFTP 將子工作執行的結果檔回送回系統,系 統將這些子工作結果檔合併後,於頁面顯示工 作狀態完成並提供結果檔下載。 檢查該物種GMAP資 料庫檔案是否存在? 取得EST,物種名稱, 動作別 EST輸入檔 指定物種資料庫檔 案,版本,及動作別 (map, align, map &
align) 計數待比對序列量 依節點效能分配 EST grid-proxy-init 利用GridFTP傳送 EST檔及子工作指 令檔到各節點 從Sequence Database複製 GMAP資料庫檔案 到執行節點 發佈子工作 取得工作編號並監 看子工作狀態 grid-proxy-init 使用者再次取得證 書 檢查使用者證書 是否即將到期? 子工作完成? 利用GridFTP將子 工作結果檔回傳到 系統 結合子工作結果檔 通知使用者工作完 成並提供結果檔下 載 即將到期 GLOBUS_JOB_DONE 尚未到期 GLOBUS_JOB_ACTIVE 工作排程 不存在 存在 圖 8 GMAP 於 BRCGrid 之服務流程圖 MUGUP 於格網上應用服務流程如圖 9 所 示。在基因體序列當中可分割的最小單位為接 合序列,使用接合序列或染色體表現序列標籤 表格對比對結果沒有差異。其優點是較以全基 因體建置的表格檔案小,除部份稍長的接合序 列或染色體外,多半都可完全被載入到機器主 記憶體內。
檢查該物種 MUGUP標籤表格 是否存在? 取得EST,物種名稱, 動作別 EST輸入檔 指定物種資料庫檔 案,版本,及動作別 (align, locate, map)
計數待比對序列量 依節點效能分配 EST grid-proxy-init 利用GridFTP傳送 序列檔及工作指令 檔及ini檔到各節點 從Sequence Database複製 MUGUP標籤表格 到執行節點 發佈子工作 取得工作編號並監 看子工作狀態 grid-proxy-init 使用者再次取得證 書 檢查使用者證書 是否即將到期? 子工作完成? 利用GridFTP將子 工作結果檔回傳到 系統 結合子工作結果檔 通知使用者工作完 成並提供結果檔下 載 即將到期 GLOBUS_JOB_DONE 尚未到期 GLOBUS_JOB_ACTIVE 工作排程 不存在 存在 圖 9 MUGUP 於 BRCGrid 之比對流程圖 使用 MUGUP 工具時,採用兩階段的序列 比對方法,第一階段使用全基因體標籤表格對 EST 進行映射,將映射結果依照 EST 對應的接 合序列將 EST 序列依對應到的接合序列編號進 行重新建立待比對 EST 檔案;第二階段則將這 些初步分類過的 EST 序列檔,參考接合序列標 籤表格或染色體標籤表格進行序列比對。可減 免一連串的 EST 在剪裁比對上更換標籤表格的 次數與時間。
四、實驗環境與效能評估
環境實驗上,我們使用三種不同物種的基 因體序列資料庫或表格以及這三個物種不同數 量的 EST 檔各五筆。分別採用兩種不同工具分 別以不同節點數分析執行剪裁比對時間、資料 檔案傳輸時間以及格網系統服務時間,並比較 序列剪裁比對參考的資料庫檔案或標籤表格是 否存在對比對時間上的差異。實驗資料集有 Homo sapiens (human) 36.3 版、Danio rerio (zebrafish) Zv7 版、Arabidopsis
thaliana (mouse-ear cress) 8.1 版的基因體序列,
並預先建立 MUGUP 使用之三種類型的表格 (Whole genome table、Chromosome table、Contig table)以及 GMAP 對應的資料庫檔案。並隨機選 取各物種的 EST 100 條、1000 條、10000 條、 100000 條各五個獨立擋案當作測試資料。 實驗上採取單一節點、兩節點、三節點、 四節點、以及五節點,各節點數所選用的節點 資料如表 2。當中 host06 這個節點地理上離其他 節點超過 3 公里。 表 2 實驗節點選定表 節點數 選用節點
單一節點 host01 或 host02 或 host03
兩節點 host06+host04 三節點 host06+host04+host03 四節點 host06+host04+host03+host02 五節點 host06+host04+host03+host02+host01 格網環境的評估首重計算效能,以單機的序列 比對時間為對照組,實驗在 BRCGrid 格網環境 下各序列檔採用不同工具在不同節點數的序列 比對時間。序列比對時間值取五筆同物種同序 列數不同序號檔實驗所得時間值取平均。評估 材料(物種序列以及 EST 數量)、工具(GMAP 及 MUGUP 的差異)、與環境(節點數、節點軟硬體、 各節點是否有標籤表格檔或資料庫檔案、系統 服務時間……等)在時間上的表現。 實驗結果 GMAP 部分
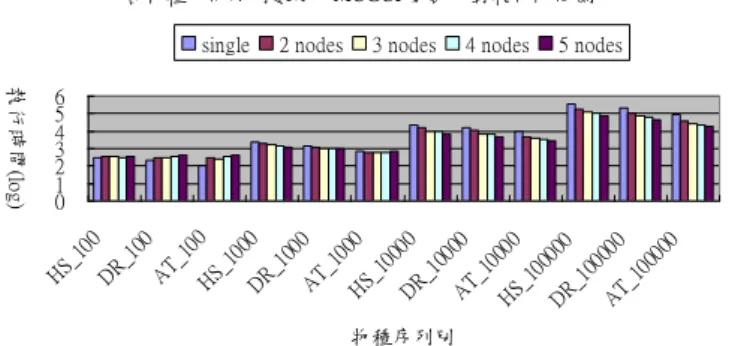
各物種不同序列數不同節點數執行時間 0 1 2 3 4 5 6 HS_1 00 HS_1 000 HS_1 0000 HS_1 0000 0 DR_1 00 DR_1 000 DR_1 0000 DR_1 0000 0 AT_1 00 AT_1 000 AT_1 0000 AT_1 0000 0 物種序列數別 執行時間(lo g)
single 2 nodes 3 nodes 4 nodes 5 nodes
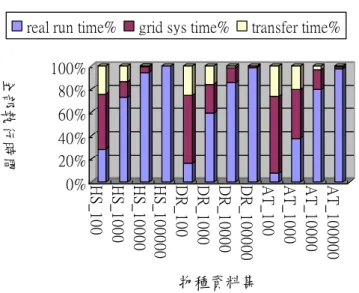
圖 10 GMAP 序列比對工具不同節點數執行時間 (包含資料庫檔案複製時間) 首先討論需做資料庫檔案複製的結果。圖 10 是各序列檔以 GMAP 為工具使用不同節點數 做剪裁序列比對時間比較圖。當中除了單一節 點外(這也是傳統單機必須包含資料庫檔案),其 餘各節點數包含資料庫檔案複製時間,兩節點 為兩個複製時間,三個節點為三個的複製時 間,四節點為四個複製時間,五節點則為五個 複製時間。 不同物種序列數於 BRCGrid 上不同節點數 的加速值如表 3;效率值如表 4。由表中可見在 相對較多的資料集下,BRCGrid 有較高的加速 值以及效率值,顯示格網環境適合大量資料的 處理。圖 11 為五節點上各動作時間比例圖。可 見隨著 EST 數增加,比對時間比例也隨著增加。 表 3 不同物種序列數不同節點數的加速值 兩節點 三節點 四節點 五節點 HS_100 0.29 0.21 0.17 0.14 DR_100 0.40 0.28 0.25 0.21 AT_100 0.36 0.18 0.19 0.20 HS_1000 0.87 0.76 0.72 0.63 DR_1000 0.92 0.85 0.80 0.74 AT_1000 0.11 0.09 0.14 0.13 HS_10000 1.32 1.52 2.14 2.42 DR_10000 1.39 1.44 2.10 2.32 AT_10000 0.77 0.94 0.82 0.99 HS_100000 1.63 2.17 3.14 3.86 DR_100000 1.85 2.33 2.77 3.72 AT_100000 1.26 1.84 1.85 2.21 表 4 不同物種序列數不同節點數的效率值 兩節點 三節點 四節點 五節點 HS_100 0.14 0.07 0.04 0.03 DR_100 0.20 0.09 0.06 0.04 AT_100 0.18 0.06 0.05 0.04 HS_1000 0.43 0.25 0.18 0.13 DR_1000 0.46 0.28 0.20 0.15 AT_1000 0.05 0.03 0.04 0.03 HS_10000 0.66 0.51 0.54 0.48 DR_10000 0.70 0.48 0.53 0.46 AT_10000 0.38 0.31 0.20 0.20 HS_100000 0.82 0.72 0.78 0.77 DR_100000 0.93 0.78 0.69 0.74 AT_100000 0.63 0.61 0.46 0.44 0% 10% 20% 30% 40% 50% 60% 70% 80% 90% 100% 全部執行時間 H S _ 1 0 0 H S _ 1 0 0 0 H S _ 1 0 0 0 0 H S _ 1 0 0 0 0 0 D R _ 1 0 0 D R _ 1 0 0 0 D R _ 1 0 0 0 0 D R _ 1 0 0 0 0 0 A T _ 1 0 0 A T _ 1 0 0 0 A T _ 1 0 0 0 0 A T _ 1 0 0 0 0 0 物種資料集 五節點上動作時間類別(包含資料庫檔案複製)
cp db time% real align time% other time% transfer time%
圖 11 五節點上各動作花費時間比例圖(包含資 料庫檔案複製時間) 各物種不同序列數不同節點數執行時間 0 1 2 3 4 5 6 HS_10 0 HS_10 00 HS_10 000 HS_10 000 0 DR_10 0 DR_10 00 DR_10 000 DR_10 000 0 AT_10 0 AT_10 00 AT_10 000 AT_10 000 0 物種序列數別 執行時間(lo g)
single 2 nodes 3 nodes 4 nodes 5 nodes
圖 12 GMAP 序列比對工具不同節點數執行時圖 (不包含資料庫檔案複製時間)
接著討論無需做資料庫檔案複製時的結 果。圖 12 是各序列檔以不同節點數做剪裁序列 比對時間比較圖。AT 在 1000 條(含)以下的數量 在比對時間上並未因增加節點降低比對時間, 反而隨著節點數增加而增加剪裁比對時間;當 超過 10000 條 EST 後執行比對時間才逐漸隨節 點數增加而降低。其餘各物種各序列數都有顯 著的隨節點數增加而降低比對時間。不同物種 序列數於 BRCGrid 上不同節點數的加速值如表 5;效率值如表 6。 表 5 不同物種序列數於 BRCGrid 上不同節點數 的加速值(不包含資料庫檔案複製) 兩節點 三節點 四節點 五節點 HS_100 1.47 1.36 1.63 1.59 DR_100 1.21 0.92 1.28 1.47 AT_100 0.54 0.23 0.30 0.36 HS_1000 1.54 1.73 2.30 2.57 DR_1000 1.34 1.45 1.65 1.82 AT_1000 0.12 0.10 0.20 0.19 HS_10000 1.44 1.76 2.85 3.70 DR_10000 1.47 1.57 2.50 2.97 AT_10000 0.84 1.10 0.99 1.34 HS_100000 1.65 2.22 3.26 4.09 DR_100000 1.87 2.37 2.84 3.88 AT_100000 1.29 1.92 1.96 2.41 資料庫檔案複製限制了剪裁比對執行時間 上的加速,這個限制僅只在第一次使用該物種 資料庫檔案不存在時,之後資料庫檔案便存在 於節點上,資料庫檔案複製的阻礙便消失。 圖 13 為五節點上各動作時間比例圖,大致 上也可看出隨 EST 數量增加也促使實際比對時 間比例也增加;接著以圖 11 與圖 13 做是否有做 資料庫檔案複製動作看動作時間比例來看,當 經過資料庫檔案複製動作後,確實發現不僅縮 短了整個分散執行的時間也提升了實際比對時 間比例。 表 6 不同物種序列數不同節點數的效率值(不包 含資料庫檔案複製) 兩節點 三節點 四節點 五節點 HS_100 0.74 0.45 0.41 0.32 DR_100 0.60 0.31 0.32 0.29 AT_100 0.27 0.08 0.07 0.07 HS_1000 0.77 0.58 0.58 0.51 DR_1000 0.67 0.48 0.41 0.36 AT_1000 0.06 0.03 0.05 0.04 HS_10000 0.72 0.59 0.71 0.74 DR_10000 0.74 0.52 0.62 0.59 AT_10000 0.42 0.37 0.25 0.27 HS_100000 0.82 0.74 0.82 0.82 DR_100000 0.94 0.79 0.71 0.78 AT_100000 0.64 0.64 0.49 0.48 0% 10% 20% 30% 40% 50% 60% 70% 80% 90% 100% 全部執行時間 H S_ 10 0 H S_ 10 00 H S_ 10 00 0 H S_ 10 00 00 D R _1 00 D R _1 00 0 D R _1 00 00 D R _1 00 00 0 A T _1 00 A T _1 00 0 A T _1 00 00 A T _1 00 00 0 物種資料集 五節點上動作時間類別(不包含資料庫檔案複製)
real align time% other time% transfer time%
圖 13 五節點上各動作花費時間比例圖(不包含 資料庫檔案複製時間)
實驗結果 MUGUP 部分 表 7 使用 MUGUP 不同物種序列數於 BRCGrid 上不同節點數的加速值 兩節點 三節點 四節點 五節點 HS_100 0.92 0.95 1.06 0.94 DR_100 0.65 0.70 0.62 0.54 AT_100 0.34 0.38 0.27 0.23 HS_1000 1.22 1.50 1.55 1.98 DR_1000 1.21 1.42 1.45 1.67 AT_1000 1.18 1.24 1.20 1.14 HS_10000 1.28 2.07 2.29 3.28 DR_10000 1.39 2.12 2.39 3.16 AT_10000 1.77 2.27 2.66 2.91 HS_100000 1.80 2.52 3.39 4.26 DR_100000 1.88 2.65 3.50 4.36 AT_100000 2.25 3.29 3.99 4.82 表 8 MUGUP 不同物種序列數於 BRCGrid 上不 同節點數的效率值 兩節點 三節點 四節點 五節點 HS_100 0.46 0.32 0.26 0.19 DR_100 0.32 0.23 0.15 0.11 AT_100 0.17 0.13 0.07 0.05 HS_1000 0.61 0.50 0.39 0.40 DR_1000 0.60 0.47 0.36 0.33 AT_1000 0.59 0.41 0.30 0.23 HS_10000 0.64 0.69 0.57 0.66 DR_10000 0.70 0.71 0.60 0.63 AT_10000 0.88 0.76 0.66 0.58 HS_100000 0.90 0.84 0.85 0.85 DR_100000 0.94 0.88 0.87 0.87 AT_100000 1.12 1.10 1.00 0.96 各物種不同序列數使用MUGUP剪裁比對執行時間圖 0 1 2 3 4 5 6 HS_1 00 DR_ 100 AT_1 00 HS_1 000 DR_ 1000 AT_1 000 HS_1 0000 DR_ 1000 0 AT_1 0000 HS_1 0000 0 DR_ 1000 00 AT_1 0000 0 物種序列別 執行時間 (l og )
single 2 nodes 3 nodes 4 nodes 5 nodes
圖 14 MUGUP 序列比對工具不同節點數執行時 間圖 由於 MUGUP 的標籤表格檔案相當大,實 驗環境受限於實驗環境有網路流量限制,因此 全不討論檔案複製,僅討論節點數與效能關 係。由圖 14 與表 7 與表 8 可見在少量的序列數 資料集(各物種 100 條 ESTs)都沒有加速現象, 即便資料集所含的資料筆數達到 1000 條 EST 時,仍沒有顯著的加速效果,僅緩慢增加;當 資料集包含的資料筆數達到 10000 條 ESTs 時, 加速值才顯著增加;資料集在 100000 條 ESTs 時,更為明顯。顯示出環境系統適合處理大量 的 EST 比對工作,與 GMAP 現象略同。 從上述現象接著由圖 15 可見,當資料集包 含的 EST 數量偏少時,系統實際執行比對工作 的時間比例不高,也就是說系統花費相對較高 的時間在資料切割與資料傳遞等相關系統服務 時間。然而在工作紀錄也發現,無論資料集所 包含的 EST 數量多寡,系統所需的檔案分割時 間、檔案傳遞時間、結果檔的合併時間以及系 統時間差異不大(略約數十秒),對相對較少 EST 數的資料集而言卻是不利的,對大資料量的資 料集而言幾乎沒有影響,也代表環境適合處理 大量量的資料集。使用 GMAP 時,也有此現象。
0% 20% 40% 60% 80% 100% 全部執行時間 H S_ 10 0 H S_ 10 00 H S_ 10 00 0 H S_ 10 00 00 D R_ 10 0 D R_ 10 00 D R_ 10 00 0 D R_ 10 00 00 A T _1 00 A T _1 00 0 A T _1 00 00 A T _1 00 00 0 物種資料集 五節點上動作時間類別
real run time% grid sys time% transfer time%
圖 15 MUGUP 五節點上各動作花費時間比例圖 此外由表以及可發現當中的 AT_100000 在 兩節點與三節點時(表中粗體字),有超乎於一般 合理的加速值與效率值,這是在多節點時使用 MUGUP 不同於單機使用的標籤表格所得到的 額外加速效果,也確定 MUGUP 的另一種使用 方式有助加速計算。
五、結論
影響序列剪裁比對時間因素包含工具的選 用、資料庫檔案或標籤表格檔案的大小、存在 與否、EST 序列長度、節點效能、序列複雜度... 等。BRCGrid 提供兩個不同的剪裁比對工具, 在大量的 EST 剪裁比對上有很好的效能,對於 少資料量而言卻是不利的;BRCGrid 環境系統 運作時間,以及資料庫檔案複製時間,都是不 利的因素。當節點存在資料庫檔案後,系統效 能幾乎完全展現在資料分割及節點效能上。六、參考文獻
[1] Aaron E. Darling, Lucas Carey, Wu-chun Feng, “The Design, Implementation, and Evaluation of mpiBLAST,” 4th International
Conference on Linux Clusters: The HPC Revolution 2003 in conjunction with ClusterWorld Conference & Expo, Jun. 2003.
[2] Altschul S. F., Gish W., Miller W., Myers E. W., Lipman D. J., “Basic local alignment search tool,”. Journal of Molecular Biology, Vol. 215, Issue 3, pp. 403-410, 1990.
[3] Arun Krishnan, “GridBLAST: a Globus-based high-throughput implementation of BLAST in a Grid computing framework: Research Articles,” Vol. 17, pp. 1607-1623, John Wiley and Sons Ltd., 2005.
[4] Chao-Tung Yang, Tsu-Fen Han and
Heng-Chuan Kan, “G-BLAST: a Grid-based solution for mpiBLAST on computational
Grids,” Concurrency and Computation:
Practice and Experience, Vol. 21, pp. 225-255,
2009.
[5] F. R. Hsu and J. F. Chen, “Aligning ESTs to Genome Using Multi-Layer Unique Makers,”
Proceedings of the IEEE Computer Society Conference on Bioinformatics, pp564-566,
Stanford University, Stanford, CA, 11-14 Aug. 2003.
[6] J. D. Grant, R. L. Dunbrack, F. J. Manion and M. F. Ochs, “BeoBLAST: distributed BLAST and PSI-BLAST on a Beowulf cluster,”
Bioinformatics, Vol. 18, No. 5, pp. 765-766,
2002.
[7] Paulo C Carvalho, Rafael V Glória, Antonio B de Miranda and Wim M Degrave, “Squid - a
simple bioinformatics grid, ” BMC
Bioinformatics, Vol. 6 , 2005.
[8] Thomas D. Wu and Colin K. Watanabe, “GMAP: a genomic mapping and alignment program for mRNA and EST sequences, ”
Bioinformatics, Vol. 21, No.9, pp. 1859-1875,
2005.
[9] Foster I, Kesselman C. “The Grid 2: Blueprint
for a New Computing Infrastructure (2nd
edition) ”. Elsevier Series in Grid Computing. Morgan Kaufmann: Los Altos, CA, 2003. [10] Globus Project