國 立 交 通 大 學
環 境 工 程 研 究 所
碩 士 論 文
生物薄膜反應槽薄膜積垢中
微生物族群分析研究
Bacterial community analysis
in fouled membrane of MBR
研 究 生:張嘉玲
指導老師:黃志彬 教授
生物薄膜反應槽薄膜積垢中微生物族群分析研究
Bacterial community analysis in fouled membrane of MBR
Student:Chialing Chang (張嘉玲) Advisor:Chihpin Huang (黃志彬)
國立交通大學
環境工程研究所
碩士論文
A Thesis
Submitted to Institute of Environmental Engineering
College of Engineering
National Chiao Tung University
in partial Fulfillment of the Requirements
For the Degree of
Master
In
Environmental Engineering
July 2008
Hsinchu, Taiwan, Republic of China
I
生物薄膜反應槽薄膜積垢中微生物族群分析研究
研究生:張嘉玲 指導老師:黃志彬 教授 國立交通大學環境工程研究所 摘 要 生物薄膜反應槽 (Membrane Bioreactor, MBR)是由傳統活性污 泥程序與薄膜技術所結合的一種生物處理程序,但因 MBR 有較高的 污泥濃度 (mixed liquor suspended solids, MLSS),所以在應用上薄膜 積垢現象為其操作及設計之問題點。又生物處理程序中微生物存在的 種類是影響處理效能的關鍵,目前對於 MBR 之微生物相變化與微生 物相與薄膜積垢的相互關係,始終無法得到解答。本研究目的為利用 分子生物技術監測微生物族群結構,透過 PCR 結合 DGGE 的方式追 蹤反應槽菌群之消長,並探討微生物菌群變化和薄膜積垢的相關性。 反應槽中之微生物相分布是均勻的,推測並不會受取樣點位置影 響,而於 MBR 前端增加選種槽單元則有助於使反應槽微生物相單純 化,其中以 Betaproteobacteria 中之 Zoogloea 最佔優勢。此外,MBR 操作在不同 SRT (10 與 60 天),反應槽內微生物相並不會有太大的變 更,有明顯差異的為薄膜上的微生物相,因此推測薄膜積垢的形成決 定於薄膜上的菌相而非反應槽內的菌相;且在 SRT 為 10 天之薄膜上 的微生物相中發現 Verrucomicrobia 與薄膜積垢時反應槽中發現的 Chlamydiae 在親緣關係上相近,因此可假設 Verrucomicrobia 將是導 致薄膜積垢的微生物族群之一。 關鍵字:生物薄膜反應槽、薄膜積垢、污泥停留時間、聚合酶鏈鎖反 應、變性梯度凝膠電泳II
Bacterial community analysis in fouled membrane of MBR
Student : Chialing Chang Advisor : Prof. Chihpin Huang Institute of Environment Engineering
National Chiao Tung University
Abstract
Membrane bioreactor (MBR) is a biological wastewater treatment process in which the conventional clarifiers are replaced by membranes. Because of the almost total retention of biomass in the bioreactor, MBR is a promising wastewater treatment technology. However, membrane fouling hinders the widespread application of MBR. Microorganisms play an important role in degradation of organic pollutants in MBR. The diversity and variation of microorganisms in MBR and their relationship with membrane fouling has not been well understood. In this study, the relationship between bacterial community and membrane fouling was investigated. Molecular biology tools targeting 16S ribosomal DNA genes were used to identify predominant bacterial community in the sludges and on the membrane surface.
The distribution of microorganisms in the reactor was homogeneous regardless of sampling location. After installing an aerobic selector prior to the MBR the diversity of the microorganisms was simplified and Zoogloea in betaproteobacteria phylum became predominant in the MBR. Under different SRT (10, 30, 60 days) operation, the bacterial communities of the sludges were almost identical, but the bacterial community on the membrane surface differentiated instead. It was suggested that the difference in bacterial community on the membranes resulted in the different fouling propensity at different SRTs. Verrucomicrobia was identified on the membrane surface at 10-day SRT and Chlamydiae which was close to Verrumicrobia in phylogeny was also identified when the MBR was seriously fouled. It was proposed that Verrucomicrobia was one of the key microorganisms which induced membrane fouling.
III
致謝
首先誠摯的感謝恩師 黃志彬老師悉心的教導使我得以一窺環工領域的深奧 且在實驗上給予我最大的發揮空間。口試期間,承蒙 李季眉老師、洪俊雄老師 與袁如馨老師爲論文的細心匡正及提供寶貴建議,使學生的論文更趨於完備,不 勝感激,深致謝忱。 兩年裡的日子,實驗室裡共同的生活點滴,學術上的討論、言不及義的閒扯、 讓人又愛又怕的宵夜、趕作業的革命情感、因為睡太晚而遮遮掩掩閃進實驗 室...,感謝眾位學長姐、同學、學妹的共同砥礪,你/妳們的陪伴讓兩年的研 究生活變得絢麗多彩。 感謝學長育俊、文彬、志麟,學姐惠玲、靜宜不厭其煩的指出我研究中的缺 失,且總能在我迷惘時為我解惑,也感謝實驗室的同窗璧如、奕甫、靜逸,恭喜 我們順利走過這兩年。實驗室的學妹怜秀、于婷、雅茹、思穎當然也不能忘記, 你/妳們的幫忙及搞笑我銘感在心。 特別感謝學長志銘與景鴻在分子生物技術領域上的協助,有了你們的加入使 我在研究的旅途增添兩盞明燈。 好友樂竹、旂瑩、俞俞、易慶、必賢在背後的默默支持更是我前進的動力, 沒有你/妳們的體諒、包容,相信這兩年的生活將是很不一樣的光景。 最後,謹以此文獻給我摯愛的雙親。 張嘉玲 戊子盛夏 于竹塹IV
目錄
中文摘要... I 英文摘要...II 致謝...III 目錄... IV 圖目錄...VIII 表目錄...X 第一章 前言...1 1.1 研究緣起 ...1 1.2 研究目的與內容 ...2 第二章 文獻回顧...3 2.1 薄膜生物反應槽 ...3 2.2 薄膜積垢 ...4 2.2.1 薄膜生物積垢 ...4 2.2.2 薄膜積垢可行之解決方法 ...5 2.2.3 胞外聚合物 ...6 2.3 分子生物技術於環境工程上之應用 ...8 2.3.1 聚合酶鏈鎖反應 ...11 2.3.2 變性梯度凝膠電泳 ...12V 2.4 分子生物技術於生物薄膜上之應用 ...13 第三章 材料與方法...15 3.1 實驗架構 ...15 3.2 實驗設備 ...16 3.2.1 沉浸式生物薄膜反應槽 ...16 3.3 反應槽操作內容及條件 ...18 3.3.1 污泥來源及進流水成分 ...18 3.3.2 反應槽操作參數 ...19 3.4 反應槽水質分析 ...20 3.4.1 胞外聚合物分析 ...20 3.5 反應槽微生物菌相分析 ...24 3.5.1 DNA 萃取...24 3.5.2 聚合酶鏈鎖反應 ...25 3.5.2.1 引子 (Primer)...25 3.5.2.2 反應操作條件 ...25 3.5.3 電泳分析 ...26 3.5.3.1 瓊脂膠糖凝膠電泳 ...26 3.5.3.2 變性梯度凝膠電泳(DGGE) ...27 3.5.4 Acrylamide/Bis 膠體之 DNA 純化 ...28
VI 3.5.5 親緣分析 ...28 第四章 結果與討論...29 4.1 分生技術之建立 ...29 4.1.1 DNA 之萃取...29 4.1.2 PCR...32 4.2 添加選種槽對反應槽所造成之影響 ...33 4.2.1 選種槽對反應槽水質分析之影響 ...33 4.2.2 選種槽對反應槽所造成之菌相變化 ...37 4.3 不同 SRT 下反應槽之水質分析與菌相變化 ...44 4.3.1 SRT 為 30 天之水質分析與菌相變化 ...44 4.3.2 SRT 為 60 天之水質分析與菌相變化 ...50 4.4 薄膜上的菌相變化 ...54 4.5 綜合討論 ...59 第五章 結論與建議...60 5.1 結論 ...60 5.2 建議 ...61 參考文獻...62 附錄A 反應槽前端架設選種槽單元之微生物序列比對列表…...71 附錄B 反應槽 SRT 為 10 與 60 天之微生物序列比對列表 ...72
VII
附錄C 反應槽 SRT 為 30 天之微生物序列比對列表 ...73
VIII
圖目錄
圖2-1 活性污泥及 MBR 之處理程序流程 ...3
圖2-2 薄膜積垢因子 ...4
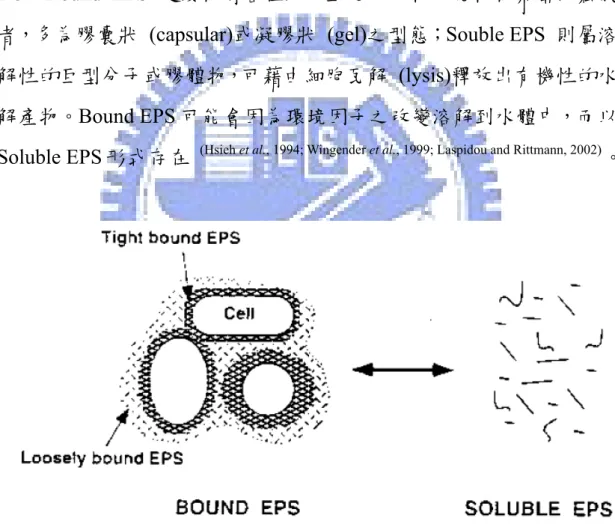
圖2-3 Bound EPS &Soluble EPS ...7
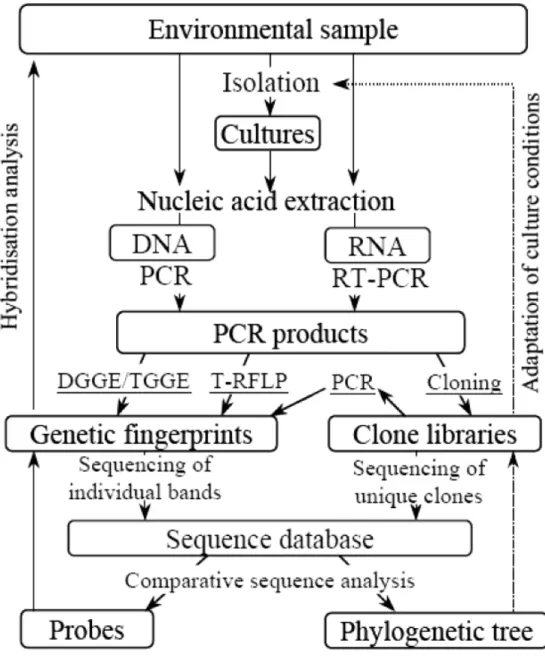
圖2-4 微生物族群在組成和結構分析上之步驟...10
圖3-1 實驗架構 ...15
圖3-2 反應槽實際照片 ...16
圖3-3 反應槽利用電腦自動控制示意圖...17
圖3-4 EPS 之萃取流程 ...21
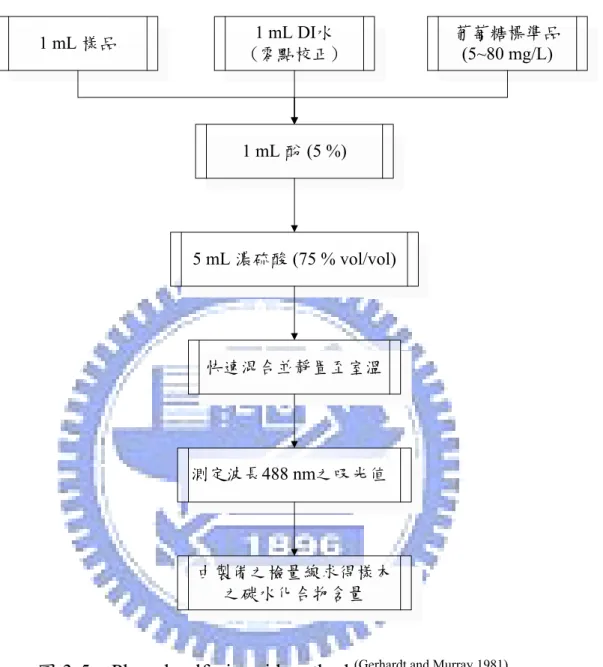
圖3-5 Phenol-sulfuric acid method...22
圖3-6 Bradford method...23
圖3-7 聚合酶鏈鎖反應操作條件 ...25
圖4-1 不同套組萃取 DNA 後之 agarose gel 圖譜 ...30
圖4-2 不同萃取套組進行 PCR 分析後之 agarose gel 圖譜 ...31 圖4-3 不同萃取套組進行 DGGE 分析後之圖譜...31 圖4-4 不同引子對之 PCR 產物 agarose gel 圖譜 ...32 圖4-6 反應槽前端是否架設選種槽之溶解性 EPS 濃度...36 圖4-7 反應槽前端未架設選種槽之 EPS 蛋白質與多醣類濃度變化36 圖4-8 反應槽前端架設選種槽隨時間變化之 DGGE 圖譜...39
IX 圖4-9 反應槽前端未架設選種槽隨時間變化之 DGGE 圖譜...40 圖4-10 反應槽前端架設選種槽之菌群結構親緣分析...42 圖4-11 反應槽前端未架設選種槽之菌群結構親緣分析...43 圖4-12 反應槽 SRT 為 30 天之 MLSS 變化 ...44 圖4-13 反應槽操作在 SRT 為 30 與 60 天之溶解性 EPS...45 圖4-14 SRT 為 30 天之反應槽隨時間變化之 DGGE 圖譜...47 圖4-15 SRT 為 30 天之菌群結構親緣分析...49 圖4-16 反應槽 SRT 為 60 天之 MLSS 變化 ...50 圖4-18 SRT 為 60 天之反應槽隨時間變化之 DGGE 圖譜...52 圖4-19 薄膜上菌種鑑定隨時間變化之 DGGE 圖譜...56 圖4-20 薄膜之菌群結構親緣分析 ...58
X
表目錄
表2-1 細菌系統分類學之種類與特性 ...8 表2-1 細菌系統分類學之種類與特性-續...9 表3-1 濃縮合成水樣之組成及濃度 ...19 表3-2 本實驗使用 primer 之目標族群及其序列 ...25 表3-3 DNA 序列長度對應所需之膠體濃度 ...27 表3-4 變性梯度膠所使用之各化學物質...28 表4-1 不同套組萃取 DNA 後之核酸定量結果 ...30 表4-2 反應槽前端架設選種槽隨時間變化之 Cs 値...41 表4-3 反應槽前端未架設選種槽隨時間變化之 Cs 値...41 表4-6 SRT 為 30 天之反應槽隨時間變化之 Cs 値...48 表4-8 SRT 為 60 天之反應槽隨時間變化之 Cs 値...53 表4-9 薄膜表面族群相似度分析之 Cs 値...571
第一章 前言
1.1 研究緣起
生物薄膜反應槽 (Membrane Bioreactor, MBR)是由傳統活性污泥 程序與薄膜技術所結合的一種程序,由於薄膜的高效率分離作用,因 此可成為分離單元中之重要角色。現今大量應用於廢污水生物處理, 其有佔地空間小、處理效率高等優點,且近年來薄膜成本逐漸下降, 使 MBR 相對於其他處理程序有其優勢性。目前,已有超過 500 個 MBR 在世界各地實廠運作 (Stephenson et al., 2000) 。 生物薄膜反應槽之應用上,薄膜積垢現象為其操作及設計之問題 點,至今仍無法有效排除,且對於積垢形成機制亦尚未完全釐清。在 MBR 系統操作中,多半可藉由控制工程參數,如:通量、曝氣量、 透膜壓力 (transmembrane pressure, TMP)等,來降低薄膜積垢現象的 發生。 生物處理程序中微生物存在的種類是影響處理效能的關鍵,然 而,目前對於生物薄膜反應槽中所發生的微生物相變化,以及微生物 相與薄膜積垢的相互關係,始終無法得到解答。隨著分子生物技術的 發展,利用PCR / DGGE 技術作為微生物相的鑑定工具,並藉以得知 系統中優勢菌種的分析方法,已逐漸應用於廢水生物處理之研究上, 故本研究亦可透過此項分析技術,追蹤反應槽在操作過程中的菌群結 構變化,探討菌相與水質變化的關聯性。2
1.2 研究目的與內容
本研究目的為利用分子生物技術監測微生物族群結構,透過PCR 結合 DGGE 的方式追蹤反應槽菌群之消長,以定序結果獲知系統中 的菌相組成及親緣關係,並探討微生物菌群變化和薄膜積垢的相關 性。 本研究內容包括: 1. 反應槽前端是否添加選種槽所造成之菌相差異。 2. 反應槽操作於不同 SRT 下所造成之菌相差異。 3. 反應槽懸浮生長與薄膜附著生長之菌相變化。3
第二章 文獻回顧
2.1 薄膜生物反應槽
傳統污水生物處理法大多採用活性污泥法,這種方式在控制操作 上容易發生絲狀菌繁殖以致污泥產生膨化現象,使污泥漂浮在水中而 無法藉著重力沈澱分離,進而造成處理水水質的惡化。近年來,薄膜 生物反應槽愈來愈受到重視。 (Ludzack and Ettinger, 1962; Graun et al., 1994) 圖2-1為活性污泥及MBR 之處理程序流程。 由圖2-1 可知,MBR 比起傳統的活性污泥反應槽,主要優點有: 1.節省用地面積,提高土地使用效率;2.操作、管理、維護更加容易; 3.節省能源之操作;4.較低的污泥產生量;5.較佳的出流水水質;6. 增加生物單元之處理效能;7.提昇氮、磷等營養物質之去除效率;8. 提昇難分解有機物之分解、去除效率。 圖2-1 活性污泥及 MBR 之處理程序流程
4
2.2 薄膜積垢
薄膜積垢形成原因相當複雜,各積垢因子之間關係交錯,目前在 探討 MBR 系統的積垢原因時,仍未有統一而完整的理論被建構出 來。MBR 是一個複雜的反應系統,因其中的生物組成包含許多不同 的微生物及其分泌之胞外物質。此外不同的處理系統,也會有不同的 積垢形成。(Chang and Lee, 1998; Chang et al., 2002; Li and Yang, 2007; Zeng, 2007) 各積垢影響因子之間,大多有相互影響之關係存在,如圖2-2 所示。 圖2-2 薄膜積垢因子 (Chang et al., 2002)
2.2.1 薄膜生物積垢
生物積垢是指微生物沉積在膜面上所形成的積垢,事實上生物積 垢就是生物膜的問題,因為當細菌一旦附著在逆滲透膜表面即會開始 成長,並逐漸發展成為黏泥狀的生物膜。在微生物附著於薄膜之前,5 腐質物、脂多醣類、蛋白質、微生物及其它物質等巨分子會先吸附在 膜面而形成條件膜。這些吸附物質形成的薄層會逐漸累積而覆蓋薄膜 表面,使薄膜表面原來的特性遭到改變,例如靜電荷和表面張力,而 且通常都是使膜面成為略帶負電性的狀態。(經濟部工業局, 2000) 在微生物方面可能影響生物膜形成的因素包括微生物種類、細胞 數目、養份狀態、親疏水性、表面電荷以及胞外聚合物 (extracellular polymeric substances, EPS)等。
在已積垢的薄膜表面曾經被發現的細菌有桿菌 (Bacillus)、微球
菌 (Micrococcus) 、 乳 酸 桿 菌 (Lactobacillus) 、 假 單 孢 菌
(Pseudomonas)、分枝桿菌(Mycobacterium)等,而真菌方面已被確認的 有鐮孢菌 (Fusarium)、青黴菌 (Penicillium)及木霉菌 (Trichoderma) 等。 大多數的細菌都帶有負電性,因此比較不容易附著在表面帶有負 電荷的膜面 (相對於不帶電或帶正電的膜面而言)。除了帶電性之外, 微生物細胞和膜面之間的疏水作用 (hydrophobic interaction)也是很 重要的影響因素。雖然帶有負電性的細胞和帶有負電性的逆滲透膜之 間,會因荷電性相同而互相排斥,但是細菌的鞭毛為疏水性,其和逆 滲透膜表面之間的疏水位址會有疏水作用,因而互相吸引並附著於膜 面上。
2.2.2 薄膜積垢可行之解決方法
為有效解決薄膜積垢現象,有以下幾個方法可供處理: 1. 對進流水做適當的前處理Al-Malack and Anderson, (1996)指出,在 MBR 系統中添加混凝劑 使小顆粒形成較大的顆粒,以快速掃過薄膜表面;Park et al., (2005) 提出在系統中添加鐵有助於使鐵氧化菌降解硫化氫氣體。此外,Kim
6
and Lee, (2003)指在生物處理系統中添加吸附劑可以減少有機物,學
者指出添加PAC 可以有效的降低 SMP 和 EPS。
2. 增加適當的物理或化學清洗步驟
物理清洗通常為利用液體或氣體反沖洗 (Kroner et al., 1984; Nikolov et al.,
1993) 。學者指出利用物理清洗方法中的氣體所產生的剪力來移除薄膜
表面的積垢物 (Chua et al., 2002; Hong et al., 2002) ,也有研究指出在操作條件中
結合反沖洗將會得到較佳的結果(Vallero et al., 2005; Zhang et al., 2005) 。在 MBR 系統中,為有效移除有機和無機積垢物,利用化學清洗方法中的酸洗 是必須的 (Choo et al., 2000; Lee et al., 2001; Kang et al., 2002) 。
3. 降低通量
降低通量通常可以減少積垢現象。而降低通量的方法通常分為兩
種,即Cho and Fane, (2002)提出之次臨界通量下操作與 Howell et al.,
(2004)提出之間歇操作。 4. 對薄膜表面進行改質
Doyle, (1991)指出由於薄膜材質和細菌間的疏水性交互作用,所
以採用親水性薄膜將會是減少積垢的一種方法 (Viero et al., 2007) 或者是使
薄膜表面較為親水 (Ma et al., 2000; Asatekin et al., 2006) 。使薄膜表面較為親水的
方法有許多種,例如:薄膜表面短暫改質 (Kim et al., 1988; Jonsson and Jonsson,
1991) 、在薄膜表面coating 親水性聚合物 (Stengaard, 1988; Hvid et al., 1990) 及利
用UV 照射光嫁接單體到薄膜上 (Yan et al., 1988; Nystrom and Jarvinen, 1991; Yamagishi et al., 1995; Ulbricht et al., 1996) 等。
2.2.3 胞外聚合物
Chang and Lee, (1998)指出胞外聚合物被認為是重要的積垢物與 薄膜積垢的參數之一。此外,Tansel et al., (2006)亦表示 EPS 和薄膜
7
et al., (2006)即指出薄膜積垢程度和薄膜材質有很大的關係,例如:在
實場廢水操作中PE 膜之積垢遠比 PVDF 膜要快的多。
EPS 以多醣體 (polysaccharide)和蛋白質 (protein)為主要成份,多
醣體為 EPS 較主要的物質,而蛋白質是較次要的成份,通常都以醣
蛋白 (glycoprotein)的形式存在,胞外物質具有高負電荷密度,胞外
物質大部份為親水性物質 (Nguyen and Westerhoff 2005) 。
EPS 可分成兩種型式,分別為 Bound EPS 和 Soluble EPS,如圖 2-3。Bound EPS 定義為附著在細胞上之胞外物可能較為緊密或鬆散 者,多為膠囊狀 (capsular)或凝膠狀 (gel)之型態;Souble EPS 則屬溶 解性的巨型分子或膠體物,可藉由細胞瓦解 (lysis)釋放出有機性的水 解產物。Bound EPS 可能會因為環境因子之改變溶解到水體中,而以 Soluble EPS 形式存在 (Hsieh et al., 1994; Wingender et al., 1999; Laspidou and Rittmann, 2002) 。
8
2.3 分子生物技術於環境工程上之應用
傳統微生物菌群研究是以分離純化和實驗室培養等方法為主,藉
由 觀 察 菌 株 的 外 觀 型 態 (morphology) 、 生 理 特 徵 (physiological
characteristics)、生化特性 (biochemical characteristics)和代謝功能等特
性來加以分類,如表2-1 所列。 表 2-1 細菌系統分類學之種類與特性 (Busse et al., 1996 ) Categorie Examples Cultural Colony morphology Color of colonies Fruiting bodies Mycelia Morphological Cell morphology Cell size Motility Flagellation type Reserve material Gram stain Acid-fast stain Physiological Temperature range pH range Salinity tolerance Biochemical
Carbon source utilization Oxidation of carbohydrates Enzyme profile Inhibitory test Selective media Antibiotics Dyes
9 表2-1 細菌系統分類學之種類與特性-續 (Busse et al., 1996 ) Categorie Examples Chemotaxonomic Fatty acids Polar lipids Mycolic acids Lipopolysaccharide composition PAGE of lipopolysaccharide Cell wall diaminoacids
Cell wall aminoacid composition Whole cell sugars
Cellular pigments
Quinone system Polyamine content Whole cell protein PAGE
Genotypic
DNA base ration (G+C -content)
Random amplified polymorphic DNA (RAPD) Restriction fragment length polymorphism (RFLP)
Pulsed field gel electrophoresis of DNA fragments DNA probe Phylogenetic DNA:DNA hybridization DNA:rRNA hybridization 16S rRNA sequence 23S rRNA sequence
Sequence of the β-subunit of ATP-synthase GroEL (Chaperonin) sequence
10
然而以傳統培養法來研究微生物菌群,面臨了諸多限制,主要因 素是大部分的微生物仍無法以人工方式分離培養。據估計,目前可以
人工方式在實驗室培養的微生物僅佔全部微生物族群的0.1 % ~ 10 %
(Amann et al., 1995; Ranjard et al., 2000) 。圖 2-4 為微生物族群在組成和結構分析上
之步驟 (Muyzer and Smalla., 1998) 。
11
在環境工程方面,分子生物技術最廣泛的應用於鑑定生物處理系 統中活性污泥之菌相,且逐漸成為目前活性污泥微生物鑑定的主要趨 勢。目前較常應用於環工之分子生物技術包括螢光原位雜合技術 (Fluorescence in situ Hybridization, FISH)、瓊脂糖凝膠電泳法、限制酵
素 法 (T-RFLP) 、 變 性 梯 度 凝 膠 電 泳 法 (Denaturing Gradient Gel
Electrophoresis, DGGE)、聚合酶連鎖反應 (Polymerase Chain Reaction,
PCR)、流式細胞儀 (Flow Cytometry)、生物晶片等。 (Amann et al., 1990;
Abughararah and Randall, 1991; Bond et al., 1995; Wagner et al., 1995; Liu et al., 1996; Regan et al., 2002)
這些應用於環工上的分子生物技術具有傳統鑑定細菌法上所不 及的優勢,除可精準的定性與定量外,更可藉由這種優勢建立物種與 環境間的模式關係,以落實於實務之應用。
2.3.1 聚合酶鏈鎖反應
聚合酶鏈鎖反應,簡單來說,就是利用酵素將特定基因在體外或 是試管內 (in vitro)進行人工的複製合成。此一方法可使少量的 DNA
在微量試管中擴增 (amplify)至 106倍以上。
PCR 的原理為:藉由加入適當的引子對 (primer set)、dNTP (即
為 ATP、TTP、GTP、CTP 等 DNA 的組成成分)、DNA 聚合酵素
(polymerase)等反應物,及目標序列 (template),經一連串循環的升溫
降溫控制,使目標DNA (template)經變性反應 (Denature)、鍊合反應
(Annealing)、延長合成反應 (Extension)循環進而放大複製目標序列。 1. 變性反應 (Denaturation):模版 DNA 在高溫狀態下,由原先互補之
兩股分離為單股DNA。
2. 鍊合反應 (Annealing):使所加入之正向引子 (forward primer)、逆 向引子 (reverse primer)與變性後單股目標 DNA 鍊合配對。
12 3. 延長合成反應 (Extension):合成模板上的互補股。 PCR 的循環操作每次可使 DNA 的量增加一倍,在 n 次的重複操 作的次數後,DNA 增加的量將會是 2n。在理想的聚合酵素鏈鎖反應 條件下,DNA 是以幾何級數增加,一個 DNA 分子若重複循環 25 次, 那麼 DNA 的分子數將會擴增到 225 ≒ 106 個分子。這個 DNA 的量 已足夠在瓊脂糖凝膠電泳中觀察到 (陳, 1999) 。
2.3.2 變性梯度凝膠電泳
變性梯度凝膠電泳主要是運用於族群分析之研究。由於各種真細 菌的核醣體核酸序列保留性相當高,若要分析樣品中生物之多樣性, 可設計所需要之引子對,並藉由PCR 將真細菌共同擁有之 16S rDNA 基因中變異的區段進行大量複製,並使雙股 DNA 一端帶有 GC clamp,再將欲分析樣品注入 DGGE 中並通電,具有差異性且帶負電 之雙股DNA 序列,受到電場驅動而泳動通過不同程度的變性梯度凝 膠時,會產生不同程度之局部變性,當變性至無法被電流推動而通過 聚丙醯胺凝膠所形成之網格時,具有不同序列之雙股DNA 將被滯留 在不同變性梯度之凝膠中,形成分開的單一亮帶。根據凝膠中 DNA 停留位置的高低不同,便可區別不同序列的 PCR 產物;換言之,停 留在相同水平位置的亮帶,一般被視為具有相同的DNA 序列。 但利用DGGE 進行相關研究時仍有其侷限性,即欲分析之 DNA 片段長度必須小於 500 bp,若欲分離之 DNA 片段長度大於 500 bp 時,不同菌群就不易被分開,使得核甘酸次序解析只能提供有限之資 訊。13
2.4 分子生物技術於生物薄膜上之應用
隨著分子生物技術在環境工程上的應用,近年來開始有學者把此 技術應用到生物薄膜上。
首先,有學者於 2003 年提出,反應槽不論在何種操作條件下皆
會有許多不同的微生物相存在 (Stamper et al., 2003) 。Klatt (2003) 學者研究
亦發現,在反應槽操作的前七天,微生物菌群有些微菌相變動的現 象,且反應槽中以 Flavobacterium-like bacterial 族群佔大多數,佔總 亮帶強度的50 %以上。 Miura et al.,(2007)亦表示,反應槽進流水的組成對於微生物的菌 群結構將會有很大的影響。因此,LaPara et al.,(2006)利用人工合成的 簡單基質作為進流水來源,指出即使是簡單的基質亦可有複雜的微生 物相變化。並探討不同薄膜孔徑,0.2μm 與 0.45μm 之反應槽菌相 變化,發現即使薄膜孔徑不同,但反應槽中的微生物相卻大致相同。 且與2003 年 klatt 學者有相同的發現,即反應槽操作在前五天會有菌 相變動的現象。Choi et al.,(2007)則利用實廠家庭廢水作為進流水來 源,比較沉浸式生物薄膜反應槽之NF 與 MF 薄膜,得到的反應槽微 生物相是大致相同的,與 LaPara 學者之研究相符。所以,由 LaPara 與 Choi 學者的研究得知,反應槽所使用的薄膜孔徑並不影響反應槽 中的微生物相。 Jinhua et al.,(2006)提出,反應槽中懸浮生長之微生物相與薄膜表 面上附著生長之微生物相有很大的差異。薄膜表面上的微生物相以 Gammaproteobacteria 佔大多數。因學者僅提出有極大的差異,促使本 研究想進一步瞭解差異性背後所代表之意義。且學者表示薄膜表面之 微生物相以 Gammaproteobacteria 佔大多數,但 Gammaproteobacteria 是利用傳統微生物培養法能佔有優勢之族群,在利用分子生物技術監
14 測下,是否薄膜表面上之微生物相仍是以 Gammaproteobacteria 佔大 多數亦是本研究欲探討的部分。 Zhang et al.,(2006)研究在不同污泥停留時間下,SRT 為 10 天之 薄膜積垢程度大於SRT 為 30 天。但學者僅利用物化實驗證明其所觀 察到的現象,並沒有進一步探討反應槽中之菌相差異,也因此使本研 究想利用分子生物技術探討在不同 SRT 下,反應槽內的微生物相是 否有其差異,因而影響薄膜積垢之程度。
15
第三章 材料與方法
本研究將利用分子生物技術監測微生物族群結構。利用 PCR 結 合DGGE 的方式追蹤反應槽於 1. 反應槽操作前端是否添加選種槽; 2. 反應槽操作於不同 SRT 下;3. 反應槽懸浮生長與薄膜表面附著生 長之菌群之消長。3.1 實驗架構
圖3-1 實驗架構16
3.2 實驗設備
3.2.1 沉浸式生物薄膜反應槽
本研究之生物薄膜反應槽是以沉浸式操作,其主體利用不透水壓 克力板製作,主要設備包括曝氣槽、薄膜單元、監控設備、自動酸鹼 加藥系統及蠕動幫浦。本實驗是將MF 平板膜置於曝氣裝置上方,以 液位控制器 (level sensor)控制 MBR 的工作體積在 30 L,並將 pH、 DO、TMP 及溫度等數值經由偵測器紀錄於電腦中,以控制微生物之 最佳生長環境。此外反應槽利用電腦控制固定的操作通量,並觀察 TMP 隨時間的變化,以瞭解薄膜操作通量與 TMP 之關係,圖 3-3 為 反應槽利用電腦自動控制示意圖。圖3-2 為反應槽實際操作時之照片。 圖3-2 反應槽實際照片17
3.3 反應槽操作內容及條件
3.3.1 污泥來源及進流水成分
污泥植種來源為國立交通大學水質淨化廠,原因在於其處理對象 為校區內之生活污水,與人工配製之合成水樣性質較為相近,同時也 因地理位置之關係,採集較為方便。 本研究所採用之進流水為模擬實際都市污水之人工合成水樣,可 提供微生物生長所需之碳、氮、磷及微量元素來源。首先配製一濃縮 水樣 (concentrated feed)於 4℃冰箱中,在進流入 MBR 前,以自來水 稀釋至所需之濃度,其 COD 約為 400 ± 10 mg/L。濃縮水樣之成份 如表3-1 所示 (李, 2006) 。本研究之進流水成份由於較一般實際污水易 生物分解,故在此MBR 中之污泥特性可能與實際水樣中有所差異。19 表3-1 濃縮合成水樣之組成及濃度 Compound Concentration (mg/L) Sodium acetate 2527 Corn starch 150 Beef extract 250 NH4Cl 670 KH2PO4 154 MgSO4.7H2O 355 CaCl2 73 FeSO4.7H2O 87 CuCl2.2H2O 0.35 MnCl2.4H2O 0.63 ZnSO4.7H2O 0.66 CoCl2.6H2O 0.15 Na2MoO4.2H2O 0.08 H3BO3 0.124 KI 0.166
3.3.2 反應槽操作參數
本研究之反應槽操作體積為30 L,有效薄膜面積為 0.2 m2,有機 物負荷濃度為1.2 kg COD/m3 day,溶氧控制在 6 ~ 7 mg/L,pH 控制 在6.8 ~ 7.2,藉由配製 1 %鹽酸做調整,污泥停留時間分別為 10 天、 30 天及 60 天,即每日於反應槽排泥 3 L、1 L 及 500 mL。20
3.4 反應槽水質分析
爲確實瞭解反應槽之操作狀況,因此進行必要之水質分析,其項 目包含污泥濃度(mixed liquor suspended solids, MLSS)、總有機碳(total organic carbon, TOC)、EPS、顆粒粒徑大小分佈等。其中 MLSS 分析
採 用 過 濾 性 餘 留 物 法 ;TOC 以 TOC 分 析 儀 分 析 ; EPS 是 以
phenol-sulfuric acid method 及 Bradford method 分別針對多醣類與蛋白 質兩部份做吸光值分析;顆粒粒徑大小分佈採用雷射粒徑分析儀測 量。
3.4.1 胞外聚合物分析
活性污泥中 EPS 主要可分為兩部份,第一部份為藉由 4000 rpm
離心20 分鐘 (4℃)所得到之溶解性 EPS,第二部份為藉由污泥萃取所
得到之萃取性EPS,其萃取方法如圖 3-4 所示 (Fang and Liu, 2002) 。EPS 的
組成會隨著污泥之來源及萃取方法不同,而有所差異,不過在此研究
中,主要針對多醣類與蛋白質做為主要 EPS 之分析對象。多醣類之
分析是參照phenol-sulfuric acid method,其是以葡萄糖做為標準品,
方法如圖 3-5 所示 (Gerhardt and Murray, 1981) 。而蛋白質之分析則是參照
Bradford method,其是以 bovine serum albumin (BSA)做為標準品,方
21 10 mL 污泥 3500 MWCO 透析2天 上澄液過濾 0.2 μm 濾膜 20000 g 離心 4 ℃ ,20 min 4 mL 1N 氫氧化鈉 4 ℃ ,3 hr 0.06 mL 甲醛 (36.5 %) 4 ℃ ,1 hr
22 1 mL DI水 (零點校正) 由製備之檢量線求得樣本 之碳水化合物含量 測定波長488 nm之吸光值 快速混合並靜置至室溫 5 mL 濃硫酸 (75 % vol/vol) 1 mL 酚 (5 %) 1 mL 樣品 葡萄糖標準品(5~80 mg/L)
23 0.1 mL DI水 (零點校正) 由製備之檢量線求得樣本 之蛋白質含量 測定波長595 nm之吸光值 均勻混合並於室溫下反 應5~45 min 3 mL Bradford 試劑 0.1 mL 樣品 BSA標準品 (0~0.5 mg/mL) 圖3-6 Bradford method
*BSA : bovine serum albumin (SIGAMA®, P 0834)
*Bradford reagent: Brilliant blue G in phosphoric acid and methanol. (SIGAMA®, B 6916)
24
3.5 反應槽微生物菌相分析
3.5.1 DNA 萃取
活性污泥樣本由於所具有之雜質及膠羽成分會使溶菌素不易破 壞細胞壁,故必須先經過前處理,以此排除掉因細胞萃取效果不佳所 造成的誤差;本研究中在針對活性污泥樣本DNA 萃取時,會先以 23G 的針頭抽放數次後將較大的污泥打散,再以 27G 的針頭抽放數次後 將較小的污泥打散,以此將細胞從膠羽結構中釋放出來,之後再用 1× PBS 清洗兩次將其餘的干擾物質清除後始進入萃取步驟。使用 VIOGENE 套組試劑萃取樣本之 DNA (Blood & Tissue
Genomic DNA Extraction Miniprep System, VIOGENE),其隨附之標準 萃取步驟如下: 1. 以 8000 rpm 離心 10 分鐘收集細菌 pellet 於 1.5 mL 微量離心管中。 2. 加入 200 μL LYS Buffer,混合均勻,於 37℃下作用 2 小時。 3. 加入 200 μL EX Buffer 及 20 μL proteinase K。 4. 60℃作用 30 分鐘,約每 5 分鐘均勻混合;70℃下靜置 30 分鐘。 5. 加入 420 μL 98%乙醇混合。
6. 將所有混合物置放於套組試劑內所附之 spin column 中,並將 spin column 放入 collection tube。
7. 以 8000rpm 下離心 2 分鐘後移除離心液。 8. 添加 500 μL Washing Buffer,以 8000 rpm 離心 2 分鐘,重複兩次。 9. 以更高速(9000 rpm) 離心至乾。 10. 將 spin column 放入新的 1.5 mL 微量離心管中。 11. 加入 200 μL 70℃無菌水,靜置 3 到 5 分鐘。 12. 以 8000 rpm 離心,獲得 DNA 樣本;最終保存於 -20℃下。
25
3.5.2 聚合酶鏈鎖反應
3.5.2.1 引子 (Primer)
本研究中聚合酶鏈鎖反應主要所使用的primer set 為 357f-gc 搭配
518r (Choi et al., 2007; Miura et al., 2007) ,此 primer set 可針對所有細菌的 16S ribosomal DNA 位於 V3 region 之序列進行放大。在本研究中,此 primer 主要用來鑑定反應槽中整體細菌族群的分析,引子序列以及目標族群
如表3-2。
表 3-2 本實驗使用 primer 之目標族群及其序列
Primer Specificity Target site Sequence (5’ 3’)
357f-gc Bacteria 16S CGCCCGCCGCGCGCGGCGG GCGGGGCGGGGGCACGGG GGGCCTACGGGAGGCAGC AG 518r Universal 16S ATTACCGCGGCTGCTGG
3.5.2.2 反應操作條件
本研究中聚合酶鏈鎖反應操作時之溫度及時間如圖3-7 所示。 圖3-7 聚合酶鏈鎖反應操作條件26
3.5.3 電泳分析
3.5.3.1 瓊脂膠糖凝膠電泳
為確認經由 PCR 後所得到的產物是目標的基因序列,因此以瓊 脂膠糖凝膠電泳分析,電泳後所跑出的結果,其亮帶若出現在目標長 度範圍中,即可判斷所得到的產物是正確的,才可進行 DGGE 的分 析;步驟如下: 1. 將 PCR 產物取出 75 μL 以紫外光分光光度計,分別以 260 nm 及 280 nm 波長偵測進行核酸數量及純度的定量,所得到的濃度再以適當的體 積比將DNA 濃度稀釋至 25~40 μg/μL。 2. 取 3 μL 稀釋後之 PCR 產物混合 3 μL 1X Loading Dye。3. 將 1.5% agarose gel 置放入含 TAE Buffer 的電泳槽中,並使 TAE Buffer 蓋過膠體。
4. 將 DNA- Loading Dye 混和液注入 1.5% agarose gel 的 well 中。 5. 並以 100bp ladder 之 DNA Marker 做比對依據。
6. 以 100V 電壓進行電泳。
7. 當 Loading Dye 移動至膠體的三分之一時停止電泳。
8. 以 0.5 μg/mL Ethidium Bromide 進行 DNA 染色,並以紫外光顯像。
不同長度之PCR products 所使用的 agarose 膠體濃度有所差異,
27
表3-3 DNA 序列長度對應所需之膠體濃度
Gel Concentration (%) DNA Size (kbp)
0.5 1 – 30 0.75 0.8 – 12 1 0.5 – 10 1.25 0.4 – 7 1.5 0.2 – 3 2 - 5 0.01 – 0.5
3.5.3.2 變性梯度凝膠電泳(DGGE)
本研究中變性梯度膠凝膠電泳為水平式DGGE,而所配製的變性 範圍為20%至 80%,並依實驗需求而做梯度範圍變動。操作步驟如下: 1. 取 40 μL 聚合酶連鎖反應後之產物混合 10 μL 6X Loading Dye。 2. 加入預熱 60℃ TAE Buffer 於電泳槽中,使膠體除 well 外完全浸於 Buffer 內。3. 將混合之 PCR 產物及 Loading Dye 注入 well 中。 4. 以 200V 電壓進行電泳 5 小時。
5. 最後利用 0.5 μg/mL Ethidium Bromide 進行 DNA 染色,並以紫外光 顯像。
配製變性梯度膠時,首先需配製變性梯度之最高及最低變性劑濃 度 (High/Low Denature Density Solution),而後利用 gradient former (Bio-Rad)進行變性梯度之配製。表 3-4 為膠體配製時所使用之各化學 物質。
28 表3-4 變性梯度膠所使用之各化學物質 Constituents 20% Denature solution 80% Denature solution 40 % Acrylamide/Bis (mL) 5 5 50 × TAE Buffer (mL) 0.4 0.4 Formamide (mL) 1.6 6.4 Urea (g) 1.64 6.56 to H2O (mL) 20 20 10 % APS (mL) 0.2 0.2 TEMED (mL) 0.02 0.02
3.5.4 Acrylamide/Bis 膠體之 DNA 純化
以引子 357f-gc 及 518r 進行 16S rDNA 放大之 PCR 產物,利用 DGGE 分析後再將 DGGE 膠體上相異的亮帶分別切下後置於無菌水 中,經過”freezing-and-thawing”過程以獲取目標 DNA 片段;爲檢視切 下的亮帶,必須重複進行 PCR-DGGE 與切膠純化的分析直至確定為 單一亮帶為止。最後,所純化的DNA 再利用無 GC clamp 的引子 (357f 及518r)將 DNA 放大後,方可進行後續實驗。3.5.5 親緣分析
本研究中 DNA 序列定序分析乃委託明欣生物科技有限公司(Mission Biotech, Taiwan) 進 行 。 將 取 得 之 序 列 結 果 利 用 NCBI (National Center for Biotechnology Information)網站所提供 Nucleotide Blast 服務,查詢定序結果與已知序列進行比對,並利用 MEGA 4 軟 體繪製出親緣樹 (Phylogenetic tree)。
29
第四章 結果與討論
4.1 分生技術之建立
本研究之菌種鑑定分析指標為16S rDNA。先萃取環境樣本中的 DNA,再利用 DGGE 做膠體回收,並由序列比對的結果來瞭解 MBR 系統操作中微生物的親緣關係。4.1.1 DNA 之萃取
16S rDNA 分析之首要工作即萃取環境樣本中的 DNA,若 DNA
受到污染或在萃取過程中被干擾,則對後續 DGGE 實驗會有很大的
影響。
本研究兩種樣本由不同時間點之MBR 取樣得來,分別利用兩組
商業套組試劑:Blood & Tissue Genomic DNA Extraction Miniprep
System (VIOGENE, Taiwan)及 UltraCleanTM Soil DNA Isolation Kit
(MO BIO, USA)來萃取環境樣本之 DNA,並比較萃取效果之優劣。圖 4-1 為不同套組萃取 DNA 後之 agarose gel 圖譜,其中 DNA marker 的
長度為每條100 bp 由下往上遞增至 1000 bp,接著往上為 1500 bp、
2000 bp 與 3000 bp。由圖 4-1 得知 MO BIO 套組所萃取之 DNA 亮帶
清晰,即 DNA 性質穩定;而 VIOGENE 套組所萃取之 DNA 亮帶較
不清晰,且亮帶似有脫尾的情形,推測原因應為 VIOGENE 套組是以
無菌水回溶DNA,不若 MO BIO 套組有提供回溶 DNA 的溶液。
表 4-1 為不同套組萃取 DNA 後之核酸定量結果,得知針對不同
樣本所得到之萃取效果不同。以 MO BIO 套組萃取,在樣本 1 中的
DNA 可能受蛋白質干擾 (A260/A280 大於 1.8),而在樣本 2 則無蛋白 質干擾且得到高純度 (A260/A280 趨近 1.8)及高濃度;在 VIOGENE
30 本2 則可能受蛋白質干擾。 進一步,將兩種萃取方式所得之DNA 進行 PCR 及 DGGE 實驗, 得到圖 4-2 及圖 4-3。由圖中可知兩種商業套組所得之菌相並無明顯 差異,又因 VIOGENE 套組在價格上較有競爭力,因此本研究選擇 VIOGENE 套組進行後續實驗。
圖4-1 不同套組萃取 DNA 後之 agarose gel 圖譜
(MO BIO:M;VIOGENE:V) 表4-1 不同套組萃取 DNA 後之核酸定量結果 Sample 1 2 濃度* A260/A280 濃度* A260/A280 MO BIO (M) 168 2.54 380 1.75 VIOGENE (V) 269 1.74 186 2.1 *DNA 濃度單位:ng/μL Marker M1 V1 M2 V2
31 圖4-2 不同萃取套組進行 PCR 分析後之 agarose gel 圖譜 (MO BIO:M;VIOGENE:V) 圖4-3 不同萃取套組進行 DGGE 分析後之圖譜 (MO BIO:M;VIOGENE:V) M1 Marker V2 M2 V1 M1 V1 M2 V2
32
4.1.2 PCR
污泥樣本以968f-gc 與 1392r 引子對進行 PCR 實驗,如圖 4-4 (a) 所示,卻無法得到亮帶出現,但對照正控制組:一般的活性污泥樣本 即圖4-4 中的 AS,卻有亮帶出現,雖濃度不夠卻已證明在 PCR 的實 驗中所添加之試劑,如引子之合成是沒有問題的。 在嘗試過多種不同條件之實驗後,改變引子對為 357f-gc 與 518r。初始仍無法得到 PCR 產物如圖 4-4 (b)所示,推測原因為 DNA 模板的濃度,環境樣本組成成分過於複雜,當DNA 濃度過與不及皆 會影響PCR 之結果。由實驗結果得知,添加之 DNA 模板體積佔 PCR 反應總體積的六十分之一最為理想,如圖 4-4 (c)所示,即 PCR 反應 總體機為30 μL 時,DNA 模板之最適宜添加體積為 5 μL。 圖4-4 不同引子對之 PCR 產物 agarose gel 圖譜 (a:968f-gc/1392r;b 與 c:357f-gc/518r) 註:DNA marker 的長度為每條 100 bp 由下往上遞增至 1000 bp,接 著往上為1500 bp、2000 bp 與 3000 bp。 Marker V2 AS V1 V3 a b Marker V2 AS V1 V3 Marker V2 V1 V3 c33
4.2 添加選種槽對反應槽所造成之影響
在活性污泥反應槽設計中,為避免絲狀菌的產生,造成污泥在後 續沉降單元處理效果不好,會在反應槽前端增設選種槽 (selector), 選種槽之目的為提高食微比比例,使膠羽生成菌 (Zoogloea)能大量生 長,而絲狀菌則被抑制。 本研究所使用之選種槽體積為 500 mL,操作在好氧條件下,食 微比控制在。 本研究控制SRT 為 10 天,兩座反應槽,其中一座前端增加選種 槽單元,觀察其MLSS、TOC、EPS 及菌相變化。4.2.1 選種槽對反應槽水質分析之影響
圖4-5 為反應槽前端是否有架設選種槽之水質參數變化。圖 4-5 (a) 為 MLSS 隨時間之變化,由圖中可知反應槽前端有架設選種槽之 MLSS 為 2580 ~ 3500 mg/L,平均約 3087 mg/L;反應槽前端未架設 選種槽之MLSS 為 2480 ~ 3760 mg/L,平均約 3050 mg/L。得知兩反 應槽 MLSS 的變化不大,原因為 SRT 相同,與反應槽前端是否有架 設選種槽無關。34 Time (d) 0 5 10 15 20 25 30 35 40 45 50 55 60 65 70 ML SS (mg/L) 0 1000 2000 3000 4000 5000 6000 Selector No selector Time (d) 0 5 10 15 20 25 30 35 40 45 50 55 60 65 70 TOC remov a l (% ) 0 1 2 3 4 5 97 98 99 100 Pe rm eat e TOC (m g/ L) 0 2 4 6 8 10
selector (removal ratio) no selector (removal ratio) selector (permeate conc.) no selector (permeate conc.)
圖4-5 反應槽前端是否有架設選種槽之水質參數變化
(a)MLSS (b)滲出水 TOC 及 TOC 去除率
(b) (a)
35 本實驗之進流水為人工合成,TOC 約 140 mg/L。圖 4-5 (b)為反 應槽滲出水TOC 量及 TOC 去除率隨時間之變化,由圖中可知反應槽 前端有架設選種槽之滲出水TOC 為 0.9 ~ 1.6 mg/L,平均約 1.4 mg/L; 反應槽前端未架設選種槽之滲出水TOC 為 1.87 ~ 12.94 mg/L,平均 約 3.29 mg/L。反應槽前端有架設選種槽之 TOC 平均去除率約為 99 %;反應槽前端未架設選種槽之 TOC 平均去除率約 97.7 %。 圖4-6 為反應槽前端是否有架設選種槽之溶解性 EPS 濃度,由圖 可知,無論反應槽前端是否有架設選種槽,反應槽溶解性 EPS 中蛋 白質濃度高於多醣類濃度。圖 4-7 為反應槽前端未架設選種槽之 EPS 中蛋白質與多醣類濃度變化圖,圖4-7 中所示之總蛋白質濃度為溶解 性蛋白質與萃取性蛋白質濃度的總合,總多醣類濃度亦同。由圖 4-7 可知,反應槽 EPS 中多醣類濃度高於蛋白質濃度。而蛋白質與多醣 類的比值則介於0.4 ~ 1.5,Bura et al., (1998)曾研究指出,蛋白質與多 醣類的比值應介於0.2 ~ 5,與本研究結果相符合 。
36 selector no selector conce nt rat ion (mg /L) 0 5 10 15 20 25 30 35 soluble protein soluble polysaccharide soluble protein soluble polysaccharide 圖 4-6 反應槽前端是否架設選種槽之溶解性 EPS 濃度 Time (d) 0 5 10 15 20 25 30 35 40 45 50 55 60 65 Co nc en tra tio n (mg /L ) 0 50 100 150 200 250 300 350 400 450 500 Pro tein/p o lysa cc ha rid e 0 2 4 6 8 10 Total protein Total polysaccharide Protein/polysaccharide 圖4-7 反應槽前端未架設選種槽之 EPS 蛋白質與多醣類濃度變化
37
4.2.2 選種槽對反應槽所造成之菌相變化
圖4-8 為反應槽前端架設選種槽隨時間變化之 DGGE 圖譜,分別 於反應槽操作時間為3、9、18 與 50 天所採集的樣本,編號 A ~ N 表 示亮帶出現的位置及在附錄A 中可得知序列比對列表。 由圖4-8 配合附錄 A 可知,反應槽前端架設選種槽之菌種大部分是Betaproteobacteria、environmental samples 與 Bacteroidetes。
Betaproteobacteria 是 proteobacteria 細菌五支中的一支,由 16S rDNA 序列顯示 proteobacteria 是由紫色光合細菌演化而來,惟現今因 要適應各種環境,所以許多此類細菌已失去原有光合作用的能力,其
中Betaproteobacteria 大部分是屬於化學自營性細菌。Bacteroidetes 則
是 CFB group 的一部分,CFB group 是由 Cytophaga
、
Flavobacteria及 Bacteroidetes 等類細菌所組成。Bacteroidetes 屬於厭氧革蘭氏陰性 細菌,CFB group 細菌大多可分解非溶解性巨大分子。
隨著時間的變化,發現反應槽操作前 10 天之菌相較為接近,且
由 DGGE 的半定量原理知,亮度愈高表示濃度愈大,所以推論在反
應槽馴養期間,反應槽的菌相含有大量的菌種A、E 與 G,由定序結
果知為 Zoogloea oryzae,並發現菌種 B (Aquabacterium sp.)與 J (Uncultured bacterium)由一開始的高濃度漸轉淡。其中 Aquabacterium sp.是學者發現在飲用水配水管網生物膜中最佔優勢的菌種之一。
當反應槽操作達 18 天後菌種開始發生變化,已不同於操作初期
之菌相。雖已無法發現菌種 A 與 G,但菌種 E 的濃度卻漸漸變高,
此外增加的菌種中,菌種L (Rhodobacter gluconicum)極佔優勢,除了
菌種F (Flexibacteraceae bacterium)與菌種 H (Candidatus nitrotoga)以
外,其他增加的菌種經鑑定後多為無法培養的環境樣本。
38 性質是紫色不含硫光合異營菌。Alphaproteobacteria 亦是 proteobacteria 中的一支,且Alphaproteobacteria 是 proteobacteria 中較多菌株還保有 光合作用能力的一支。 圖4-9 為反應槽前端未架設選種槽隨時間變化之 DGGE 圖譜,分 別於反應槽操作時間為 3、16、37、51 與 65 天所採集的樣本,編號 A ~ P 表示亮帶出現的位置及在附錄 B 中可得知序列比對列表。 由圖4-9 配合附錄 B 可知,反應槽前端未架設選種槽之菌種中,
大部分是Betaproteobacteria、Bacteroidetes 與 environmental samples,
另外在反應槽操作第51 天新發現 Chlamydia,推測 Chlamydia 的出現 與圖4-7 蛋白質濃度的竄升有關係,且在此時也發現反應槽出現薄膜 積垢現象。 此外,反應槽豐富的菌相中有將近一半是無法利用傳統微生物培 養法偵測的environmental samples 亦驗證利用分子生物技術來監測反 應槽中的微生物菌相,除可節省時間外在菌種的鑑別上亦有相當的準 確性。 並且,由圖中得知菌種L (Zoogloea sp.)不但隨著時間的變化濃度 漸 漸 增 加 , 而 且 亦 是 反 應 槽 中 最 佔 優 勢 的 菌 種 , 另 外 菌 種 B
(Uncultured bacterium)與 O (Zoogloea sp.)雖濃度不高,但卻是隨著時 間的變化一直存在於反應槽中的,而在反應時間後期優勢性提高的尚
有 菌 種 J (Uncultured bacterium) 與 K (Uncultured Bacteroidetes
bacterium)。
比較圖4-8 與 4-9 得知,反應槽前端若增加選種槽,將可使菌相
單純化且多為助沉降性對反應槽有益的 Zoogloea oryzae,雖隨著時間 反應槽菌相將更為複雜,但有益於反應槽的菌種並不會因此而失去優 勢。
39 將定序後之序列與NCBI 序列資料庫進行比對,建構微生物相之親緣 演化樹,推測微生物相之演化關係,並利用軟體繪製親緣樹,如圖4-10 與4-11 分別為反應槽前端是否架設選種槽之菌群結構親緣分析。
3 9 18 50
A B C D E F G H I J K L M N 圖4-8 反應槽前端架設選種槽隨時間變化之 DGGE 圖譜40 A B C D E F G H I J K L M N O P
3 16 37 51 65
圖4-9 反應槽前端未架設選種槽隨時間變化之 DGGE 圖譜利用Dice, (1945)提出的公式可計算族群間的相似度。Dice index
為
Cs
=
a2+jb,其中j 表示樣本 A 和 B 相同的亮帶數目;a 表示樣本 A 的亮帶數目;b 表示樣本 B 的亮帶數目。Cs 値介於 0 ~ 1 之間。表 4-2 與 4-3 分別表示反應槽前端架設選種槽隨時間變化之 Cs 値與反應槽 前端未架設選種槽隨時間變化之Cs 値。 由表4-2 可知,反應槽在操作初期族群間的相似度完全相同,Cs 値為 1,學者提出當 Cs 値大於 0.85 時代表族群間的相似度為高度相41 似 (Lapara et al., 2002) 。當反應槽操作到第18 天,族群間的相似度銳減到 0.32,表示菌相有極大的轉變。但反應槽操作至第 50 天的Cs値為 1, 表示菌種完全相同。 表4-3 為反應槽前端未架設選種槽隨時間變化之 Cs 値,得知 Cs 値介於0.71 ~ 0.81 之間。表示反應槽雖有豐富菌相卻不影響反應槽菌 群結構的穩定性。 利用Cs 値來比較反應槽中 PCR-DGGE 指紋譜所代表的微生物族 群結構,亦有學者做相關研究,Lapara et al., (2000)發現反應槽之 Cs 値介於0.10 ~ 0.93;Konopka et al., (1999)則研究指出反應槽之 Cs 値 介於0 ~ 0.10。 表4-2 反應槽前端架設選種槽隨時間變化之 Cs 値
3 days 9 days 18 days
3 days
9 days 1
18 days 0.32 0.32
50 days 0.32 0.32 1
表 4-3 反應槽前端未架設選種槽隨時間變化之 Cs 値
3 days 16 days 37 days 51 days
3 days
16 days 0.81
37 days 0.74 0.71
51 days 0.81 0.79 0.79
圖4-10 反應槽前端架設選種槽之菌群結構親緣分析
圖4-11 反應槽前端未架設選種槽之菌群結構親緣分析
4.3 不同 SRT 下反應槽之水質分析與菌相變化
4.3.1 SRT 為 30 天之水質分析與菌相變化
反應槽操作在SRT 為 30 天時,MLSS 約 6060 ~ 7160 mg/L,平 均約6722 mg/L,如圖 4-12 所示。本實驗之進流水為人工合成,TOC 約140 mg/L。出流水 TOC 為 1.61 ~ 2.36 mg/L,平均約 1.83 mg/L; TOC 去除率平均約98.7 %。 圖4-13 表示當反應槽操作在 SRT 為 30 與 60 天之溶解性 EPS。 由圖4-13 可知,反應槽中溶解性 EPS 之蛋白質濃度高於多醣類濃度。 Time (day) 0 5 10 15 20 25 30 35 40 45 50 55 60 65 70 MLSS ( m g/L) 0 1000 2000 3000 4000 5000 6000 7000 8000 SRT = 30 d 圖4-12 反應槽 SRT 為 30 天之 MLSS 變化 44SRT = 30 d SRT = 60 d concen tration (mg/L ) 0 10 20 30 40 50 soluble protein soluble polysaccharide soluble protein soluble polysaccharide 圖4-13 反應槽操作在 SRT 為 30 與 60 天之溶解性 EPS 圖 4-14 表示反應槽隨時間變化 DGGE 圖譜,分別於反應槽操作 時間為 43、49、58 與 90 天所採集的樣本,,a、b 分別表示同一天 的不同取樣位置,a 表示由反應槽上方取樣,b 表示由反應槽下方取 樣;編號A ~ K 表示亮帶出現的位置及在附錄 C 中可得知序列比對列 表。 由 圖 4-14 配 合 附 錄 C 可 知 , 反 應 槽 之 菌 種 大 部 分 是
Betaproteobacteria 、 Alphaproteobacteria 、 Bacteroidetes 、
Deltaproteobacteria與environmental samples。Deltaproteobacteria亦是
46 proteobacteria中的一支,此類細菌許多是屬於硫還原菌或硫酸還原 菌,這類菌通常為極度厭氧,可利用硫或硫酸鹽在無氧呼吸時,作為 呼吸鏈中的電子接受者。 隨著時間的變化,發現菌種 A 與 E (Zoogloea oryzae)在反應槽操 作後期出現且極佔優勢,菌種B (Aquabacterium sp.)與 C (Bdellovibrio sp.)隨著反應槽的操作一直存在,且菌種 C 漸趨優勢。菌種 D
(Flexibacteraceae bacterium)、G (Candidatus nitrotoga)與 I (Uncultured
alpha proteobacterium)在反應槽初期存在,特別是菌種 I 一開始極佔優 勢。 此外,由圖4-14 亦可以得知,在總體積為 30 L 的反應槽中,不 論是從反應槽上方取樣抑或從反應槽下方取樣,所得到之菌相結果是 相同的,即反應槽的菌相分佈是均勻的,並不會因取樣位置而有所改 變。 將定序後之序列與NCBI 序列資料庫進行比對,建構微生物相之 親緣演化樹,推測微生物相之演化關係,並利用軟體繪製親緣樹,如 圖4-15 表示反應槽操作在 SRT 為 30 天之菌群結構親緣分析。
圖4-14 SRT 為 30 天之反應槽隨時間變化之 DGGE 圖譜
註:a 表示由反應槽上方取樣
b 表示由反應槽下方取樣
48
表 4-6 為反應槽操作在 SRT 為 30 天之隨時間變化 Cs 値,藉由
Cs 値亦可驗證無論從反應槽的上方或是下方取樣做菌相分析,並不
會得到不同的族群。
表4-6 SRT 為 30 天之反應槽隨時間變化之 Cs 値
43 days a 43 days b 49 days a 49 days b 58 days a 58 days b
43 days a 43 days b 1 49 days a 1 1 49 days b 1 1 1 58 days a 0.73 0.73 0.73 0.73 58 days b 0.73 0.73 0.73 0.73 1 90 days 0.67 0.67 0.67 0.67 0.95 0.95
圖4-15 SRT 為 30 天之菌群結構親緣分析
4.3.2 SRT 為 60 天之水質分析與菌相變化
當反應槽操作在SRT 為 60 天下時,發現持續監測長達 5 個月後 仍在馴養期,MLSS 並未達穩定,MLSS 為 1090 ~ 12260 mg/L,如圖 4-16 所示。本實驗之進流水為人工合成,TOC 約 170 mg/L。出流水 TOC 為 1.9 ~ 4.05 mg/L,平均約 2.45 mg/L;TOC 去除率平均約 98.6 %。 Time (day) 0 20 40 60 80 100 120 140 160 ML SS (mg/ L) 0 2000 4000 6000 8000 10000 12000 14000 SRT = 60 d 圖4-16 反應槽 SRT 為 60 天之 MLSS 變化 5051 圖 4-18 表示反應槽隨時間變化 DGGE 圖譜,分別於反應槽操作 時間為 3、16、37、51、65、86、146 與 162 天所採集的樣本,編號 A ~ P 表示亮帶出現的位置及在附錄 B 中可得知序列比對列表。 反應槽操作在 SRT 為 60 天之菌種鑑定結果與 4-2 節中反應槽前 端未架設選種槽 (SRT 為 10 天)之菌種鑑定結果相同,大都為
Betaproteobacteria、Bacteroidetes 與 environmental samples。
由圖4-18 配合附錄B得知菌種L (Zoogloea sp.)不但隨著時間的變
化濃度漸漸增加,而且亦是反應槽中最佔優勢的菌種,另外菌種B
(Uncultured bacterium)與O (Zoogloea sp.)雖濃度不高,但卻是隨著時
間的變化一直存在於反應槽中的,而在反應槽操作初期發現優勢菌種
變多有E (Uncultured bacterium)、F (Flavobacterium sp.)、G (Uncultured
bacterium)、H (Flexibacteraceae bacterium)、I (Uncultured bacterium)、
J (Uncultured bacterium)與K (Uncultured Bacteroidetes bacterium)。
由圖 4-16、4-17 與 4-18 可知,反應槽操作始終未達到穩定,無
論是在MLSS、EPS 或菌相分析,致使反應槽薄膜容易產生積垢現象,
3 16 37 51 65 86 146 162
A B C D E F G H I J K L M N O P 圖4-18 SRT 為 60 天之反應槽隨時間變化之 DGGE 圖譜 5253 表 4-8 為反應槽操作在 SRT 為 60 天之隨時間變化 Cs 値,得知 Cs 値介於 0.55 ~ 0.93 之間。Cs 值之結果亦近一步證明反應槽菌相隨 時間之變化。 表4-8 SRT 為 60 天之反應槽隨時間變化之 Cs 値 3 16 37 51 65 86 146 3 16 0.93 37 0.79 0.79 51 0.67 0.64 0.67 65 0.72 0.80 0.72 0.76 86 0.69 0.62 0.62 0.64 0.70 146 0.69 0.55 0.69 0.64 0.69 0.81 162 0.64 0.64 0.71 0.67 0.72 0.77 0.90
54
4.4 薄膜上的菌相變化
圖 4-19 表示薄膜於不同 SRT 下之 DGGE 圖譜,編號 10a ~ 10c 表示反應槽操作在SRT 為 10 天;編號 60a ~ 60c 表示反應槽操作在 SRT 為 60 天,a ~ c 表示不同取樣位置,分別為薄膜上方、中心與下 方;編號A ~ L 表示亮帶出現的位置及在附錄 D 中可得知序列比對列 表。 由圖4-19 配合附錄D可知,大致來說薄膜上之菌種鑑定結果較為 多 樣 , 分 別 有 Alphaproteobacteria 、 Betaproteobacteria 、 Deltaproteobacteria、Bacteroidetes、
Verrucomicrobia與環境樣本。不同 於反應槽中的菌相,薄膜上的菌相多了Verrucomicrobia。 Verrucomicrobia (疣黴菌門)是一門被劃出不久的細菌,主要被發 現 於 水 生 、 土 壤 環 境 及 人 類 糞 便 中 。 Verrucomicrobia 中 之 Prosthecobacter (突柄桿菌屬)具有胞質突出形成的兩個到多個突起, 它們的最近類群可能是衣原體門 (Chlamydiae)。 當反應槽操作在不同 SRT 下,由圖 4-19 可知薄膜上的菌相有蠻大的差異,唯有菌種 A (Uncultured Sphingobacteria bacterium)、B
(F.ferrugineum) 、 J (Uncultured bacterium) 及 K (Uncultured
Flexibacteraceae bacterium)可在兩種 SRT 下存在。
55
槽中發現,雖在研究中不被認為是去除2,4-DNP的優勢菌種,但藉由
菌種存在於反應槽中,可推論其有忍受2,4-DNP的能力(Kimura et al., 2003) 。
在SRT 為 10 天的薄膜上,可發現優勢菌種為 C (Comamonadaceae
bacterium)、D (Bacteriovorax sp.)、F (Prosthecobacter vanneervenii)及
J;而在 SRT 為 60 天的薄膜上,可發現優勢菌種為 E (Flexibacter flexilis)
與I (Uncultured alpha proteobacterium)。Hirashi et al., (1995)曾研究
Comamonadaceae bacterium 於反應槽中有脫硝的能力。 另外,對反應槽沉降性有益的 Zoogloea oryzae (菌種 G)只在 SRT 為60 天的薄膜上發現,且在薄膜菌相中不佔優勢。 將定序後之序列與NCBI 序列資料庫進行比對,建構微生物相之 親緣演化樹,推測微生物相之演化關係,並利用軟體繪製親緣樹,如 圖4-20 為薄膜之菌群結構親緣分析。
圖 4-19 薄膜上菌種鑑定隨時間變化之 DGGE 圖譜 註:a 表示由薄膜上方取樣 b 表示由薄膜中央取樣 c 表示由薄膜下方取樣 表4-9 為薄膜表面族群相似度分析之 Cs 値,得知 Cs 値介於 0.32 56
57 ~ 1 之間。但若以 SRT 作區分可發現,在同一個 SRT 下其 Cs 值介於 0.96 ~ 1,即在相同 SRT 下薄膜表面之族群相似度極高。而當在不同 SRT 時,彼此間的族群相似度就很低。 表4-9 薄膜表面族群相似度分析之 Cs 値 10a 10b 10c 60a 60b 10a 10b 0.96 10c 0.96 1 60a 0.33 0.32 0.32 60b 0.32 0.38 0.38 0.96 60c 0.32 0.38 0.38 0.96 1
59
4.5 綜合討論
綜合以上結果得知,在反應槽操作條間中,若有添加選種槽單元 於反應槽前端,將有助於使微生物相簡單化。即使是利用簡單的人工 合成廢水,同樣可於反應槽內馴養出複雜的微生物相 (Lapara et al., 2006) 。 此外,在反應槽操作初期,會有微生物相的變動,當反應槽操作達到 穩定後,菌相變化將減低;當反應槽操作條件發生變化時,菌相會因 而有些微變動,待反應槽操作條件回復後,菌相也會一並回復 (Klatt andLapara, 2003; Lapara et al., 2006) 。
當反應槽操作在不同條件時,不論是改變SRT或者是於反應槽前
端 添 加 選 種 槽 單 元 , 反 應 槽 內 的 菌 相 都 包 括Betaproteobacteria與
Bacteroidetes,此結果與學者發現在其四種不同反應槽中皆出現CFB
之結果相符合 (Choi et al., 2006) 。學者指出紙廠廢水中含有組成複雜的有
機碳,提供了CFB良好的生長條件 (Ross and Hollis, 1976; Oppong et al., 2003) 。此外
CFB亦被認為是親緣樹族群中的前驅族群 (Choi et al., 2006) 。 此外反應槽中與薄膜表面之微生物相差別極大,薄膜表面菌相以 Bacteroidetes 為主;反應槽中菌相則以 Betaproteobacteria 與無法培養 的環境樣本為主;不同的是當反應槽操作在SRT 為 30 天時,其次要 菌群為Alphaproteobacteria 而非無法培養的環境樣本。薄膜上的微生 物相與反應槽內的微生物相,有極大的差異。推測薄膜積垢的形成決 定於薄膜上的菌相而非反應槽內的菌相。
60
第五章 結論與建議
5.1 結論
本研究發現反應槽中之微生物相分布是均勻的,並不會因取樣點 位置的不同而影響研究結果,而於MBR 前端增加選種槽單元則有助 於使反應槽微生物相單純化,其中以Betaproteobacteria 中之 Zoogloea 最佔優勢。此外,操作在不同SRT (10 與 60 天),反應槽內之微生物 相並不會有太大的變更,有明顯差異的為薄膜上的微生物相,因此推 測薄膜積垢的形成決定於薄膜上的菌相而非反應槽內的菌相;且在 SRT 為 10 天之薄膜上的微生物相中發現 Verrucomicrobia 與薄膜積垢 時反應槽中發現的 Chlamydiae 在親緣關係上相近,因此可假設 Verrucomicrobia 將是導致薄膜積垢的微生物族群之一。61
5.2 建議
本研究利用分子生物技術對MBR 進行菌相鑑定與探討,在偵測 上雖可比傳統的微生物培養法來的精確,卻也有其限制,當菌種濃度 過低便無法在後續實驗中將其純化做進一步定序,因此建議後續相關 研究時,可採用多種不同技術同步進行比較與查核。且本研究所使用 之引子對長度約200 bp,在菌種比對上代表性稍嫌不足,建議往後之 相關研究若仍希望藉由 PCR-DGGE 進行菌相鑑定,可設計出長度約 500 bp 的引子對,將會有較高的代表性助於研究。62
參考文獻
Abughararah, Z.H. and Randall, C.W. (1991) The Effect of Organic Compounds on Biological Phosphorus Removal. Water Science and Technology 23: 585-594.
Al-Malack, M.H. and Anderson, G.K. (1996) Coagulation-Crossflow Microfiltration of Domestic Wastewater. Journal of Membrane Science
121: 59-70.
Amann, R.I., Krumholz, L., and Stahl, D.A. (1990) Fluorescent-Oligonucleotide Probing of Whole Cells for Determinative, Phylogenetic, and Environmental Studies in Microbiology. Journal of Bacteriology 172: 762-770.
Amann, R.I., Ludwig, W., and Schleifer, K.H. (1995) Phylogenetic Identification and in Situ Detection of Individual Microbial Cells without Cultivation. Microbiological Reviews: 143-169.
Asatekin, A., Menniti, A., Kang, S., Elimelech, M., Morgenroth, E., and Mayes, A.M. (2006) Antifouling Nanofiltration Membranes for Membrane Bioreactors from Self-Assembling Graft Copolymers. Journal of Membrane Science 285: 81-89.
Bond, P.L., Hugenholtz, P., Keller, J., and Blackall, L.L. (1995) Bacterial Community Structures of Phophate-Removing and Non-Phosphate-Removing Activated Sludge from Sequencing Batch Reactors. Applied and Environmental Microbiology 61: 1910-1916.
Bura, R., Cheung, M., Liao, B., Finlayson, J., Lee, B.C., Droppo, I.G., Leppard, G.G., and Liss, S.N. (1998) Composition of Extracellular Polymeric Substances in the Activated Sludge Floc Matrix. Water Science and Technology 325-333.
Busse, H.J., E. M. B. Denner, and Lubitz, W. (1996) Classfication and Identification of Bacteria: Current Approaches to an Old Problem. Overview of Methods Used in Bacterial Systematics. Journal of Biotechnology 3-38.
63
Chang, I.-S., Le Clech, P., Jefferson, B., and Judd, S. (2002) Membrane Fouling in Mbrs for Wastewater Treatment. Journal of Environmental Engineering 1018–1029.
Chang, I.S. and Lee, C.H. (1998) Membrane Filtration Characteristics in Membrane-Coupled Activated Sludge System -- the Effect of Physiological States of Activated Sludge on Membrane Fouling. Desalination 120: 221-233.
Cho, B.D. and Fane, A.G. (2002) Fouling Transients in Nominally Sub-Critical Flux Operation of a Membrane Bioreactor. Journal of Membrane Science 209: 391-403.
Choi, H., Zhang, K., Dionysiou, D.D., Oerther, D.B., and Sorial, G.A. (2006) Effect of Activated Sludge Properties and Membrane Operation Conditions on Fouling Characteristics in Membrane Bioreactors. Chemosphere 63: 1699-1708.
Choi, J.H., Lee, S.H., Fukushi, K., and Yamamoto, K. (2007) Comparison of Sludge Characteristics and PCR-DGGE Based Microbial Diversity of Nanofiltration and Microfiltration Membrane Bioreactors. Chemosphere
67: 1543-1550.
Choo, K.H., Kang, I.J., Yoon, S.H., Park, H., Kim, J.H., Adlya, S., and Lee, C.H. (2000) Approaches to Membrane Fouling Control in Anaerobic Membrane Bioreactor. Water Science and Technology 41: 363.
Chua, H.C., Arnot, T.C., and Howell, J.A. (2002) Controlling Fouling in Membrane Bioreactors Operated with a Variable Throughput. Desalination 149: 225-229.
Dice, L.R. (1945) Measures of the Amount of Ecologic Association between Species. Ecology 26: 297-302.
Doyle, R.J. (1991) Strategies in Experimental Microbial Research, In: N. Mozes, P.S. Handley, H.J. Busscher, P.G. Rouxhet (Eds.). Microbial Cell Surface Analysis, VCH Publishers, New York: pp.292-316.
Fang, H.H.P. and Liu, H. (2002) Extraction of Extracellular Polymeric Substance (EPS) of Sludge. Journal of Biotechnology.
64
Ferris, M.J., and Ward, D.M. (1997) Seasonal Distributions of Dominant 16s Rrna-Defined Populations in a Hot Spring Microbial Mat Examined by Denaturing Gradient Gel Electrophoresis. Applied and Environment Microbiology 63: 1375-1381.
Gerhardt, P. and Murray, R.G.E. (1981) Manual of Methods for General Bacteriology. American Society for Microbiology, Washington, D.C. Graun, G.F., Bull, R.J., Clark, R.M., Doull, J., Grabow, W., Marsh, G.M., Okum, D.A., Regli, S., D., S.M., and Symons, J.M. (1994) Balancing Chimical and Microbial Risk of Drinking Water Disinfection, Part1. Benefits and Potential Risk. Journal of Water SRT-Aqua 43: 192-199.
Hirashi, A., Shin, Y.K., and Sugiyama, J. (1995) Brachymonas Denitrificans Gen. Nov., Sp. Nov., an Aerobic Chemoorganotrophic Bacterium Which Contains Rhodoquinones, and Evolutionary Relationships of Rhodoquinone Producers to Bacterial Species with Various Quinine Classes. Journal of General and Applied Microbiology
41: 99-117.
Hong, S.P., Bae, T.H., Tak, T.M., Hong, S., and Randall, A. (2002) Fouling Control in Activated Sludge Submerged Hollow Fiber Membrane Bioreactors. Desalination 143: 219-228.
Howell, J.A., Chua, H.C., and Arnot, T.C. (2004) In Situ Manipulation of Critical Flux in a Submerged Membrane Bioreactor Using Variable Aeration Rates, and Effects of Membrane History. Journal of Membrane Science 242: 13-19.
Hsieh, K.M., Murgel, G.A., Lion, L.W., and Shuler, M.L. (1994) Interactions of Microbial Biofilms with Toxic Trace Metals: 1. Observation and Modeling of Cell Growth, Attachment, and Production of Extracellular Polymer. Biotechnology and Bioengineering 44: 219-231.
Hvid, K.B., Nielsen, P.S., and Stengaard, F.F. (1990) Preparation and Characterization of a New Ultrafiltration Membrane. Journal of Membrane Science 53: 189-202.
Ingraham, J.L., Maaloe, O., and Neidhardt, F.C. (1983) Growth of the Bacteria Cell. Sunderland, Massachusetts.