行政院國家科學委員會專題研究計畫 成果報告
識別子設計最佳化及基因晶片應用之研究
計畫類別: 個別型計畫 計畫編號: NSC91-2622-E-002-035-CC3 執行期間: 91 年 06 月 01 日至 92 年 05 月 31 日 執行單位: 國立臺灣大學資訊工程學系暨研究所 計畫主持人: 高成炎 計畫參與人員: 陳權忠, 黃友正, 黃崇隆, 黃玟憲, 蕭子昌, 陳士傑 報告類型: 完整報告 處理方式: 本計畫為提升產業技術及人才培育研究計畫,不提供公開查詢中
華
民
國 92 年 8 月 15 日
國科會補助提升產業技術及人才培育研究計畫成果精
簡報告
學門領域:資訊
計畫名稱:識別子設計最佳化及基因晶片應用之研究
計畫編號:NSC91-2622-E-002-035-CC3
執行期間:20020601~20030531
執行單位:國立臺灣大學資訊工程學系暨研究所
主 持 人:高成炎
參與學生:
姓 名
年 級
(大學部、碩士 班、博士班)已發表論文或已申請之專利
(含大學部專題研究論文、碩博士論文)工作內容
陳權忠
碩二
黃友正
碩一
Efficient Primer design
algorithm with partial order
graphs, ISMB2003.
研發及設計
黃崇隆
碩二
研發及設計
黃玟憲
碩二
協助設計開發
蕭子昌
碩二
協助設計開發
陳士傑
碩二
研發及設計
合作企業簡介
合作企業名稱:百恩諾生物科技股份有限公司
計畫聯絡人:陳麗貞
資本額:27,580,000
產品簡介:微陣列生物晶片生產
網址:http://www.asiabioinnovations.com.tw 電話:(02)33651872
ext:114
研究摘要:
基因晶片(DNA Chip)應用的起源是因為成千上萬的基因所形成的產物為構 成生命最基本的要素,故發展出以鹼基配對(base pairing)與核甘酸雜交(nucleotide hybridization)原理為基礎的技術。其原理是由 1962 年諾貝爾獎得主華生(J. Watson)和克里克(F.H. Crick)所建構 DNA 分子模型,遵循 A-T 與 C-G 互補配對 規則,A 鹼基與配對股(complimentary strain)的 T 鹼基結合,而 C 鹼基會與配對 股(complimentary strain)的 G 鹼基結合。基因晶片(DNA Chip)利用此項原理, 將 設計好的探針(probe)以不同的方式排列固定在晶片上, 使得樣本能夠與其反應, 並產生具特異性的雜交結果,其中識別子(primer)的設計亦是此項技術實驗所需 的核心關鍵。隨著後基因體時代的來臨,各物種的基因陸續被一一定序出來,尤 其是人類基因的定序,被視為是未來生物醫療方面的重要里程碑,定序完成只提 供一窺遺傳密碼全貌的機會,欲瞭解與應用基因資訊,最重要的還是了解其功 能,人類的功能基因 (functional genes) 控制人類疾病、遺傳、行為等生理現象, 探索基因的功能及其間之交互作用,過去的傳統生物學卻只能研究觀察數量很少 的基因表現量,尋找基因、探索其功能更有如大海撈針般困難。而識別子(primer) 的設計也在生物實驗中扮演著舉足輕重的角色,將微量的基因(DNA)放大來進行 實驗更是目前生物基因實驗上一個必要的過程。而在大量基因(DNA)的情形下, 如何找出具有專一性且最符合經濟效益的識別子(primer)來放大標的物(target DNA),是目前最廣為討論的課題。在這問題下,需求一個最基本的技巧,就是 基因序列的比對。因為設計識別子(primer)的方法絕大多數都需要面臨這方面的 問題,如何在既有的設計條件下,最佳化設計識別子(primer)的時間,更是一個 需要關心的問題。此計畫中,會提到一個好的資料結構來表達基因序列,稱之為 局部順序圖形(partial order graph),不僅可以保留原有的基因序列資訊,更可以加 速設計識別子(primer)的過程。
人才培育成果說明:
在此計畫中,以研究基因晶片(DNA chip)與識別子(primer) 設計為主軸,使 多位研究相關問題的研究者得以順利解決一些重要的問題。
以研究基因晶片(DNA Chip)為例,幾位研究者鑽研二維基因(cDNA)微陣列分 群方法,並從已發表的期刊上獲得許可的相關資訊,並繼續朝解決此問題邁進。 識別子(primer) 設計更是讓研究者整合了相當重要的演算法(set covering)及 遺傳式演算法(Genetic Algorithm)來設計可以用聚合脢連鎖反應(PCR) 來放大最 多標的基因(target gene)之最小的識別子(primer)集合。這是一個生物實驗上的重 要議題,因為在有限的經費下,可以放大基因(Gene),得到最佳的結果。 於基礎研究上,更發現了可以由基因序列的角度,將其以新的表示法來進行 序列比對,在序列極為相似的情形下,可以大幅降低設計識別子(primer) 的時 間,如此一來,可加入更多的篩選條件,獲得更具專一性且更佳的識別子(primer)。
技術研發成果說明:
發展了一套整合識別子(primer)設計的演算法。由於同時要處理多條的基因 (DNA)序列,因此一開始想要成為識別子(primer)的引子往往非常多,可以多至 數百萬條,但真正成為識別子(primer)的引子可能只剩數十條,因此加速最基礎 的基因(Gene)序列比對,使設計識別子(primer)的方法得以更快速更全面的考量所 有情況,是這演算法的重點之一,包括 : 檢查重複的鹼基序列、二級結構的識 別子(primer)、熔化溫度(melting temperature)、G/C 鹼基佔序列的百分比… 等,這 些都是設計識別子(primer)的重要參考依據。 除了識別子(primer)基本設計外,隨著基因體資料庫不斷膨脹,有越來越多需 要處理的基因(DNA)序列,如果想針對多重基因(DNA)序列做聚合脢連鎖反應 (PCR)時,更希望用最少的識別子(primer)增幅最多的基因(DNA)序列,因此亦利 用了集合理論上的想法與遺傳式演算法(Genetic Algorithm),搭配電腦來設計識 別子(primer),如此一來可用最少合成識別子(primer)的經費,來獲得最大的實驗 成果。技術特點說明:
由於同時要處理多條的基因(DNA)序列,因此加速最基礎的基因(Gene)序列 比對,使設計識別子(primer)的方法得以更快速更全面的考量所有情況,是這演 算法的特點。
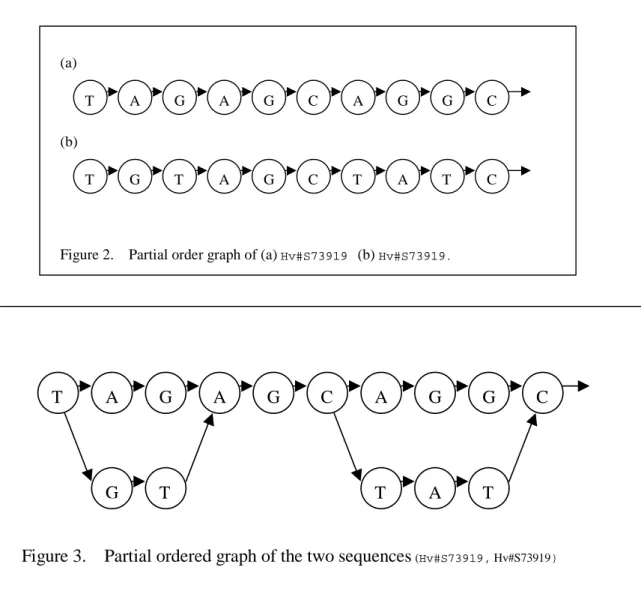
將基因(DNA)序列先行轉成局部順序圖形(partial order graph) (Figure 1)。
再將每個局部順序圖形(partial order graph) (Figure 2)合成一緊密的局部順序 圖形(compact partial order graph) (Figure 3)。之後開始利用遺傳式演算法(Genetic Algorithm)設計識別子(primer)。
Figure 1
T A G A G C A G G C
Figure 2. Partial order graph of (a) Hv#S73919 (b) Hv#S73919.
T A G A G C A G G C (a)
T G T A G C T A T C (b)
Figure 3. Partial ordered graph of the two sequences (Hv#S73919, Hv#S73919)
T A G A G C A G G C
根據局部順序圖形(partial order graph)的資料結構,可以更有效率的設計識別 子(primer) (Figure 3)。
將標的序列(target sequence)依照每個局部順序圖形(partial order graph)合成一 緊密的局部順序圖形(compact partial order graph)後開始利用遺傳式演算法 (Genetic Algorithm)設計識別子(primer) (Figure 4)。
Figure 3. Polymerase Chain Reaction
Forward primer
Amplified
Reverse primer
region
3
3
5
5
Figure 7. Partial ordered graph of the two sequences (Target 1, Target 2)
A T C A T C G A T t1 A t2 p1 T C A C A G T A Target T = (t1, . . . , tn), Primer p = (p1, . . . , pm) t1 = ATCATCGAT t2 = ATAATCGAT . = .. . = .. . = ..
We construct the partial order graph of target sequences. Primer p1 = TTCACAGTA