台灣產土肉桂、山肉桂、及胡氏肉桂(樟科)之遺傳變異與分類地位
何坤益
*國立嘉義大學森林暨自然資源學系 (收稿日期:2006.8.4,接受日期:2006.12.6)
摘 要
山肉桂(Cinnamomum insulari-montanum Hayata)、胡氏肉桂(C. macrostemon Hayata)與土肉 桂(C. osmophloeum Kanehira)等為台灣樟科特有種植物,此三種肉桂類植物在植物分類上一直存 有爭議及困難,本研究應用簡單序列重複(Inter-Simple Sequence Repeat,簡稱 ISSR)分子指紋技 術探討其遺傳變異與分類地位關係。研究分析了在臺灣自然分佈之 22 地區 144 個樣本,試驗共使 用 12 個 ISSR 引子進行 PCR 擴增,產生了 52 個多型性條帶,遺傳變異分析結果,分子變方分析 (AMOVA)顯示種間之變方成分是 27.24%%(p<0.01),而種內之變方成分是 72.67%(p<0.001), 顯示變異來自各種內。根據 POPGENE 分析各種內之遺傳分化程度,山肉桂與胡氏肉桂可能受到地 理隔離影響具有明顯的地理分化,Nm 值分別為 0.5024 及 0.6533;而土肉桂基因流值為 1.2626,顯 示較低的遺傳分化現象。探討分類地位關係所進行之歸群分析樹狀圖顯示土肉桂、山肉桂與胡氏肉 桂族群各自形成三群團,這樣的結果亦受到主座標分析(Principal Coordinate Analysis, PCOA)之 支持。 關鍵詞:土肉桂、山肉桂、胡氏肉桂、簡單序列重複、遺傳變異、分類地位、分子變方分析
緒 言
肉桂類(肉桂節)植物屬樟科(Lauraceae) 樟屬(Cinnamomum),樟屬植物全世界約有 250 種,主要分佈在亞洲熱帶、亞熱帶、太平洋群島 及澳洲等,台灣有 12 種,其中肉桂類有 7 種, 分 別 為 牡 丹 葉 桂 皮 ( C. austro-sinense H.T.Chang )、 山 肉 桂 ( C. insulari-montanum Hayata )、 蘭 嶼 肉 桂 ( C. kotoense Kanehira & Sasaki)、胡氏肉桂(C. macrostemon Hayata)、土 肉 桂 ( C. osmophloeum Kanehira )、 香 桂 ( C.subavenium Miq)及天竺桂(C. tenuifolium form. nervosum Sieb.)。山肉桂、蘭嶼肉桂、胡氏肉桂 及土肉桂為台灣特有種;另外亦有引進之肉桂栽 培如肉桂(C. cassia Presl)及錫蘭肉桂(C. verum Bl.,)(Liu et al., 1994,Yang et al., 1999,Liao,
1995,Liao, 1996)。肉桂具有醫藥及食品添加香 料等用途,另外亦有抗菌的作用,甚具經濟價 值 。 過 往 研 究 發 現 台 灣 之 土 肉 桂 葉 部 肉 桂 醛 ( cinnamaldehyde) 含 量 甚 高 , 極 具 發 展 淺 力 (Wang, 1987),但精油的含量與產地有關,似乎 顯示土肉桂族群間已有分化產生的現象(Yin, 1991)。 土肉桂俗稱假肉桂,分布由低海拔至 1500 m 闊葉樹林,全株富含單寧成分,尤其是樹皮有辛 辣的肉桂香味,可代替肉桂供藥用及香料(胡大 維,1992)。山肉桂俗名為台灣肉桂或桂枝,全 島雖有分佈,但族群集中於中部的奧萬大及霧 社,自其樹皮提取之精油,亦可供藥用或香料(Liu et al., 1994)。胡氏肉桂則為常綠喬木,分佈於 中、低海拔森林,族群零散分布於臺北五指山、 翡翠水庫等地區,本種已列為IUCN 物種保育等 級評估為易受害(vulnerable)之稀有植物(Lu and Chiu, 1994)。雖在植株形態上此三種肉桂在植物 分類仍有區別性狀,如山肉桂之芽無鱗片,葉為 廣橢圓形,花梗及花被片外有絹毛。土肉桂之芽 具鱗片且光滑,葉為狹長橢圓形,花梗與花被片 光滑。胡氏肉桂之芽具覆瓦狀排列 9 鱗片被毛, 葉為橢圓至披針形,花梗與花被片白色絹毛;惟 天然林分中植株形態常因環境因素,每有中間形 態與變異者,故其之分類一直有困難。 三種肉桂在植物分類訂正過程中,僅土肉桂 自 Hayata(1913)命名,經 Sasaki(1928)、Kanehira (1936)、Li(1963)等,一直到 Liao(1988) 訂定為獨立種,其分類地位較為穩固。然而山肉 桂及胡氏肉之命名發表後,卻一再經分類學家之
Kuen-Yih Ho 94 訂正與不同分類處理,此亦可見其族群之變異由 來已久(潘富俊,1992)。山肉桂最早由 Hayata (1913)命名,Kanehira(1936)將之處理為土 肉桂之異名;Li(1963)將之併於日本香桂(C. jponicum),直到 Liu and Liao(1971)才又將土 肉桂自日本香桂分離出來,山肉桂目前國內分類 處理雖趨於一致,如 Liu et al.,(1988)及 Liao (1988)視為獨立種,惟 Li(1984)所編著之中 國植物誌並未列本種仍將處理為日本香桂。胡氏 肉桂亦為 Hayata(1913)所命名,發表後一直受 到質疑,Kanehira(1936)認為應與山肉桂同種。
Li(1963)、Chang(1976)與 Li(1984)之中國
植物誌未列胡氏肉桂。Liu and Liao(1971)將胡 氏肉桂列為日本肉桂之異名。最近 Lu and Yang (1986)根據芽鱗之排列,及其上之褐色毛等特 徵肯定此種之分類地位。但隨後 Liao(1988)再 將胡氏肉桂處理成山肉桂之異名。因此,除土肉 桂 之 命 名 被 訂 定 為 獨 立 種 分 類 地 位 較 為 穩 固 外,胡氏肉桂與山肉桂,則從其命名與後續訂正 過程即可見其爭議。 近年來,分子標記廣泛應用在生物種內、種 間、族群內、族群間的研究。Ziekiewicz et al. ( 1994 ) 發 展 的 簡 單 序 列 重 複 ( Inter-Simple Sequence Repeat,簡稱 ISSR)分子指紋技術,不 須事先知道材料 DNA 的序列,且由相同單元組 成的 ISSR 可以出現在親緣很遠的分類群間,使 得這項技術可以同時放大許多個基因座,且同一 個引子可以應用到不同的分類群研究上。加上 ISSR 引子能提高專一性,分析結果更可靠,其操 作簡單、不需特殊設備與大量的 DNA,符合 Mendelian 遺傳定律,因此可得到較高可信度的 遺傳多型性資料。目前 ISSR 被應用到遺傳變異 的偵測(Ge et al., 2003,Li et al., 2002,Li and Ge, 2001,Qian et al., 2001)、品種品系之區分(Prevost
and Wilkinson, 1999)、自然族群雜交現象之研究
(Hollingsworth et al., 1998,Wolfe et al., 1998) 等。 由於台灣各地海拔、氣候變化與生育地環境 差異,是否因生育地的不同而族群間產生遺傳變 異與形態之分化,本研究即應用 ISSR 分子標記, 探討三種肉桂之遺傳變異與分類地位。
材料與方法
一、材料 本研究針對山肉桂、胡氏肉桂及土肉桂等三 種肉桂類植物之天然族群進行取樣,採得產於台 灣之 22 個自然分佈地區(Table 1 及 Fig.1),每 區取 3-10 個樣株,並避免採集自同源母樹,共採 得 144 單株,每株選取無病蟲害 5-10 葉片,以矽 膠乾燥劑脫水乾燥。 二、方法 (一)DNA 之萃取及定量:採用 Doyle and Doyle(1987)CTAB 萃取法, 分離 genomic DNA,利用分光光度計進行 DNA 濃度測定。 (二)ISSR 的擴大反應: 參考 Ziekiewicz et al.(1994)的方法加以修 改,方法如下: 1.反應溶液總體積為 25μL,其反應試劑濃度為: 10mM Tris-HCl, 50mM KCl, 1.0 mM MgCl, 0.1% gelatin(w/v), 1% Triton X-100(w/v), 10 ng template DNA, 100μM dNTPs, 0.2 μM primer, and 0.5 units Taq polymerase(HT Biotechnology, England)。
2. 溫 度 循 環 機 ( thermocycler ; Perkin Elmer Geneamp PCR System 9700)進行 PCR 程式:94 ℃ 6 min (initial denaturing step),39 次循環: 50℃ 50 sec, 72℃ 2 min,及 72℃7 min(final elongation step),最後降溫至 4 ℃備用。 3.電泳:以 1.5% agarose(GIBCO BRL),將擴增 的 DNA 產物進行電泳。 4.染色及照相:使用 ethidium bromide 染色,並以 Polaroid 667 拍照記錄。 三、統計分析 ( 一 ) 記 錄 多 型 性 條 帶 與 計 算 相 似 度 (similarity): 讀取清晰、亮度大且具多型性之條帶,以代 碼 1(出現)和 0(不出現)記錄條帶出現狀況。 利用 NTSYS-pc ver 2.0 套裝軟體(Rohlf, 1993), 以 Simple Matching 公式計算兩兩樣本間之相似 度矩陣(SSM),另以 Dice(Dice, 1945)公式計 算兩兩樣本間之相似度(SAB)矩陣: SSM=m/n;m:兩兩樣本一致出現及一致不出現 條帶數,n:多型性條帶總數 SAB=2NAB/(2NAB+NA+NB) NA:樣本 A 出現 但樣本 B 不出現的條帶數;NB:樣本 B 出現但 樣本 A 不出現的條帶數;NAB:樣本 A 與樣本 B 皆出現的條帶數。
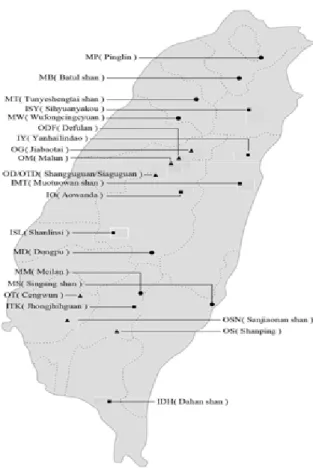
表一、試驗編號、採樣地點、海拔高度與樣本數目。
Table 1. Sources Code, Taxon, Collected site, Elevation, Sample size.
Prov.code Taxon Collected site Elevation(m) Sample size 1.OS C. osmophloeum Shanping(高雄扇平) 1400 8 2.OM C. osmophloeum Malun(台中馬崙山) 1600 5 3.OG C. osmophloeum Jiabaotai(台中佳保台) 800 6 4.OD C. osmophloeum Shangguguan(台中上谷關) 500 8 5.OTD C. osmophloeum Siaguguan(台中下谷關) 500 2 6.OT C. osmophloeum Cengwun(台南曾文) 300 10 7.ODF C. osmophloeum Defulan(台中德芙蘭) 400 12 8.OSN C. osmophloeum Sanjiaonan shan(台南三腳南山) 800 5 9.MP C. macrostemon Pinglin(台北坪林) 500 7 10.MB C. macrostemon Batul shan(台北拔刀爾山) 850 9 11.MS C. macrostemon Singang shan(台東新港山) 650 6 12.MD C. macrostemon Dongpu(台中東埔) 800 10 13.MM C. macrostemon Meilan(高雄梅蘭) 1300 8 14.MT C. macrostemon Tunyeshengtai shan(新竹屯野生台山) 1900 9 15.MW C. macrostemon Wufongcingcyuan(新竹五峰清泉) 1000 3 16.IMT C. insulari-montanum Motuowan shan(花蓮莫托灣山) 1600 4 17.IDH C. insulari-montanum Dahan shan(高雄大漢山) 1400 8 18.IY C. insulari-montanum Yanhailindao(花蓮研海林道) 1100 7 19.ITK C. insulari-montanum Jhongjhihguan(高雄中之關) 1700 3 20.IO C. insulari-montanum Aowanda(南投奧萬大) 1200 8 21.ISL C. insulari-montanum Shanlinsi(南投杉林溪) 1500 3 22.ISY C. insulari-montanum Sihyuanyakou(宜蘭思源啞口) 2000 3
(二)AMOVA(Analysis of Molecular Variance) 與 POPGENE 分析: 將 SSM 相似度矩陣轉換成 Excoffier et al. (1992)發表的 D 距離矩陣。 D=n(1-SSM) ;n=多型性條帶總數 求出距離矩陣後,以 AMOVA vl.55 程式(Excoffier et al., 1992)進行分析,計算族群間、族群內個 體 間 的 變 方 成 分 值 及 所 佔 總 變 方 成 分 之 百 分 比,並以 9999 次隨機重排測驗各變方成分的顯 著性。並計算出族群間的遺傳距離(Φst)矩陣。
以 POPGENE v.1.31(Yeh et al., 1997)套裝 軟體計算族群間遺傳資料,假設各族群達哈溫平 衡(Hardy-Weinberg equilibrium),則可計算 Nei's 遺傳歧異度指數(H)(Nei, 1973);假設各族群 之近親交配指數(Fis)為 0,則可計算族群間遺 傳分化係數 Gst [coefficient of gene differentiation (Nei, 1973)],再利用 Gst 估算基因流傳值 Nm
圖一、三種肉桂之採集地理位置圖。
Figure 1. Geographic locations of the studied of three
Kuen-Yih Ho
96
(Nm=0.5×(l-Gst)/Gst)(Slakin and Barton, 1989)。
(三)歸群分析(Clustering analysis)與主座標 分析(Principal Coordinate Analysis,PCOA):
將 NTSYS-pc ver 2.0 所計算出之各樣本間 Dice 相似度矩陣及 AMOVA 所算出之地區間距離 ( Φ st) 矩 陣 , 以 NTSYS-pc v.2.0 的 SAHN ( Sequential, Agglomerative, Hierarchial, and Nested clustering method ) 程 式 、 UPGMA (Unweighted Pair-Group Method using Arithmetic averages)方法進行各地區的歸群分析建立樹狀 圖(dendrogram),及進行主座標分析,以三維所 佔變異最大的主要軸繪成立體圖及平面圖,求出 各地區在三維空間上族群的分佈關係。
結 果
一、引子與條帶記錄 經試 125 個隨機序列的引子(University of British Columbia, Vancouver, British Columbia,Canada),選取擴大效果佳,條帶亮度強,產生 的條帶具有多型性及再現性引子,共選用 12 引 子,得到 52 個多型性條帶(Table 2)。 二、族群之遺傳多樣性 (一)分子變方分析(Analysis of Molecular Variance,簡稱 AMOVA): 經由分子變方分析(AMOVA) 9999 次隨機 重排後的結果,顯示三種肉桂植物間之變方成分 是 27.24%%(p<0.01),在種內之變方成分是 72.67%(p<0.001),顯示變異來自各種內(Table 3)。 (二)遺傳分化差異(POPGENE 分析): POPGENE 計算族群間遺傳分化,假設各族 群之近親交配指數(Fis)為 0,所計算 Nei's 遺 傳分化係數結果(Table 4),山肉桂其遺傳分化 係數值(Gst)為 0.2837,胡氏肉桂為 0.4219,土 肉 桂 為 0.4988 。 再 由 分 化 係 數 換 算 得 基 因 流 (Nm),山肉桂基因流值為 0.5024、胡氏肉桂為 0.6533、土肉桂為 1.2626。 三、族群之遺傳變異與分類地位關係 進行歸群分析(Clustering analysis)經計算兩 兩族群之遺傳距離(Table 5),進行族群歸群分 析,經比對遺傳距離矩陣與樹狀圖關係矩陣,得 到協表相關係數(cophenetic correlation 表二、ISSR 引子與多型性條帶數條帶記錄。
Table 2. ISSR primers with number of polymorphic bands(pb).
UBC Primer No. Sequence (5’~ 3’) Number of polymorphic fragments Number of monomorphic fragments Percentage of polymorphic fragments (%) UBC815 CTC TCT CTC TCT CTC TG 7 3 70 UBC823 TCT CTC TCT CTC TCT CC 4 4 50 UBC825 ACA CAC ACA CAC ACA CT 6 1 85.7 UBC827 ACA CAC ACA CAC ACA CG 5 2 71.4 UBC834 AGA GAG AGA GAG AGA GYT 4 1 80 UBC848 CAC ACA CAC ACA CAC ARG 3 2 60 UBC855 ACA CAC ACA CAC ACA CYT 6 1 85.7 UBC857 ACA CAC ACA CAC ACA CYG 6 4 60 UBC864 ATG ATG ATG ATG ATG ATG 4 3 57.1 UBC866 CTC CTC CTC CTC CTC CTC 4 1 80 UBC889 DBD ACA CAC ACA CAC AC 2 2 50 UBC891 HVH TGT GTG TGT GTG TG 3 2 60 表三、分子變方成分分析表(分子變方成分分析)。
Table 3. Hierarchical analysis of molecular variance (AMOVA).
Source of variation df SSD MSD Variation component Total var.(%) p - value
Analysis among species
Variance among species 2 326.47 168.17 3.27 27.24 < 0.001 Variance within specie 141 1231.70 8.74 8.74 72.67 < 0.001
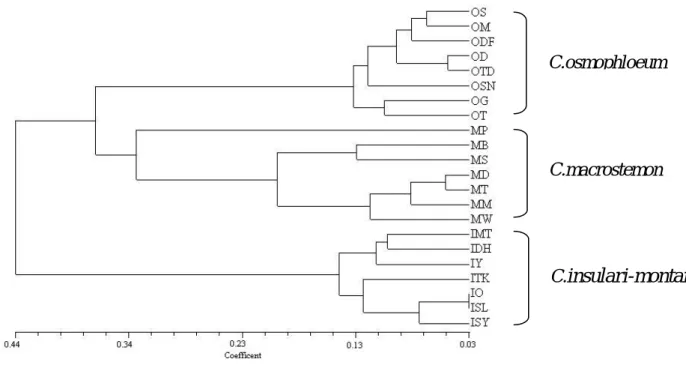
圖二、三種肉桂 ISSR 遺傳變異分析之歸群分析(UPGMA)樹狀圖。
Figure 2. UPGMA dendrogram based on ISSR bands of three Cinnamomum species.
coefficient)為 0.8143,顯示該歸群圖未扭曲可 表現出真實之群團狀態。由樹狀圖所呈現三群歸 群關係(Fig. 2),顯示出三種肉桂類植物之遺傳 分 化 。 主 座 標 分 析 ( Principal Coordinate Analysis,PCOA)分析結果之三維空間分佈座標 圖亦支持三群團關係(Fig. 3)。
討 論
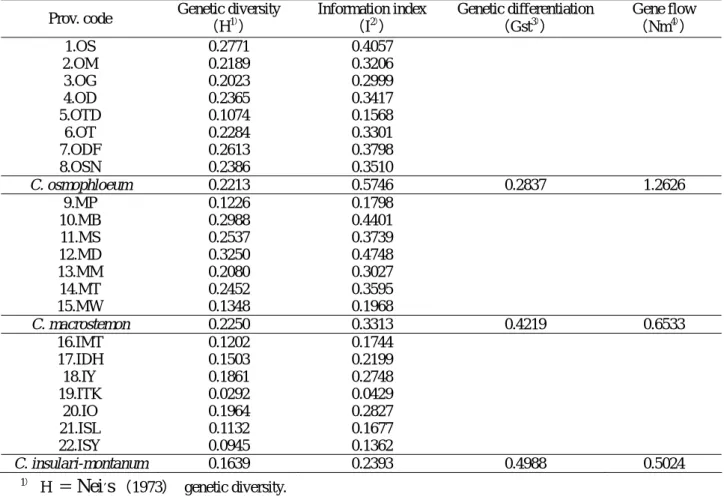
一、遺傳多樣性及族群分化 經由 AMOVA 分析結果(Table 3),顯示三種 肉桂植物間之變方成分是 27.24%(p<0.01),在 種內之變方成分是 72.67%(p<0.001),主要變異 來自各種內,同時顯示三種肉桂植物間之顯著分 化。同時,以 POPGENE 計算三種肉桂族群間之 遺傳分化程度(Nei, 1973),土肉桂之 Nei’s 基因 歧異度介於 0.1074 - 0.2771,總 Nei 基因歧異度 為 0.2213;胡氏肉桂之 Nei’s 基因歧異度介於 0.1226 - 0.3250,總 Nei 基因歧異度為 0.2250;山 肉桂之 Nei’s 基因歧異度介於 0.0292 - 0.1964,總 Nei 基因歧異度為 0.1639(Table 3)。依據 Hamricket al.(1992)研究所做的 Isozymeg 數據之統計結
果,蟲媒花異交植物之 Nei's 基因歧異度值平均 為 0.211,特有種到廣泛分佈種之 Nei's 基因歧異 度值為 0.078-0.257。因此,三種肉桂植物與同樣
圖三、三種肉桂 ISSR 分析之主座標分析座標圖。
Figure 3. Results of 3-dimensional principal coordinate
analysis based on ISSR bands of three Cinnamomum species. 為木本植物且具有蟲媒花、種子由風傳播等物種 之 遺 傳 歧 異 度 特 性 相 比 有 偏 高 現 象 。 Cheng (2003)應用 ISSR 研究玉山杜鵑(Rhododendron psudochrysanthum Hay.)之族 群遺 傳變異 結果 Nei’s 基因歧異度介於 0.1868 -0.2924,表現族群 具較度之遺傳岐異度之結構;Chen(2005)應用 ISSR 研究唐杜鵑(Rhododendron simsii Planch) 之族群遺傳變異,其 Nei’s 基因歧異度為介於 0.0647 - 0.2389 間表現族群具高度之遺傳岐異 度,參考上述相關研究所得之結果,土肉桂與胡 氏肉桂自然分佈族群具較高遺傳岐異度,山肉桂
C.osmophloeum
C.macrostemon
C.
insulari-montan
Kuen-Yih Ho
98 表四、POPGENE 分析表。
Table 4. POPGEN analysis of genetic diversities, Shannon’s information index, genetic differentiation, and gene flow.
Prov. code Genetic diversity (H1)
) Information index (I2) ) Genetic differentiation (Gst3) ) Gene flow (Nm4) ) 1.OS 0.2771 0.4057 2.OM 0.2189 0.3206 3.OG 0.2023 0.2999 4.OD 0.2365 0.3417 5.OTD 0.1074 0.1568 6.OT 0.2284 0.3301 7.ODF 0.2613 0.3798 8.OSN 0.2386 0.3510 C. osmophloeum 0.2213 0.5746 0.2837 1.2626 9.MP 0.1226 0.1798 10.MB 0.2988 0.4401 11.MS 0.2537 0.3739 12.MD 0.3250 0.4748 13.MM 0.2080 0.3027 14.MT 0.2452 0.3595 15.MW 0.1348 0.1968 C. macrostemon 0.2250 0.3313 0.4219 0.6533 16.IMT 0.1202 0.1744 17.IDH 0.1503 0.2199 18.IY 0.1861 0.2748 19.ITK 0.0292 0.0429 20.IO 0.1964 0.2827 21.ISL 0.1132 0.1677 22.ISY 0.0945 0.1362 C. insulari-montanum 0.1639 0.2393 0.4988 0.5024 1)
H =
Nei
,s
(1973) genetic diversity.2)
I = Shannon's information index
3) Gst = genetic differentiation. 4) Nm = gene flow. 之基因歧異度較前兩者為低之現象。 再由其遺傳分化係數 Gst 之差異,以土肉桂 遺傳分化係數為 0.2837 最低,而胡氏肉桂與山肉 桂遺傳分化係數比較高分別為 0.4219 及 0.4988; 由遺傳分化係數換算得基因流值,山肉桂與胡氏 肉桂基因流值比較低分別為 0.5024 及 0.6533,而 土肉桂基因流值則為 1.2626。Li et al.(2002)以 RAPD 研究瀕危植物 Vatica guangxiensis X.L.Mo 發現族群間存有高度分化的現象,而其 Gst 為 0.3764; Ge et al.(2003)以 ISSR 研究植物
Tetraena mongolica Maxim 其 Gst 為 0.169;本研
究胡氏肉桂、山肉桂之族群之遺傳分化係數超過 V. guangxiensis 與 T. mongolica 遺傳分化,顯然兩 者族群遺傳分化值偏高。 根據 Hamrick et al.(1989)自交植物具有最高的 Gst 值(平均 0.51),異交的風媒花植物之 Gst 值 最低(平均 0.143),異交的蟲媒花植物之 Gst 值 介於中間(平均 0.197),由此可見胡氏肉桂、山 肉桂之族群之遺傳分化係數極高,幾乎趨近於自 交植物。而與其他 ISSR 研究相比,Tsai(2001) 研究發現台灣地區水柳族群(Salix warburgii O. Seem)之 Gst 為 0.182,Cheng(2003)研究玉山 杜鵑(Rhododendron pseudochrysanthum Hay.) 之遺傳分化指數(Gst)為 0.2659,Chen(2005) 研究唐杜鵑之遺傳分化指數(Gst)為 0.5081,可 知胡氏肉桂、山肉桂之族群之遺傳分化係數上明 顯偏高,然而土肉桂遺傳分化係數為 0.2837,由 遺傳分化係數換算得基因流值,土肉桂基因流值 則為 1.2626,表現族群之遺傳基因流傳順暢,即 依據 Wright(1931)分析族群間的基因流值若大 值小於 1 則表示基因流可能受到地理阻礙,由於 基因流傳是族群遺傳結構分化的主因之一,若基 於 1,易使遺傳結構傾向均值化,反之,若 Nm 因流使兩個地方性族群的基因庫(gene pool)產
表五、三種肉桂遺傳變異分析之Nei's 遺傳距離(Φst)矩陣。
Table 5. Nei's genetic distance(Φst)based on ISSR data of three Cinnamomum species.
生交流,則會降低族群分化程度(Slatkin 1987), Ho et al.(2002)研究木賊葉木麻黃(Casuarina equisetifolia Forst.)發現了原生族群間基因流傳 低,受遺傳漂變影響族群間的分化程度趨於明 顯。因此,胡氏肉桂、山肉桂之族群具較高之遺 傳分化係數,可能是受到地理隔離影響,而使基 因流傳受到阻礙;土肉桂族群間之遺傳分化較 低,可能是目前族群廣泛分佈與數量多而較順暢 繁衍機制,使其基因流傳未受限制之結果。 二、族群之遺傳變異與分類地位關係 探討三種肉桂類植物族群之遺傳變異與分 類地位關係,族群歸群分析之歸群圖所呈現族群 親緣關係(Fig.2),其遺傳距離座標值介於 0.03 到0.44 之間,在距離 0. 20 處可將三種肉桂類植 物族群進行分類,第一群由土肉桂族群間之族群 組成;第二群是胡氏肉桂之族群,惟其中台北坪 林(MP)之介於與土肉桂群團之間,由於 Nei’s 基因歧異度為胡氏肉桂族群中較低者,其變異分 化相對明顯,顯示土肉桂與胡氏肉桂族群間之可 能有中間型之族群,因此兩群先合併再與第三群 合併,而第三群是山肉桂之族群。 整體而言三種肉桂之族群歸成三群團,土肉 桂、胡氏肉桂與山肉桂各自形成一群團,表現其 遺傳間之親緣關係。主座標分析結果其三維空間 座標圖(Fig.3),支持此三種肉桂族群遺傳變異 之關聯性。同時,族群間之自然分佈範圍亦多有 分隔趨勢,土肉桂族群如台中縣馬崙、台中縣 佳 保台、台中縣上谷關之族群則歸成一群,其間除 南投縣奧萬大、杉林溪等族群分佈位置相鄰而表 現密切關聯,餘族群於廣泛之地理區域。胡氏肉 桂在台北縣坪林、拔刀爾山族群等其間地理位置 十分相近。山肉桂則以台中縣東埔、高雄縣梅 蘭、新竹縣屯野生台山、新竹縣五峰清泉趨於成 一群,亦反映族群分佈之群聚關聯。 植物群落在不同環境選擇壓力下,外表形 態往往會顯出差異,由於三種肉桂族群自然分佈 於臺灣各生育地間,可能與當地環境發生地理分 化或生態變異,經由AMOVA 分析結果,顯示三 種肉桂植物間之遺傳分化明顯;歸群及主座標分 析結果印證了三種肉桂群團之分類趨勢,胡氏肉 桂之分類處理與 Lu and Yang(1986)之處理吻 合,同時反映一般辨識肉桂植物的特徵,如山肉 桂的芽麟是包被的且呈圓形,土肉桂的芽是裸露 的呈尖狀;雖胡氏肉桂與土肉桂外觀形態較相 似,但其頂芽芽鱗及葉、樹皮無肉桂味等明顯與 山肉桂特徵相似,故處理成另一群較為適宜。本 研究以三種肉桂之遺傳變異與其分化趨勢,處理 成三個分類群,即土肉桂(C. osmophloeum)、山 肉 桂 ( C. insulari-montanum )、 胡 氏 肉 桂 ( C. macrostemon)。
誌 謝
本 研 究 承 行 政 院 國 科 會 ( 93-2313-B-054- 013)計畫補助,林業試驗所育林組簡慶德博士OS OM OG OD OTD OT ODF OSN MP MB MS MD MM MT MW IMT IDH IY ITK IO ISL ISY OS 0 OM 0.09 0 OG 0.21 0.14 0 OD 0.11 0.10 0.16 0 OTD 0.17 0.13 0.19 0.08 0 OT 0.15 0.14 0.12 0.11 0.14 0 ODF 0.10 0.10 0.12 0.09 0.11 0.10 0 OSN 0.12 0.17 0.23 0.16 0.16 0.18 0.13 0 MP 0.52 0.54 0.64 0.59 0.65 0.64 0.59 0.57 0 MB 0.27 0.37 0.37 0.40 0.42 0.43 0.34 0.28 0.22 0 MS 0.31 0.38 0.44 0.48 0.55 0.46 0.38 0.31 0.31 0.16 0 MD 0.23 0.29 0.28 0.32 0.43 0.31 0.28 0.29 0.37 0.17 0.14 0 MM 0.33 0.35 0.33 0.35 0.45 0.33 0.34 0.35 0.41 0.32 0.21 0.09 0 MT 0.27 0.30 0.32 0.34 0.41 0.33 0.27 0.27 0.34 0.21 0.11 0.07 0.11 0 MW 0.43 0.39 0.38 0.42 0.48 0.45 0.41 0.44 0.46 0.38 0.29 0.16 0.14 0.12 0 IMT 0.51 0.63 0.54 0.49 0.67 0.61 0.50 0.48 0.62 0.40 0.54 0.41 0.45 0.42 0.44 0 IDH 0.46 0.50 0.43 0.43 0.65 0.49 0.43 0.48 0.58 0.38 0.54 0.32 0.35 0.38 0.45 0.12 0 IY 0.39 0.49 0.49 0.43 0.58 0.49 0.39 0.37 0.63 0.31 0.49 0.31 0.40 0.36 0.47 0.12 0.13 0 ITK 0.62 0.69 0.59 0.52 0.74 0.60 0.50 0.56 0.64 0.55 0.59 0.36 0.34 0.39 0.43 0.23 0.19 0.21 0 IO 0.41 0.52 0.46 0.43 0.56 0.45 0.37 0.40 0.56 0.32 0.41 0.23 0.29 0.26 0.36 0.17 0.12 0.11 0.11 0 ISL 0.38 0.50 0.45 0.45 0.57 0.46 0.33 0.39 0.71 0.39 0.44 0.26 0.30 0.28 0.36 0.20 0.15 0.15 0.17 0.05 0 ISY 0.51 0.57 0.55 0.48 0.66 0.53 0.44 0.47 0.72 0.48 0.50 0.32 0.28 0.33 0.36 0.15 0.12 0.15 0.14 0.08 0.11 0
Kuen-Yih Ho 100 提供部分樣品,蓮華池研究中心黃主任正良及同 仁等,中埔研究中心沈主任勇強等同仁全力協 助,謹致最大謝忱。
參考文獻
胡大維。1992。土肉桂專論--土肉桂的分佈、營 養系收集與營養系庫的建立。林業試驗所林 業叢刊第 38 號 1-6。 潘富俊。1992。土肉桂專論--土肉桂的分類地位。 林業試驗所林業叢刊第 38 號 7-13。Chang CE. 1976. Lauraceae in Li et al. Fl. Taiwan, Taipei:Epoch Pub.Co. 2:406-68.
Cheng JW. 2003. Genetic Variation of
Rhododendron pseudochrysanthum Based on
ISSR Markers . [MSc thesis]. Tai Chung, Taiwan : National Chung Hsing University. 78 p. [in Chinese with English summary].
Chen YJ. 2005. Genetic Variation of Rhododendron
simsii Based on ISSR Markers [MSc
thesis].Tai Chung, Taiwan: National Chung Hsing University. 90 p. [in Chinese with English summary].
Dice LR. 1945. Measures of the amount of ecologic association between species. Ecology 26: 297-302
Doyle JJ and Doyle JL. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissues. Phytochem Bull 19:11-5. Ge XJ, Yu Y, Zhao NX, Chen HS and WQ QI. 2003.
Genetic variation in the endangered Inner
Mongolia endemic shrub Tetraena Mongolia
Maxim. ( Zygophyllaceae ) . Biological Conservation 111: 427-34.
Excoffier L, Smouse PE and Quattro JM. 1992. Analysis of molecular variance inferred from metric distance among DNA haplotypes : Application to human mitochondrial DNA restriction data. Genetics. 131: 479-91.
Hamrick JL and Godt MJW. 1989. Allozyme diversity in plant species. In: Brown AHD, Clegg MT, Kahler AL, Weir BS, eds. Plant population genetics, breeding and genetic resources. Sunderland, MA: Sinauer Associates, 43-63.
Hamrick JL, Godt MJW and Sherman-Broyles SL. 1992. Factors influencing levels of genetic diversity in woody plant species. New Forests
6: 95-124.
Hayata B. 1913. Icones plantarum formosamarum Ⅲ.166-167, Bureau of Productive Industrices, Government of Formosa.
Ho KY, Ou CH, Yang JC and Hsiao JY. 2002. An assessment of DNA polymorphisms and genetic relationships of Casuarina
equisetifolia using RAPD markers. Bot Bull
Acad Sin 43: 93-8.
Hollinsworth ML, Hollinsworth PM, Jenkins GI, Bailey JP and Ferris C. 1998. The use of molecular markers to study patterns of genotypic diversity in some invasive alien Fallopia spp. ( Polygonaceae ) . Molecular Ecology 7: 1681-91.
Kanehira R. 1936. Formosan trees. rev. ed. Dept. Forestry, Government Research Institute. Li A and Ge S. 2001. Genetic variation and clonal
diversity of Psammochloa villosa (Poaceae) detected by ISSR markers. Annals of Botany 87: 585-90.
Li HL. 1963. Woody Flora of Taiwan. Livingston Publication Co. Pennsylvania.
Li HW. 1984. FLORA. Science Press. Beijing. Tomus31: 166-228.
Li Q, Xu Z and He T. 2002.Ex situ genetic conservation of endangered Vatica
guangxiensis ( Dipterocarpaceae ) in China.
Biological Conservation 106:151-6.
Liao JC. 1988. The taxonomic revisions of the family Lauraceae in Taiwan. Department of Forestry, College of Agriculture, National Taiwan University.
Liao JC. 1995. The taxonomic revisions of the family Lauraceae in Taiwan ( ED Ⅱ ) . Department of Forestry, College of Agriculture, National Taiwan University.
Liao JC. 1996. Lauraceae. In Flora of Taiwan Editorial Committee[eds.]. Flora of Taiwan. 2st edition. Vol.2: p.437-48.
Liu TS and Liao JC. 1971. A revision of the family Lauraceae in Taiwan. Annual of Taiwan Museum.14(1-28).
Liu YC, Lu FY and Ou CH. 1988. Trees of Taiwan. College of Agriculture, National Chung-Shing University. Monographic Publication No.7. No.7. p.105-13.
Liu YC, Lu FY and Ou CH. 1994. Trees of Taiwan (EDⅡ). College of Agriculture, National Chung-Shing University. Monographic
Publication No.7. p.107-12.
Lu SY, and Yang YP. 1986. Determination of the
Cinnamomum macrostemon Hayata. Quarterly
journal of Chinese forestry19(4): 113-7. Lu SY and Chiu WL. 1994. Rage and endangered
plants in Taiwan. The Council of Agriculture.p: 162
Nei M. 1973. Analysis of gene diversity in subdivided populations. Proc Nat Acad Sci USA 70: 3321-3.
Nei M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89: 583-90.
Prevost A and Wilkinson MJ. 1999. A new system of comparing PCR primers applied to ISSR fingerprinting of potato cultivars. Theor. Appl. Genet. 98: 107-12.
Qian W, Ge S and Hong DY. 2001. Genetic variation within and among populations of a wild rice Oryza granulata from China detected by RAPD and ISSR markers. Theor. Appl. Genet. 102: 440-9.
Rohlf FJ. 1993. NTSYS-pc numerical taxonomy and multivariate analysis system. New York:Applied Biostatistics. 256p
Sasaki S. 1928. List plants formosa. Natural History Society of Formosa Taihoku. P 192.
Shukla J, Tuteja U, Ratnaparkhe MB and Batra HV. 2001. Inter-simple-sequence repeat ( ISSR ) -PCR for the identification of saprophytic strains of Leptospira. World Journal of Microbiology & Biotechnology. 17 ( 5 ) : 529-33.
Slatkin M. 1987. Gene flow and the geographic structure of natural populations. Science 236: 787-92.
Tansley SA and Brown CR. 2000. RAPD variation in the rare and endangered Leucadedron
elimense(Proteaceae): implications for their
conservation. Biological conservation 95: 39-48.
Tsai CS. 2001. Genetic variation of Salix warburgii and S. kusanoi based on ISSR markers [MSc thesis].Tai Chung, Taiwan:National Chung Hsing University. 89 p. [in Chinese with English summary].
Wang CL. 1987. The yields and composition analyses of essential oils from planting
Cinnamomun osmophloeum Taiwan For Res
Inst New Series, 2(2): 129-44.
Wolfe AD, Xiang QY and Kephart SR. 1998. Assessing hybridization in natural populations of penstemon ( Scrophulariaceae ) using hypervariable intersimple sequence repeat ( ISSR ) bands. Molecular Ecology 7: 1107-25.
Wright S. 1931. Evolution in Mendelian populations. Genetics 16: 97-159.
Yang YP, Liu HY and Lu SY. 1999. Manual of Taiwan vascular plants. The Council of Agriculture Executive Yuan. Vol.2:131-4. Yeh FC, Yang RC, Boyle TBJ, Ye ZH and Mao JX.
1999. POPGENE 3.2, the user-friendly shareware for population genetic analysis. Molecular Biology and Biotechnology Centre, University of Alberta, Edmonton.
Yin HW. 1991. Yield and composition variation of essential oils from leaves of different
Cinnamomum osmophloeum Kanehira clones
in Taiwan. Quarterly Journal of Chinese Forestry 24(1): 83-104.
Zietkiewicz E, Rafalski A and Labuda D. 1994. Genome fingerprinting by simple sequence repeat ( SSR ) -anchored polymerase chain reaction amplification. Genomic 20: 176-88.
BioFormosa (2006) 41(2): 93-102
*Corresponding author: Kuen-Yih Ho; FAX: 886-5-2717467; E-mail: kyho@mail.ncyu.edu.tw 102
Genetic Variation and Taxonomic Relationship of Cinnamomum
osmophloeum, C. macrostemon and C. insulari-montanum
(Lauraceae)in Taiwan
Kuen-Yih Ho
*Department of Forestry and Natural Resouerces, National Chiayi University Chiayi, Taiwan
(Received: 4 August 2006, accepted: 6 December 2006)
ABSTRACT
Cinnamomum insulari-montanum Hayata, C. macrostemon Hayata and C. osmophloeum Kaneh. are
endemic species of the Lauraceae in Taiwan. Taxonomy of these closely related species remains unclear. In this study, ISSR fingerprinting using 12 microsatellite primers was employed to investigate the taxonomic status of the tree species and intraspecific diversity and differentiation. Analysis of molecular variance (AMOVA) revealed 27.24% variance residing among species(P<0.001), while 72.67% variance distributed within species ( P<0.001). Significant structuring exists in C. Insulari-montanum and
C.macrostemon; while gene flow ,as indicated by Nm=1.26, was detected in C.osmophloeum. Both
clustering and principal coordinates analyses Identified three major clusters that correspond to each species and revealed a close Phonetic relations between C. Insulari-montanum and C. macrostemon.
Key words: C. insulari-montanum, C. macrostemon, C. osmophloeum, ISSR, genetic variation,