計畫名稱:蝴蝶蘭光合作用酵素 PEPC 基因之研究
Studies on the PEPC genes of photosynthesis in phalaenopsis
計畫編號:NSC 89-2311-B-006-011
執行期限:89 年 8 月 1 日至 91 年 1 月 31 日
主持人:
吳文鑾計畫參與人員:黃明德 吳雨哲
執行單位:國立成功大學
一、 中文摘要
磷酸烯醇丙酮酸羧化 (phosphoenolpyruvate carboxylase, PEPC)經由β-carboxylation 將磷酸烯醇丙酮酸(phosphoenolpyruvate, PEP)轉化為草醯乙酸(oxaloacetate)及 Pi,為光合作用 的關鍵酵素。前人的研究指出 PEPC 基因具有由小型基因族系所轉錄、轉譯的異構型,大致 可以分為:C3 型、C4 型與 CAM 型。截至目前為止已有多種植物的 C3、C4 型 PEPC 基因被 選殖出來,但對於 CAM 植物之 PEPC 基因的研究卻仍相當有限。蝴蝶蘭為絕對性的 CAM 植 物,是研究 PEPC 基因的理想材料,因此本研究利用分子生物技術選殖台灣原生種蝴蝶蘭:
姬蝴蝶蘭(Phalaenopsis equestris)與台灣阿媽(Phalaenopsis amabilis)之 PEPC 基因,並探討其基 因組成、結構、基因表現與親源演化關係。首先由基因資料庫中植物 PEPC 基因序列之高保 留區域設計簡併引子(degenerate primer),擴增姬蝴蝶蘭與台灣阿媽的 PEPC 基因片段並選殖 定序,得到 1855 與 1848 bp 的基因組序列。接著利用此 PEPC 基因片段為探針與蝴蝶蘭基因 組 DNA 進行南方雜合反應,研究蝴蝶蘭 PEPC 基因的組成情形,結果顯示蝴蝶蘭 PEPC 基因 族系為至少包含兩個成員的小型基因族系。由於上述蝴蝶蘭 PEPC 基因片段在 5’端不完整,
因此以快速擴增基因組末端法(Rapid Amplification of Genomic Ends)進行蝴蝶蘭 PEPC 基因 5’
端序列的選殖,得到包含起始密碼上游 63 bp 至下游 168 bp 處的基因片段。此外,也以 RT-PCR 產物作為模板擴增 PEPC 基因之 cDNA 片段並進行選殖與定序,得到之 PEPC 基因 cDNA 序 列再與上述選殖到的基因組序列之表現子序列組合。在姬蝴蝶蘭與台灣阿媽之 cDNA 序列都 可轉譯出 965 個氨基酸之完整啟解讀碼 (open reading frame, ORF)的 2898 bp 基因序列。將此 兩種蝴蝶蘭 PEPC 基因序列與其他植物 PEPC 基因序列進行比對分析,並以聚類分析法構築 基因演化樹,結果顯示 PEPC 基因異構型之分群現象傾向於支持 CAM 型是由較原始的 C3 型 演化而來的論點。
關鍵詞:蝴蝶蘭 磷酸烯醇丙酮酸羧化酵素 基因表現 分子親緣
Abstract
Phosphoenolpyruvate carboxylase (PEPC) catalyzes theβ-carboxylation of phosphoenolpyruvate to yield oxaloacetate and Pi, and is the key enzyme of photosynthesis. It has been reported that plant PEPC including C3, C4 and CAM isoforms are encoded by small gene family. Many PEPC isoforms of C3 and C4 plants have been identified, but little is known about the PEPC genes of CAM plants. Phalaenopsis orchids are obligate CAM plants, and therefore being suitable materials for studying PEPC genes. In this study, we cloned PEPC genes from two native orchids of Taiwan:
Phalaenopsis equestris and Phalaenopsis amabilis, and investigated gene organization, structure, expression and phylogenetic relationships. Primers were designed from highly conserved regions of other plant PEPC genes, and used for the PEPC genomic sequence amplification. Two clones coding for 1855 and 1848 bp PEPC genomic sequences from Phalaenopsis equestris and Phalaenopsis amabilis were isolated and sequenced, respectively. Southern hybridization of Phalaenopsis genomic DNA with these fragments suggested that there are at least two members in Phalaenopsis PEPC gene family. Since these PEPC fragments mentioned above are not complete in the 5’ region, their upstream genomic sequences were cloned by RAGE (Rapid Amplification of Genomic Ends)( Cormack and Somssich, 1997) method. The resulting sequences are ranging from 63 bp upstream to 168 bp downstream of start codon. Furthermore, cDNA sequences of PEPC gene were also cloned by RT-PCR and subjected to sequencing. These obtained cDNA sequences were then assembled with the exon sequences of genomic clones described above. Both Phalaenopsis equestris and Phalaenopsis amabilis cDNA contig are with an open reading frame (ORF) of 965 amino acids. Both nucleotide and deduced amino acid sequences of PEPC genes of Phalaenopsis orchids and other plants were aligned and analyzed by neighbor-joining method. Phylogenetic analysis of PEPC genes provided support in favor of the view that CAM isoform was derived from
the primary C3 isoform under the stress of environments.
Keywords: Phalaenopsis, phosphoenolpyruvate carboxylase, gene expression, molecular phylogeny,
二、緣由與目的
磷酸烯醇丙酮酸羧化酵素(phosphoenolpyruvate carboxylase, PEPC)是光合作用型式 為 CAM 型及 C4 型植物中初級固定二氧化碳的關鍵酵素之一,使植物能更有效率地利 用水分和氮源,以適應強日照、高溫、乾旱的環境。磷酸烯醇丙酮酸羧化酵素具有不 同異構型(isoform),在植物基因組中大多是由小型的多基因族系(multigene family)所轉 譯(Gehring et al., 1995; Cushman and Bohert, 1998a, 1998b; Kawamura et al. 1990)。有關 CAM 特異表現的 PEPC 異構型基因成員,目前只在很少數兼性(facultative)CAM 型、
一種絕對性(obligate)CAM 型植物被研究過。台灣蝴蝶蘭(Phal. amabilis)為絕對性 CAM 植物,對二氧化碳的吸收呈現日韻律現象(diurnal rhythm) (Avadhani et al., 1982; Hew et al., 1996),依據前人研究發現其初生葉主行 C3 型代謝,十天後轉為完全 CAM 型代謝,
此種光合作用型式轉換與發育時期之調控機制仍不清楚,因此蝴蝶蘭是作為探討不同 PEPC 異構型基因的功能、組織與器官特異性表現以及光合作用型式代謝調控之理想模 式植物。本計畫由選殖蝴蝶蘭 PEPC 基因著手,比較分析不同原生種之基因組成、結 構與基因族系之基因成員間分子演化親緣,並由基因上游序列進行功能分析,鑑定與
基因表現有關之序列,並進一步探討其與光合作用型式代謝調控之分子機制。因此本
研究結果將對蝴蝶蘭光合作用的分子層次特性有較清楚的瞭解,並能提供蝴蝶蘭產業 應用上的有益資料。
三、結果與討論
(一)蝴蝶蘭 PEPC 基因片段之選殖與序列分析
依據選殖自七種植物的九個 PEPC 基因之譯碼區中的高保留序列設計引子分別以台灣原 生種的姬蝴蝶蘭 (Phalaenopsis equestris) 與台灣阿媽 (Phalaenopsis amabilis var. formosa) 之 基因組 DNA 為模板,進行聚合酵素連鎖反應擴增蝴蝶蘭的 PEPC 基因片段,產物經電泳分析 後於 2 kb 處出現可能為 PEPC 基因片段的 DNA 條帶,將 DNA 條帶回收並純化後與 pGEM-T Easy Vector 進行接合作用。由姬蝴蝶蘭與台灣阿媽選殖質體分別定序出 1855 bp 與 1848 bp 的 PEPC 基因片段。與已發表之高粱 (Sorghum vulgare) 及香草 (Vanilla planifolia) 之 PEPC 基因序列相比較,結果顯示此二個 PEPC 基因選殖片段中皆含有三個插入子,在 5’端尚缺少 包含轉譯起始密碼 (start codon) 的核甘酸序列,在 3’端則還缺少包括六個表現子的序列。以 此二個蝴蝶蘭 PEPC 基因片段序列進行初步的核 酸相似度比對,發現除了在表現子 III 的相 似度較低 (89 %),二個蝴蝶蘭 PEPC 基因片段序列在插入子及表現子的序列相似度皆大於 94
%,顯示姬蝴蝶蘭與台灣阿媽的 PEPC 基因序列具有頗高的保留性。分析蝴蝶蘭 PEPC 基因組 片段之序列,發現其中包含表現子 I~IV 與插入子 I~III,於所有插入子的剪接位置 (splice site) 上,皆存在著插入子 5’端以 GT 為開端,3’端以 AG 做為結尾的剪接保留序列 (consensus sequence)。與其他植物如: 玉米 (Zea mays, accession No. X15642), 高粱 (Sorghum vulgare, accession No. X59925)之 PEPC 基因組序列比對發現在表現子序列保留性,相似度介於 64-1%;而插入子 I~III 在序列長度上也是變化頗大,因此序列保留性也偏低。總結來說,PEPC 基因的表現子序列在不同物種間具有較高的保留性,而插入子序列則缺乏保留性,但同一物 種的插入子序列卻仍具有頗高的保留性,如:姬蝴蝶蘭與台灣阿媽;因此推論插入子序列雖
然在物種間的保留性偏低,卻可能是用來區別不同物種的依據;也可解釋為何 PEPC 基因的 插入子會多達九個。分析姬蝴蝶蘭與台灣阿媽 PEPC 基因之 cDNA 序列並將其轉譯成氨基酸 序列,結果 PEPC 基因之 cDNA 序列可轉譯成包含十個表現子,全長為 965 個氨基酸的完整 PEPC 基因,姬蝴蝶蘭與台灣阿媽 PEPC 基因之 cDNA 序列及氨基酸序列的相似度非常高,皆 為 97%,顯示蘭科植物之 PEPC 基因序列具有很高的保留性。在姬蝴蝶蘭與台灣阿媽 PEPC 基因之氨基酸序列中都可找到與其他植物相同的重要氨基酸 motif, 如:可調節的磷酸化位置 (regulatory phosphorylation site)— SIDA (氨基酸位置 11-14);活化中心 (active centre)—
GYSDSGKDAG (氨基酸位置 594-603) 以及受質結合位— FHGRGGTVGRGGGP (氨基酸位置 632-645);而且其分佈位置與 1998 年發表之蘭科植
物
:香草(Vanilla planifolia)的 PEPC 基因 序列相當接近 (其氨基酸位置分別是 11-14, 597-606, 635-648) (Gehrig et al., 1998a);但在高粱 (Sorghum vulgare) 與玉米 (Zea mays) 之 PEPC 基因序列中,這些區域的氨基酸位置則分別是 8-11, 698-707, 736-749 與 15-18, 600-609, 638-651 (Lepiniec et al., 1993 ; Dong et al., 1998),顯 示這些區域不但具有極高的保留性,而且在蘭科植物之 PEPC 基因序列的分佈位置也很保守。(二) 蝴蝶蘭 PEPC 基因族系的組成
將姬蝴蝶蘭與台灣阿媽之基因組 DNA 以特定限制酵素切割並電泳分離後,進行南方轉印 實驗,由於姬蝴蝶蘭與台灣阿媽的 PEPC 基因序列的保留性頗高,因此選用限制酵素 EcoR I 切割選殖質體,電泳分離並純化回收 740 bp 的插入片段 (包含姬蝴蝶蘭 PEPC 基因部份表現 子 I 及 II 與插入子 I) 以隨機引子法製作蝴蝶蘭 PEPC 基因序列的放射性探針。結果顯示經限 制酵素 EcoR I、BamH I 與 Hind III 切割的姬蝴蝶蘭基因組 DNA 都只顯示一個雜合反應條帶;
經限制酵素 EcoR I、BamH I 與 Hind III 切割的台灣阿媽基因組 DNA 則分別顯示二個、三個 與一個的雜合反應條帶。另外,也將其他品系之蝴蝶蘭與朵麗蘭之基因組 DNA 以限制酵素 EcoR I 切割並電泳分離後,進行南方轉印實驗,並利用上述之探針進行南方雜合實驗,結果 除了姬蝴蝶蘭只顯示一個雜合反應條帶,其他品系之蝴蝶蘭與朵麗蘭皆顯示二至三個雜合反 應條帶。由於用於製備探針的 PEPC 基因序列沒有 EcoR I 與 Hind III 的切點,因此初步推測 大部份蝴蝶蘭的 PEPC 基因為至少包含二個成員的小型基因族系。
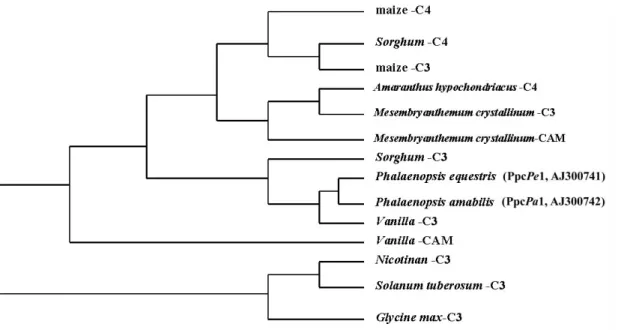
(三) 蝴蝶蘭 PEPC 基因之演化分析
將姬蝴蝶蘭與台灣阿媽 PEPC 基因之 cDNA 序列及台灣阿媽 PEPC 基因譯碼區之氨基酸 序列以網際網路傳送至國家衛生研究院 GCG 之 SeqWeb,與基因庫中其他植物已登錄之 PEPC 基因核甘酸與氨基酸序列分別進行排序與比對,然後以聚類分析法 (neighbor-joining method) 建構 PEPC 基因演化樹。結果無論是以核甘酸序列或氨基酸序列所建構之基因演化樹皆顯示 蝴蝶蘭 PEPC 基因與 Vanilla planifolia 之根型 PEPC 基因 (VpR) 最為相近,推測可能是由於 蝴蝶蘭與 Vanilla planifolia 同為蘭科植物,故其序列相似度較高。此外,觀察以 PEPC 核甘酸 序列所構築之演化樹,發現其可明顯區分為單子葉與雙子葉植物兩大群,而單、雙子葉植物 的 PEPC 基因異構型可再各自分為 CAM 型及 C3 型的小分群;在 PEPC 氨基酸序列所建構的 演化樹也可發現這種類似的分群現象;這樣的分群結果與 Gehrig 等人於 1998 年以涵蓋藻類、
苔蘚、蕨類及種子植物門的 12 種植物之 PEPC 基因 3’端的 1100 bp cDNA 序列所構築之演化 樹相吻合(Gehrig, 1998b);Gehrig 等人依據這種分群現象推論 PEPC 基因在很古老的年代就已 存在於植物體的基因組中,甚至比單、雙子葉植物之分化更早,所以利用 PEPC 基因序列建 構之演化樹都可區分為單子葉與雙子葉兩大分群;而 C3 型 PEPC 為最原始的 PEPC 基因異 構型,PEPC 的其他異構型 (CAM 型及 C4 型) 則是後來植物為了適應各種惡劣環境而演變出 來的 (Gehrig et al., 1998b);因此依據本研究構築之基因演化樹的分群現象也傾向於支持這種 推論。
四、參考文獻
Avadhani, P. N., Goh, C. J., Rao, A. N. and Arditti, J. (1982) Carbon fixation in orchids. p.173-193.
In: Orchid Biology: Reviews and Perspective II. J. Arditti (ed). Cornell Univ. Press. New York.
Cormack, R. S. and Somssich, I. E. (1997) Rapid amplification of genomic ends (RAGE) as a simple method to clone flanking genomic DNA. Gene 194: 273-276.
Cushman, J. C. and Bohnert, H. J. (1989a) Nucleotide sequence of the Ppc2 gene encoding a housekeeping isoform of PEPC from M. crystallinum. Nucleic Acids Res. 17: 6743-6744.
Cushman, J. C. and Bohnert, H. J. (1989b) Nucleotide sequence of the gene encoding a CAM specific isoform of PEPC from M. crystallinum. Nucleic Acids Res. 17: 6745-6746.
Dong, L. Y., Masuda, T., Kawamura, T., Hata, S. and Izui, K. (1998) Cloning, expression and characterization of a root-form phosphoenol-pyruvate carboxylase from Zea mays: comparison with the C4-form enzyme. Plant Cell Physiol. 39: 865-873.
Gehrig, H., Taybi, T., Kluge, M. and Brulfert, J. (1995) Identification of multiple PEPC isogenes in leaves of the facultative Crassulacean acid metabolism plant Kalanchoe blossfeldiana. FEBS Letters 377: 399-402.
Gehrig, H., Faist, K. and Kluge, M. (1998a) Identification of phosphoenolpyruvate carboxylase isoforms in leaf, stem and roots of the obligate CAM plant Vanilla planifolia Salib. (Orchidaceae):
a physiological and molecular approach. Plant Mol. Biol. 38: 1215-1223.
Gehrig, H., Heute, V. and Kluge, M. (1998b) Toward a better knowledge of the evolution of phosphoenolpyruvate carboxylase by comparison of partial cDNA sequences. J. Mol. Evol. 46:
107-114.
Hew, C. J. and Yong, J. W. H. (1996) Photosynthesis. p.38-92. The physiology of tropical orchids in relation to the industry. World scientific publishing.
Kawamura, T., Shigesada, K., Yanagisawa, S. and Izui, K. (1990) Phosphoenolpyruvate carboxylase prevalent in maize roots: isolation of a cDNA clone and its use for analyses of the gene and gene expression. J. Biochem. 107: 165-168.
Lepiniec, L., Keryer, E., Philippe, H., Gadal, P. and Cretin, C. (1993) Sorghum phosphoenolpyruvate carboxylase gene family: structure, function and molecular evolution. Plant Mol. Biol. 21: 487-502.
P. equestris 1 MSRSTVERHASIDAQLRLLAPRKVSEDDKLVEYDALLLDRFLDLLQEIHG 50 P. amabilis 1 MSRSTVERHASIDAQLRLLAPRKVSEDDKLVEYDALLLDRFLDILQEIHG 50 P. equestris 51 EDIRETVQECYELSAEYEGNHDSKKLEELGHVLTSLDPGDSIVAAKSFSN 100 P. amabilis 51 EDIRETVQECYELSAEYEATHDSKKLEELGHVLTSLDPGDSIVVAKSFSN 100 P. equestris 101 MLNLANLAEEVQIAFRRRIKLKKGDFVDENSAATESDIEETLRRLVHELK 150 P. amabilis 101 MLNLANLAEEVQIAFRRRIKLKKGDFVDENSAATESDIEETLRRLVHELK 150 P. equestris 151 KFPEEVLDALKNQTIDLVFTAHPTQSVRRSLLQKHGRIRNCLAQLYAKDI 200 P. amabilis 151 KSPEEVFDALKNQTIDLVFTAHPTQSVRRSLLQKHGRIRNWLLQLYAKDI 200 P. equestris 201 TPDAKQELDEALQREIQAAFRTDEIRRTAPTPQDEMRAGMSYFHETIWKG 250 P. amabilis 201 TPDDEQELDETXQREIQAAFRTDEIRRTAPTPQDEMRAGMSYFHETIWKG 250 P. equestris 251 VPKFLRRVDTALKNIGINERVPYNAPLIQFSSWMGGDRDGNPRVTPEVTR 300 P. amabilis 251 VPKFLRRVDTALKNIGINERVPYNAPLIQFSSWMGGDRDGNPRVTPEVTR 300 P. equestris 301 DVCLLARMMAANLYFSQIEDLMFELSMWRCSDELRVRADELHRASKKDAE 350 P. amabilis 301 DVCLLARMMAANLYFSQIEDLMFELSMWRCSDELRVRADELHRASKKDAK 350 P. equestris 351 HYIEFWKQVPPNEPYRVVLADVRDKLYNTRERSRHLLSSGYSDIPEETTL 400 P. amabilis 351 HYIEFWKQVPPNEPYRVVLADVRDKLYNTRERSRHLLSSGSSDIPEETTL 400 P. equestris 401 TNVEQFLEPLELCYSSLCTCGDRPIADGSLLDFMRQVSTFGLSLVRLDIR 450 P. amabilis 401 TNVEQFLEPLELCYSSLCTCGDRPIADGSLLDFMRQVSTFGLSLVRLDIR 450 P. equestris 451 QESDRRTDVLDAITTHLGIGSYRDWSEEQRQEWLLSELSGKRPLFGPDLP 500 P. amabilis 451 QESDRHTDVLDAITTHLGIGSYRDWSEEQRQEWLLSELSGKRPLFGPDLP 500 P. equestris 501 KTEEIADVLDTLRVIAELPHDSFGAYIISMATAASDVLAVELLQRECQVK 550 P. amabilis 501 KTEEIADVLDTLRVIAELPHDSFGAYIISMATAASDVLAVELLQRECQVK 550 P. equestris 551 KPLRVVPLFEKLADLEAAPAALSRPFSINWYRNRIDGKQEVMIGYSDSGK 600 P. amabilis 551 KPLRVVPLFEKLADLEAAPAALSRLFSINWYRNRIDGKQEVMIGYSDSGK 600 P. equestris 601 DAGRLSAAWQLYKAQEDLIKVAKEFGVKLTMFHGRGGTVGRGGGPTHLAI 650 P. amabilis 601 DAGRLSAAWQLYKAQEDLIKVAKEFGVKLTMFHGRGGTVGRGGGPTHLAI 650 P. equestris 651 LSQPPDTIHGSLRVTVQGEVIEQSFGEEHLCFRTLQRFTAATLEHGMHPP 700 P. amabilis 651 LSQPPDTIHGSLRVTVQGEVIEQSFGEEHLCFRTLQRFTAATLEHGMHPP 700 P. equestris 701 ISPKPEWRALLDEMAVVATKEYRSIVFQEPRFVEYFRLATPETEYGRMNI 750 P. amabilis 701 ISPKPEWRALLDEMAVVATKEYRSIVFQEPRFAEYFRLATPETEYGRMNI 750 P. equestris 751 GSRPSKRKPSGGIESLRAIPWIFAWTQTRFHLPVWLGFGTAFKHAIAKDI 800 P. amabilis 751 GSRPSKRKPSGGIESLRAIPWIFAWTQARFHLPVWLGSGTAFKHVVAKDI 800 P. equestris 801 KNLNMLQEMYNEWPFFRVTIDLVEMVFAKGDPGIAALYDKLLVSDDLGHF 850 P. amabilis 801 KNLYMLQEMYNEWPFFRVTIDLVEMVFAKGDPGIAALYDKLLVSDDLWSF 850 P. equestris 851 GERLRANFEETKTLLLQVAGHRDLLEGDPYLKQRLRLRDAYITTLNVCQA 900 P. amabilis 851 GERLRANFEETKTLLLQVAGHRDLLEGDPYLKQRLRLRDAYITTLNVCQA 900 P. equestris 901 FTLKRIRDPSFHVNLRPHLSREIMNSNKPAAELVKLNPTSEYAPGLEDTL 950 P. amabilis 901 FTLKRIRDPSFHVNLRSHLSREIMNSNKPAAELVKLNPTSEYAPGLEDTL 950 P. equestris 951 ILTMKGIAAGLQNTG* 965
P. amabilis 951 ILAMKGIAAGMQNTG* 965
Fig. 1 Alignment of Phalaenopsis equestrisand Phalaenopsis amabilis PEPC amino acid Sequences
Both of Phalaenopsis equestris(PpcPe1, GenBanK accession number AJ300741) and Phalaenopsis amabilis(PpcPa1, GenBanK accession number AJ300742) PEPC encode 965 amino acids, and the deduced amino acid sequences were aligned, showing 97% identity. The different amino acids between two PEPCs are singly underlined. The amino acid
motifs also known from the PEPC of other plants, the regulatory phosphorylation, active centre and the substrate-binding site are doubly underlined, respectively.
Fig. 2 Putative phylogenetic r elationship of the twoPhalaenopsis PEPC genes
Amino acid sequences of PEPC used here are taken from the GeneBank database. The tree was constructed according to the neighbor-joining method.