平行運算應用於 SOM 神經網路:以生物群聚分析為例
1蔡維仁
2陳彥璋
3許逸揚
4謝昆霖
123國立台東大學資訊管理學系
4國立台東大學資訊管理學系暨電子計算機中心
1siaoming.tsai@gmail.com
2joke12@msn.com
3renovartio@gmail.com
4klhsieh@nttu.edu.tw
摘要
近年來,由於多重處理器架構以及叢集式電腦架構的提出,平行式運算也越來越受到產官學研的重 視,隨著多核心的處理器逐漸普及,站在資源充分利用的角度,如何有效利用這些額外的運算資源便成 為一值得省思的問題。在本研究中,我們利用一具多重處理器特性之混合式叢集電腦架構搭配 MPI 函式 庫來達成平行運算的目的,每個節點配置兩個邏輯處理器,並利用電腦教室的環境搭配各種不同的測試 狀況來探討電腦教室中實施平行運算的可行性。其中,我們測試了包括 NAT 環境、開放式網路環境以及 完全阻絕廣域網路三種不同架構的測試,在 NAT 之下我們架設一獨立之子網路,透過 NAT Gateway 對 廣域網路連結,此為一常見的狀況。而在開放式架構下叢集電腦子網路並沒有和廣域網路有明顯的區隔, 由此可以測試在校園內不同組織部門間叢集電腦系統的效率。最後也安排一完全阻絕對外連結之網路架 構,以測試 Noise 對叢集電腦效能之影響,最後,我們以一個生物群聚分析個案為例,利用類神經網路之 自組織映射圖為基礎,藉以驗證叢集架構統整資源達成群聚分析之可行性與合理性。 關鍵詞:叢集運算、群聚分析、MPI、自組織映射圖網路。1. 前言
1.1 動機 目前,為執行一些龐大運算需求的應用如進行 生物 DNA 序列的物種群聚分析(Clustering analysis)(Jeng et al., 2005)或是利用電腦設備進行 即時性的資料挖掘群集應用(Data mining)等相關 工作(吳旭智、賴淑貞,2001),在建構系統時大多 傾向於購置昂貴的大型主機或是工作站來完成這 些工作,不僅耗費大量的金錢,也浪費許多現有的 資源。而現今大學的實驗室或電腦教室之中有著許 多的計算資源,包括像是電腦、工作站以及各類伺 服器等,而這些設備並不是全天候保持著全速運作 著;即使是有人在使用,也並非無時無刻保持滿載 的情形。在不是所有的程式都對多核心處理器最佳 化的情況下,經常我們可以發現到處理器有一個核 心是完全閒置的;即使是較舊款的單核心處理器, 也並非無時無刻保持在 Full Load 的狀況。於是我們 開始思考如何利用這些閒置的運算資源,而叢集電 腦正是我們思考的一個方向。有鑑於此,如果能夠 善加利用電腦教室或實驗室中閒置的運算資源,便 可有效的降低計算成本,而達到資源整合的目的。 1.2 目的 根據前述的動機,我們將置重點於發展可達成 群聚分析(Clustering Technology)的叢集式電腦應用 架構,並以現階段在產官學研中最為廣泛應用的智 慧型群聚技術-類神經網路之自組織映設圖網路 (Self-Organizing Map,簡稱 SOM)為主要的實踐對 象(Kohonen 1984; Kohonen 1982; 葉怡成,2003), 透過實驗性的網路環境建置叢集式電腦的架構來 實現線上群聚分析的功能,並且也透過要所規劃的 實驗網路架構(封閉型或是開放型的網路環境搭配 不同數量的叢集電腦數)來進行各項系統效率或校 能的比較與評析。2. 文獻整理
2.1 叢集式運算介紹 將許多個人電腦以乙太網路連結,用以實現分 散式運算,這樣的網路環境便可以稱為叢集式電腦 環境(Sterling et al., 1999),何謂叢集式電腦?目前 常見的 Cluster (叢集)架構有兩種,一種是 Web / Internet cluster system,這種架構主要是將資料分配 在各個節點上面,也就是由多部主機同時負責一項 服務;另一種則是本案所使用的平行運算系統。平行運算就是將原來由單一主機所負責的工作,分配 給整個叢集系統內的所有處理器來進行同步運算 的一個功能。 最初這樣的架構大多用在大型主機上面,隨著 技術逐漸成熟,一般的 PC 個人電腦也可以使用類 似的技術了。由於 Cluster 技術可以使用到多顆處理 器,對於效能的提升將會有莫大的助益,且他不需 要特殊規格的高速電腦,只要一般的 PC 個人電腦 便可以實現,在資源充分利用的邏輯下,叢集電腦 讓許多舊電腦起死回生,有著聚沙成塔的效果,正 因為如此,它又稱為「窮人的超級電腦」。不過, 也需要特別留意的是,由於叢集電腦的架構之中, 會將一個工作平均分配給各節點,故若有某些節點 完成時間較慢,會導致其餘節點暫停工作,等待這 些節點完成,故一般而言叢集電腦的各節點最好是 等級相同的電腦,才不會耗費無謂的時間在等待上 (蔡德明,2003;Sterling et al., 1999; Sloan, 2004)。
2.2 主從式架構介紹 圖 1. 主從式架構示意圖 上圖中每一個點都代表一顆處理器。以 MPI 架構為例,Master 主機上必須要有 MPI 的函式庫, 此 Master 節點主要的任務是負責分配工作,而在 Master 主機上最重要的軟體就是:(1)MPICH;(2) 編譯器(compiler, 例如 Fortran)而在以乙太網路作 為各節點間溝通媒介的叢集電腦系統中,我們主要 利用三套重要的軟體來協助使用者操作: 1) R Shell, 亦即稱為 RSH;
2) NIS,使 Master 與 Slave 具有相同的帳號 群組關係 3) NFS,使讀取寫入的資料可以在同一個 partition 上面。 2.3 RSH 一般而言,Linux 主機上我們大多以 Bash 作為 Shell System,但若是有在不同主機間下達指令的需 求,那就可以使用 R Shell 來進行指令的下達。如 此一來,在管理與設定上將會更加方便。須要注意 的是,RSH 本身是一種相當危險的服務,由於使用 者可以直接登入 RSH 主機,並且在上面進行指令 的下達,為了避免還在各主機之間切換還要再次輸 入密碼的問題,因此通常 RSH 已經自動將叢集系 統內的各節點都設定為信任來源。不過,RSH 會啟 動一些 port 來監聽 Clients 的需求,故在 Master 系統的設定上安全性便變得非常的重要,一般建議 整組系統放在私有網域當中,並給予私有 IP 位址 (鄭士豪、林英超、蕭景鴻,2002)。
2.4 MPICH
MPI 是 Messages Passing Interface 的縮寫,是 一個規格相當嚴謹的通訊標準,用以處理平行運算 之間各節點間的資料交換,MPICH 就是符合 MPI 標準通訊協定所設計出來的一套軟體。因此,我們 可以利用 MPICH 所提供的 MPI 函式庫來達成平 行運算的功能。MPICH 是由 Mathematics and Computer Science Division 的 Argonne 實驗室所發 展,詳細的資料可以參考:
http://www-unix.mcs.anl.gov/mpi/mpich/
2.5 SOMNN 之介紹
自 組 織 映 射 神 經 網 路 (self-organizing map, SOM) , 或 又 稱 作 自 我 組 織 特 徵 映 射 圖 網 路 (self-organizing feature map network, SOFM),是由芬 蘭 赫 爾 辛 基 大 學 Teuvo Kohonen 教 授 所 提 出 (Kohonen 1984; Kohonen 1982)。自組織神經網路基 本為一個一維或二維空間的網路圖, 輸入向量可 藉由映射(mapping)投影的方式,將維度之輸入向量 對映至二維或一維的空間上,此一功能可提供高維 度之資料可以二維空間來呈現其彼此之相似性,以 利資料分群之進行(Vesanto and Alhoniemi 2000)。自 組織神經網路具有臨近區域相似性高的特色,輸入 層與輸出層皆同屬在一個一維或二維的空間上,以 特 定 拓 樸 結 構 (topological structure) 如 矩 陣 (grid matrix)或多邊型之排列法來聚集網路層之神經元。 SOM 的 正 式 演 算 法 如 圖 2 所 示 (Jang, Sun & Mizutani, 1997)。演算法說明如下表:
圖 2. SOM 網路演算法說明 2.6 IPTABLES 與 NAT 2.6.1 NAT NAT 定義於 RFC 1631,其原理在於藉由置換 IP Header 之動作,讓進入路由器的封包轉送到他們 應去的子網路節點,以便讓多台電腦能共用一個 IP 位址連結至網際網路。目前許多的 router 目前有都 有支援 NAT 這項功能, 其它如 Linux 中的 IP Masquerade, FreeBSD 中的 NATD, 或是 Win95 上的 Sygate 軟體也都是為了支援 NAT 服務而開發 的。. NAT 一般多用在虛擬區域網路,讓網路內的各 個電腦透過一台 NAT Server 上網。 NAT 的優點如下: z 由於對外只使用一個 IP address, 因此內 部使用的 IP 可重覆地在不同單位使用。 z 只要少數真實 IP 就能讓單位內所有電腦 都連上 Internet。 z 只有使用真實 IP 的電腦會被單位外部網 路所存取, 使用虛擬 IP 的電腦不會直接 被存取, 在安全性的考量上這種架構形 同天然的防火牆。 而 NAT 的缺點如下: z 通訊協定資料中如含有其 IP address 者 將無法使用(一般的 NAT 實作會修改出現 在 FTP 和 ICMP 通訊協定資料中的 IP address,但其它協定就不一定了。通常得 分別為每個不同的通訊協定作特別的設 定。) z 以 IP address 作為安全檢查的方式將不 可行。 2.6.2 IPTABLES 在 linux 核心之中,負責處理網路封包的子系 統稱為 netfilter,而負責設定 netfilter 的命令則為 iptables。 舊版本的 netfilter 設定工具稱為 ipchains,在 ipchains 當中每一個 rules 都是一個 chain,而新版本 的 iptables 改以 tables 的方式去分門別類每一個 chain。 iptables 以篩表(table)的觀念來組織各種網路封 包的處理規則(rules),不同用途的規則被放在不同 的篩表,而規則是以目標(target)與篩選條件(match) 兩個部分構成,前者決定如何處置符合條件的封 包,後者決定如何挑選欲處理的封包。 2.7 NFS
NFS(Network File System) 是一種在網路上分 享檔案系統的方式,透過掛載(mount)的方式得以用 存取本機硬碟的方式去存取網路上的 NFS 伺服器。 在 linux 系統中,除了自己可以架設 NFS 伺服 器之外,也可透過安裝 NFS Client 軟體,掛載其他 由網路上 NFS 伺服器所匯出的檔案系統。 在叢集電腦系統中,利用 NFS 去建立共同的工 作區域是很有效的,所有節點透過 NFS 可以去分享 相同的程式、相同的輸出輸入檔等等,也方便管理 叢集電腦上每個節點。 2.8 NIS
NIS ,全名為 Network Information Service 。 原是 Sun Microsystem (R) 公司所研發的產品,一 開始被稱為 yp (Yellow Page),但因為 Yellow Page 這個名字和電信公司所用的電話簿「黃頁」的登記 名稱衝突,故 Sun 只好把它的正式名稱改成
Network Information Service,也就是 NIS。 事實上,NIS 就是設計來當成區域網路上的 「黃頁」使用的,專門提供網路上所有電腦應該要 知道的資訊,在電腦彼此間如果需要交換資訊,必 須要有一些兩端共同擁有的資訊,譬如對方主機的 名稱、具有權限的帳號和密碼、允許使用的資源等 等, 然後才能實際向對方電腦發出連結的要求(如 SSH)。這種服務被稱為目錄服務 (Directory Service)。 2.9 雙核心技術與 Hyper Threading 之介紹 2.9.1 雙核心技術 Intel 在 2005 年推出首顆採用雙核心技術的 Pentium D 處理器,將兩個 Pentium 4 的處理器核心 封裝在同一顆晶圓裡面,以提供更好的效能。但由 於兩個處理器核心直接封裝的耗電量與發熱量非 常之嚇人,介於 95~130W 之間,故在 Pentium D 之 後的雙核心處理器在設計階段就以兩組處理器單 元為設計原則,而非原來的單一基板直接嵌入二核 心設計。新版的 Core 2 Duo 處理器已降到 65W 左 右,耗電量與發熱量均得到顯著的改善。而由於雙 核心/多核心技術的普及,現在許多的應用程式開發 重點將置於如何善加利用多核心的優勢,如檔案壓 縮軟體等。 2.9.2 Hyper Threading 為雙核心技術的前身,中文名為超執行緒,其 原理為在單一處理器之中提供兩個邏輯處理單 元,形同有兩個邏輯處理器,在特定情況下可以提 供 5%~15%不同的效能增益。最初僅被 Xeon 處理 器所採用,後來的 Pentium 4 處理器也提供這樣的 功能。 而隨著 Core 架構的推出,HT 技術也漸趨沒落 了。不過由於本實驗中採用的是 Pentium 4 處理器, 故本實驗中的多核心是以 HT 技術為基礎的,和實 際的雙核心效能會有一段差距。但在實做上是沒有 差異的,未來若是有升級成雙核心甚至多核心的處 理器,程式碼也不需要改寫。
3. 實驗結果
3.1 實驗環境之規劃 大部分的情形下,校園的電腦教室大多被獨立 切割成一子網路,VLAN 中的電腦透過路由器連結 至 WAN,除了確保 VLAN 內部的順暢與單純,也 方便網管人員維護作業,如下圖所示:圖 3. 網路拓樸圖 本實驗中,我們採用主從式架構來建構我們的 叢集電腦環境,為了建構採主從式架構之獨立區域 網路以模擬一般校園電腦教室之實際應用,本實驗 以五台 PC 個人電腦組成一區域網路,包含一個主 節點以及四個子節點。每個節點都是相同的配備, 以確保平行運算的穩定性。唯一例外的是主節點將 會多配備一張網路介面卡,以提供 NAT 之功能。 而主節點除了分擔運算,還必須提供 NAT 伺服器、 SSH 遠端伺服器、NFS 伺服器、NIS 伺服器等功能, 以維持叢集電腦系統的運作。 表 1.設備一覽表
CPU Intel Pentium 4 3.2Ghz
RAM 512MB DDR-400
HDD Hitachi 80GB UATA-100
OS Debian Linux 3.1, with kernel v2.6
3.2 叢集電腦與單機之比較 首先我們必須先證明叢集電腦的效能比起單 機運算確實有著顯著的提升,在這裡我們利用大量 的 資 料 來 測試 單 機 系 統以 及 叢 集 電腦 系 統 的 差 異,結果如下圖所示: 表 2.單機與叢集電腦之效能比較表 (單位:秒,計 算時間越少代表效能越佳)
Comparison for Single PC and Cluster
資料量 8k 10k 12k 14k 16k 8 CPUs 4.107 4.927 5.66 7.573 8.549 2 CPUs 2.998 3.833 12.955 21.877 30.324 18k 20k 22k 24k 26k 8 CPUs 9.402 10.127 11.104 12.272 13.015 2 CPUs 40.494 52.381 66.036 79.566 91.936
圖 4.單機與叢集電腦之效能比較圖 (縱軸表時間 sec,橫軸表資料量;時間越短表效能越高) 根據圖表 2 跟圖 4,我們可以發現到叢集電腦 確實帶來驚人的效能,雖然在資料量不足的情形之 下效能並不能凸顯出來,甚至因為多節點平行處理 器間切換的損耗導致效能稍微降低,但一旦資料量 增加,則叢集電腦和單機系統的差異立刻顯現出 來,且資料量越大,速度上的差異越明顯。 3.3 SOMNN 在叢集環境下之應用 3.3.1 資料來源 生物的分類可讓我們了解生命的起源。直到現 在,很多研究皆進行處理這類的問題。而正如我們 所知道的生物學的有機體 DNA 序列的編碼的結構 經常被作為物種不同型態的討論及研究,因此透過 DNA 序列編碼的結構進行群聚分析將是一個值得 思考的方向。然而,多數的定量工具似乎並不適合 直接應用於 DNA 序列分析上,因此需要將 DNA 序 列進行某種形式上的轉換,以利引入相關的解析工 具;根據生物遺傳的中心法則,遺傳信息的傳遞是 由 DNA 到 mRNA,再由 mRNA 到蛋白質。遺傳信 息在由 mRNA 到蛋白質的傳遞過程中是以三連密 碼的形式來進行傳遞,每種氨基酸至少對應一個密 碼子(codon),最多的有 6 種對應的密碼子。編碼同 一種氨基酸的密碼子稱為同義密碼子,在蛋白質的 編碼過程中,同義密碼子的使用概率並不相同 (Ghosh,2000;Karlin & Mrazek,1996),某一物 種或某一基因通常傾向於使用一種或幾種特定的 同義密碼子,密碼子使用概率的問題是一個較為複 雜的現象,它的影響因素較多,並且各種影響因素 之間也會產生疊加或消除效應,從這些複雜的現象 中分析並發現內在規律,無論在理論上、還是在實 際應用中,都將具有重要的意義。近年來,人們發 現不同功能的基因其密碼子使用存在較大的差 異,但多數的研究主要還是在探討氨基酸和密碼子 的對應關係,較少探討如何有效地利用密碼子來進 行生物物種的分類議題(Sharp & Mosurski,1986; Mathe & Rouze,1999)。
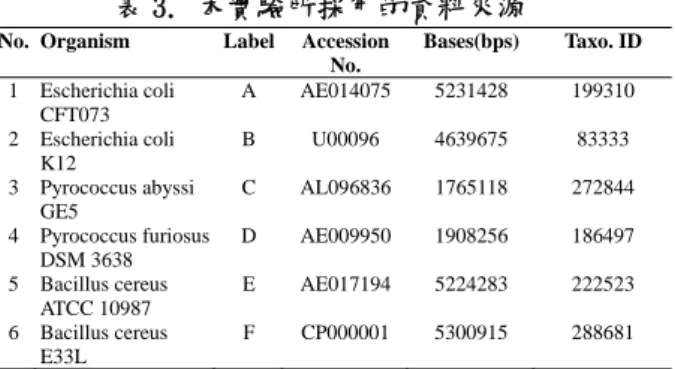
本 實 驗 中 , 我 們 採 用 了 六 隻 從 GONOME ALTAS DATABASE ( 網 址 http:// www.cbs.Dtu.dk/services/GenomeAtlas/)取得的細菌 DNA 來進行分群,在進行程序前,首先必須先針對 物種 DNA 進行資料的前處理程序,而這些前處理 程序包含了 DNA 序列的轉換。在概念邏輯上,因 為 DNA 約為 20 種氨基酸所組成,每種氨基酸與密 碼子的對應方式不盡相同(有些是三連密碼形式、 有些則是六連形式),為考量能提供較多的潛藏資 訊,因此我們考量有較多密碼型式的氨基酸,故選 定了 LEU、SER、ARG 等三種,因為這三種氨基酸 均為六連密碼形式(參考文獻)。再取得每一個密 碼子在編碼區中出現次數的資料,即可換算每一個 密碼子在同義密碼子中出現的比例,換言之,即可 求得所謂的 codon usage。 表 3. 本實驗所採用的資料來源
No. Organism Label Accession No. Bases(bps) Taxo. ID 1 Escherichia coli CFT073 A AE014075 5231428 199310 2 Escherichia coli K12 B U00096 4639675 83333 3 Pyrococcus abyssi GE5 C AL096836 1765118 272844 4 Pyrococcus furiosus DSM 3638 D AE009950 1908256 186497 5 Bacillus cereus ATCC 10987 E AE017194 5224283 222523 6 Bacillus cereus E33L F CP000001 5300915 288681 3.3.2 基於 MPI 之演算程序 我們引入 MPI 函式庫,其中許多的平行演算程 序在 MPI 中都已經有內建,可以大大的減少我們開 發程序的時程。且 MPI 在學界應用已久,成熟度跟 完整度自然是相當的高。由下圖我們可以發現到基 於 MPI 函式庫的叢集電腦效能遠勝於基於 NFS 以 及 thread 架構的叢集電腦。
表 4. MPI Cluster 與非 MPI Cluster 之叢集運算 效能比較
Method Operations time
Single PC 512 dims * 1024 data 45.287sec clusters based on NFS/Thread 512 dims * 1024 data 12.620sec clusters based on MPI 512 dims * 1024 data 2.231sec int main(){ MPI_Init(); MPI_Comm_rank(); MPI_Comm_size();
if (masterNode){
seperateData(&inputFileContent, &buffer); for(nodeID = 0; nodeID < totalNode; nodeID++)
MPI_Send(&buffer[nodeID], nodeID); }
//receive data from master PC
MPI_Recv(&subNodeData, masterNodeID);
//Start SOM
subResult = SOMLearn( &subNodeData ); subGroupCenterPoint = SOMgCP( &subNodeData );
//Return result of each node and the center point data
MPI_Send(&subResult, masterNodeID); MPI_Send(&subGroupCenterPoint, masterNodeID);
for(nodeID = 0; nodeID < totalNode; nodeID++){ MPI_Recv(&collectedData[nodeID], nodeID); MPI_Recv(&eachNodeGroupCenterPoint[nodeID] , nodeID);
}
//Perform SOMNN by using the center point of cluster
eachGroupBelonging =
SOMLearn(eachNodeGroupCenterPoint);
//deriving the result depending on the results //from the SOMNN procedure
// and the result of each node
summary = makeResult(eachGroupBelonging, collectedData); }
圖 4. 基於 MPI 架構之演算程序 圖 5. 基於 NFS/Thread 之演算架構圖 3.3.3 應用結果 最後,我們在基於 MPI 函式庫的架構之下,來 檢驗在不同的網路環境中的演算結果。根據已知的 資料群數跟演算出來的資料群數加以比對,以驗證 其正確性。 表 5. 正確性驗證比較表 Tuned LAN 原資料群數/分群後 3/3 由上圖可知,結果符合我們的預期,證明我們 所設計的演算程序是可行的。 4.結論 我們所設計的應用叢集式電腦環境來達成群 聚分析的詳細演算程序在實驗中已經驗證其可行 性,而很明顯的,叢集電腦環境對於網路環境要求 較高,雜訊不僅影響效能,連帶使得資料正確率降 低,大大降低了叢集電腦系統的實用性。 然而,在一般電腦教室環境中難以保證網路環 境能夠清淨無瑕,故我們認為,在 MPI 程式的演算 法中增加一些增進資料正確率的演算法 (如 CRC 演算法) 應該能增進系統的正確率,而這是我們未 來努力的方向。 最後,我們已經證明了一般校園電腦環境下叢 集電腦系統的發展可行性,未來若有類似資料量龐 大且費時研究之如財務分析、趨勢分析、影像處理 等,在資源有限亟需降低成本之考量下,本案例實 為一有效的解決方案。
參考文獻
[1] C.-C. Jeng, I.-C. Yang, K.-L. Hsieh, J.-N. Lin, (2005), “Bacteria Classification on Power Spectrums of Complete DNA Sequences by Self-Organizing Map”, Neural Information Processing – Letter and Review, 2005
[2] T.Kohonen,, “Self-organized formation of topologically correct feature maps,” Biological Cybernetics, Vol. 66, pp 59-69, 1982.
[3] T.Kohonen, “Self-organization and associate memory”, Springer-Verlag London,1984.
[4] J. Vesanto, E. Alhoniemi, “Clustering of the Self-Organizing Map”, IEEE TRANSACTIONS ON NEURAL NETWORKS, VOL. 11, NO.3, May 2000 [5] J.-S. R. Jang, C.-T. Sun, E. Mizutani,
“Neuro-Fuzzy and Soft Computing, A Computational Approach to Learning and Machine Intelligence.” Prentice-Hall 1997 pp.306~307 [6] T. L. Sterling, J. Salmon, D. J. Becker, and D. F.
Savarese, “How to Build a Beowulf-A Guide to the Implementation and Application of PC Clusters”, Cambridge, Ma, The MIT Press, 1999. [7] J. Alspector and D. Lippe, “A study of parallel
weight perturbative Gradient Descent,” In Proc. of Advances in Neural Information Processing System (NIPS ’96), pp. 803-810, Cambridge, Ma.: The MIT Press, 1996.
[8] J. D. Sloan, “High Performance Clusters with OSCAR”, Rocks, OpenMosix, and MPI, CA: O`Reilly, 2004.
[9] Mathematics and Computer Science Division,“MPICH-A Portable Implementation of
MPI”, Available from: HTTP://www-unix.mcs.anl.gov/mpi/mpich/, 2003.
[10] T. Bermmel, “MP-MPICH: Multi-Platform
MPICH”, Available from: HTTP://www.lfbs.rwth-aachen.de/mpmpich/, 2003. [11] 蔡德明,「簡易的 Cluster 架設」,鳥哥的 Linux 私房菜,http://linux.vbird.org,(2005/12/20),2003 年。 [12] 吳佳諺,「Linux 叢集伺服器」,吳佳諺工作室, http://ftp.ncnu.edu.tw/Documentation/Linux/RedH at_Fedora_set_site/ch21.pdf (2006/2/28),2003 [13] 吳旭智、賴淑貞譯, 「資料採礦理論與實務-顧客關係管理的技巧與科學」,台北:維科圖書 有限公司,2001 年。 [14] 葉怡成,「類神經網路模式應用與實作」,台 北:儒林圖書有限公司,2003 年。 [15] 鄭士豪、林英超、蕭景鴻,UNIX 系統管理手 冊 「 UNIX System Administration Handbook 3/e」,台北:培生教育出版股份有限公司,2002 年。
[16] 簡祥全(2002) 知識經濟國家時間群聚分析, 朝陽科技大學資訊管理研究所碩士論文,台中 縣。.
[17] Berks G, DG Keyserlingk, J Jantzen, M Dotoli, H Axer (2000) Fuzzy Clustering- A Versatile Mean to Explore Medical Database. Aachen, Germany, 450pp.
[18] Bezdek JC (1981) Pattern Recognition with Fuzzy Objective Function Algorithms. Plenum Press, New York, 582pp.
[19] Chen YH, SL Nyeo, JP Yu (2005) Power-laws in the complete sequences of human genome. Journal of Biological Systems, 13:105-115.
[20] De Sousa Vieira M (1999) Statistics of DNA sequences: A low-frequency analysis. Physical Review E, 60: 5932-5937.
[21] Doolittle RF, DF Feng, S Tsang, G Cho, E Little (1996) Determining divergence times of the major Kingdoms of living organisms with a protein clock. Science, 271:470-477.
[22] Dunn JC (1973) A Fuzzy Relative of the ISODATA Process and Its Use in Detecting Compact Well-Separated Clusters. Journal of
Cybernetics, 3: 32-57.
[23] Herzel H, I Grosse (1995) Measuring correlations in symbolic sequences. Physica A, 216: 518-542.
[24] Hsu TH (1999) An Application of Fuzzy Clustering in Group-Positioning Analysis. Department of Business Administration, I-Shou University, 122pp.
[25] Isohata Y, M Hayashi (2005) Analyses of DNA base sequences for eukaryotes in terms of power spectrum method. Japanese Journal of Applied Physics, 44: 1143-1146.
[26] Jang JS, CT Sun, E Mizutani (1997) Neuro-Fuzzy and Soft Computing. Prentice Hall, New York
[27] Nyeo SL, IC Yang, CH Wu (2002) Spectral Classification of Archaeal and Bacterial Genomes. Journal of Biological Systems, 10: 233-241. [28] Odile L, R Raymond, PB Valerie, D Simone, H
Roland, D Jacques, JC Thierry, P Olivier (2001) Genome Evolution at the Genus Level: Comparison of Three Complete Genomes of Hyperthermophilic Archaea. Genome Research, 11: 981–993.
[29] TIGR, the Institute for Genomic Research (2005) Genome pages. http://www.tigr.org/ tigr-scripts/ CMR2/GenomePage3.spl?database=ntec02. [30] Voss RF (1992) Evolution of long-range fractal
correlations and 1/f noise in DNA base sequences. Physical Review Letter, 68: 3805-3808.
[31] Zimmermann JJ (1991) Fuzzy Set Theory and Its Applications. Kluwer Academi, Boston.
[32] Ghosh T. (2000) Studies on codon usage in entamoeba histolytica. International Journal of Parasitology, 30 :715-722
[33] Karlin S , Mrazek J . (1996) What drives condon choices in human genes Journal of Molecular Biology, 262 :459-472
[34] Sharp P , Mosurski K. (1986) Codon usage in yeast :cluster analysis clearly differentiates highly and lowly expressed genes. Nucleic Acids Re2 search, 14 :5125-5143
[35] Mathe C , Rouze P. (1999) Classification of arbidopsis thaliana gene sequences : clustering of coding sequences into two groups according to codon usage improves gene prediction. Journal of Molecular Biology, 285 :1977-1991
[36] The Institute for Genomic Research (TIGR).
(2005) Genome pages. http://www.tigr.org/tigr-scripts/CMR2/GenomePag
e3.spl?database=ntec02.
[37] E. Helgason, O. A. Økstad, D. A. Caugant, H. A. Johansen, A. Fouet, M. Mock, I. Hegna, and A.-B. Kolstø. (2000) Bacillus anthracis, Bacillus cereus, and Bacillus thuringiensis--One Species on the Basis of Genetic Evidence. Applied and
Environmental Microbiology, 66:2627-2630. [38] Intel Official site. www.intel.com, California,