限制型利基柏拉圖基因演算法及其於製程參數最佳化之應用
全文
(2) 限制型利基柏拉圖基因演算法及其於製程參數最佳化之應用. 學生:彭加景. 指導教授:蘇朝墩 教授 沙永傑 教授. 國立交通大學工業工程與管理學系碩士班. 摘要 特徵選取的目的在於將不重要的特徵消除,保留有用的特徵,使分析過程更為迅 速,分析結果更為可靠。利基柏拉圖基因演算法是一種針對多目標最佳化問題所發展出 來的工具,可找到多個目標之間的折衷最佳解,以供分析人員選擇,而這些折衷最佳解 所形成之理想曲線,稱為柏拉圖最佳邊際。當此方法使用於分類問題之特徵選取上時, 會發生在挑選過程中,基因演算法以減少使用特徵數為優先的狀況。此將導致最後所找 到之柏拉圖最佳邊際發生特徵數刪減過多,且分類準確率沒有明顯提升的狀況,此問題 在原始的總特徵數越多時,會越加明顯。由於在實際問題中,提升分類準確率,常常會 比大幅刪減使用特徵數來得重要,本研究提出限制型利基柏拉圖基因演算法,透過在基 因演算法的挑選過程中,增加一準確率限制的手段,調整基因演算法之搜尋方向,以避 免特徵的過度刪減,並確保分類準確率的提升。 本研究使用了三筆數值資料,比較原始的利基柏拉圖基因演算法與限制型利基柏拉 圖基因演算法之差別。結果顯示限制型利基柏拉圖基因演算法的確能找到分類準確率較 高的特徵組合,且不會發生特徵數刪減過多的問題。本研究亦將此方法應用於一 DVD 光碟片之製程問題上,找出製程中的重要參數;接著利用這些製程中的重要參數,建立 製程關係模型,再針對此模型進行製程參數最佳化,達到提升品質、降低不良率的目的。 關鍵詞:特徵選取、利基柏拉圖基因演算法、柏拉圖最佳邊際. I.
(3) Constraint Based Niched Pareto Genetic Algorithm and Its Application in Process Parameter Optimization Student: Chia-Ching Peng. Advisor: Dr. Chao-Ton Su Dr. David Yung-Jye Sha. Department of Industrial Engineering and Management National Chiao Tung University ABSTRACT Feature selection is an important step to deal with classification problem. The essential effect of feature selection is to improve the accuracy and speed of classification systems. Niched Pareto Genetic Algorithm (NPGA) is a powerful method to solve optimization problems with multiple objectives. It can yield a diverse population of solutions among the multiple, conflicting objectives. As NPGA is applied to feature selection in the classification problem, it will show a preference to reduce the feature used, but not to improve the classification accuracy. This will cause the final Pareto optimal frontier perform feature subsets with many features erasing and less improvement of classification rate. In order to improve the accuracy of classification, it is necessary to avoid the over trimming of the features. In this study, we propose a constraint based niched Pareto genetic algorithm to adjust the thread of search. Three numerical examples are employed to demonstrate the difference between original and constraint based niched Pareto genetic algorithm in this study. We discuss the constraint can avoid genetic algorithm from erasing too many features and improving the classification accuracy. The proposed method is also applied to find key factors in a Digital Video Disk manufacturing system. The key factors will be used to help modeling and optimizing the manufacturing process parameters. Keywords: Feature selection, Niched Pareto Genetic Algorithm, Pareto optimal frontier. II.
(4) 誌. 謝. 本論文能夠順利完成,需要感謝許多人。首先要感謝指導教授蘇朝墩博士兩年來的 悉心指導。不論在研究方向的決定、研究方法的訂正、實驗的進行及論文的撰寫上,均 給予我很大的幫助,在此謹向蘇教授表達最深的謝意。感謝系上沙永傑教授的苦心叮嚀 及幫助,使得本論文可以順利完成。感謝元智大學鄭春生教授及明新科大姜台林副教授 等口試委員的寶貴意見,使得論文的若干疏失,得以框正。再者,感謝萊德科技於實驗 資料及驗證上的鼎力相助。感謝許志華、許俊欽、陳隆昇、楊健炘、林敬森及周家任等 學長的鼓勵及指導。感謝宇翔與系上同學,讓我過了兩年愉快的碩士生活。. 最後,我要感謝我的雙親及姐姐,他們的照顧及支持,讓我能夠專心於學業上,沒 有後顧之憂。感謝在這段時間,所有關心及幫忙我的人。. III.
(5) 目 中文摘要 英文摘要 誌謝 目錄 表目錄 圖目錄 第一章 1.1 1.2 1.3 第二章 2.1 2.2 第三章 3.1 3.1.1 3.1.2 3.1.3 3.1.4 3.1.5 3.2 第四章 4.1 4.1.1 4.1.2 4.2 4.2.1 4.2.2 4.2.3 第五章 5.1 5.2 5.2.1 5.2.2 5.3 5.4. 錄. ……………………………………………………………… ……………………………………………………………… ……………………………………………………………… ……………………………………………………………… ……………………………………………………………… ……………………………………………………………… 導論………………………………………………………… 背景與動機………………………………………………… 研究目的…………………………………………………… 研究架構…………………………………………………… 相關研究…………………………………………………… 基因演算法………………………………………………… 基因演算法於特徵選取上的應用………………………… 研究方法…………………………………………………… 限制型利基柏拉圖基因演算法…………………………… 染色體編碼………………………………………………… 分類準確率之計算………………………………………… 染色體挑選方法…………………………………………… 交配及突變………………………………………………… 分類準確率限制之決定…………………………………… 混合類神經網路及基因演算法之參數最佳化方法……… 實驗與結果比較…………………………………………… 實驗方法說明……………………………………………… 實驗資料…………………………………………………… 實驗進行…………………………………………………… 實驗結果…………………………………………………… 肝病診斷…………………………………………………… 威斯康辛乳癌……………………………………………… 聲納金屬探測……………………………………………… DVD製程參數之最佳化………………………………… 背景簡介…………………………………………………… 製程重要製程參數之選擇………………………………… 循軌誤差之特徵選取……………………………………… 聚焦誤差之特徵選取……………………………………… 類神經網路模型之構建…………………………………… 重要製程參數之最佳化及評估…………………………… IV. I II III IV VI VII 1 1 3 4 5 5 7 11 11 11 12 12 14 14 16 17 17 17 18 21 21 26 32 39 39 40 40 42 43 44.
(6) 第六章 參考文獻. 結論………………………………………………………… ………………………………………………………………. V. 47 48.
(7) 表目錄 表 4.1 表 4.2 表 4.3 表 5.1 表 5.2 表 5.3 表 5.4 表 5.5 表 5.6. 實驗一參數設定…………………………………………… 實驗二參數設定…………………………………………… 實驗三參數設定…………………………………………… 限制型利基柏拉圖基因演算法之參數設定……………… 循軌誤差之特徵選取結果………………………………… 聚焦誤差之特徵選取結果………………………………… 學習率與動量之設定……………………………………… 利基柏拉圖基因演算法之參數設定……………………… 進行實機測試之最佳設定…………………………………. VI. 19 20 21 41 41 42 43 44 46.
(8) 圖目錄 圖 1.1 圖 1.2 圖 2.1 圖 3.1 圖 3.2 圖 3.3 圖 4.1 圖 4.2 圖 4.3 圖 4.4 圖 4.5 圖 4.6 圖 4.7 圖 4.8 圖 4.9 圖 4.10 圖 4.11 圖 4.12 圖 4.13 圖 4.14 圖 4.15 圖 4.16 圖 4.17 圖 4.18 圖 4.19 圖 4.20 圖 4.21 圖 4.22 圖 4.23 圖 4.24 圖 4.25 圖 4.26 圖 4.27 圖 4.28 圖 4.29 圖 4.30. 特徵選取方法……………………………………………… 使用染色體多寡與準確率限制對柏拉圖邊際之影響…… 基因演算法流程圖………………………………………… 特徵選取基本流程………………………………………… 染色體編碼方式…………………………………………… 限制型利基柏拉圖基因演算法…………………………… 前 2 0 代趨勢圖(肝病)…………………………………… 前 5 0 代趨勢圖(肝病)…………………………………… 前 100 代趨勢圖(肝病)…………………………………… 特徵使用率變異趨勢圖(肝病)…………………………… 分類準確率率變異趨勢圖(肝病)………………………… 5 0 代之最佳解(肝病 實驗二)………………………… 100 代之最佳解(肝病 實驗二)………………………… 200 代之最佳解(肝病 實驗二)………………………… 5 0 代之最佳解(肝病 實驗三)………………………… 100 代之最佳解(肝病 實驗三)………………………… 200 代之最佳解(肝病 實驗三)………………………… 前 5 0 代趨勢圖(乳癌)…………………………………… 前 100 代趨勢圖(乳癌)…………………………………… 前 200 代趨勢圖(乳癌)…………………………………… 特徵使用率變異紀錄圖(乳癌)…………………………… 分類準確率率變異紀錄圖(乳癌)………………………… 5 0 代之最佳解(乳癌 實驗二)………………………… 100 代之最佳解(乳癌 實驗二)………………………… 200 代之最佳解(乳癌 實驗二)………………………… 5 0 代之最佳解(乳癌 實驗三)………………………… 100 代之最佳解(乳癌 實驗三)………………………… 200 代之最佳解(乳癌 實驗三)………………………… 前 5 0 代趨勢圖(聲納)…………………………………… 前 100 代趨勢圖(聲納)…………………………………… 前 200 代趨勢圖(聲納)…………………………………… 前 300 代趨勢圖(聲納)…………………………………… 特徵使用率變異紀錄圖(聲納)…………………………… 分類準確率率變異紀錄圖(聲納)………………………… 100 代之最佳解(聲納 實驗二)………………………… 200 代之最佳解(聲納 實驗二)………………………… VII. 1 4 6 11 12 15 21 22 22 22 23 24 24 24 25 26 26 27 27 27 28 28 29 29 30 31 31 31 32 33 33 33 34 34 35 35.
(9) 圖 4.31 圖 4.32 圖 4.33 圖 4.34 圖 5.1 圖 5.2. 300 代之最佳解(聲納 實驗二)………………………… 100 代之最佳解(聲納 實驗三)………………………… 200 代之最佳解(聲納 實驗三)………………………… 300 代之最佳解(聲納 實驗三)………………………… 利基柏拉圖基因演算法之結果…………………………… 最佳製程參數設定預測值…………………………………. VIII. 36 37 37 37 45 45.
(10) 第一章 導論 1.1 背景與動機 分類問題的目的是根據已知類別的資料之屬性或特徵,訓練出分類的規則或模型, 然後透過建立好的規則或模型,對未知類別的資料進行判別類別的工作。在分類問題 中,會影響分類準確率的主要三項因素是:訓練模型的資料樣本、資料輸入的屬性及分 類器。當訓練模型的樣本包含離群值或錯誤值時,將影響分類器的預測效果,但這通常 是無法避免的。至於分類器方面,不同的分類器適用的資料類型各不相同,很難找到一 種分類器能在各種資料類型下均有最好的表現。對於資料輸入屬性而言,當輸入屬性愈 多,除了會增加分類過程的計算複雜度外,並不一定保證會有更佳的分類準確率,甚至 可能會使分類器的表現更差。如何選取良好的資料輸入屬性,使分類器具有良好的分類 績效,即稱之為特徵選取(feature selection)問題。圖 1.1 為常見的特徵選取方法[7]。 特徵選取. 統計辨識方法. 區域最佳解. 單一解. 類神經網路 (節點懲罰) 最佳解 (窮舉法) (截枝法). 非單一解. 具再現性 無再現性 具再現性 無再現性 (累加搜尋法)(模擬退火法)(光柱搜尋法)(基因演算法) (累減搜尋法) (流動搜尋法) 圖 1.1 特徵選取方法[1]. 1.
(11) 在特徵選取問題上,主要分成兩種方向,第一種是找出錯誤率最低的特徵組合,主 要目標在於降低分類錯誤率;第二種是找出在可容忍的錯誤率內,特徵數最少的特徵組 合,主要目標在於降低使用特徵數,增進運算效率。當特徵選取問題具有「選取的特徵 數越少,分類準確率越低」的性質時,此種性質即稱之為單調性(monotonic),而此種特 性在一些線性迴歸分類器上,是較常見到的。當特徵選取問題具有單調性時,截枝法 (branch and bound)是一種有效的特徵選取方法[11]。但截枝法在實施上有兩個主要的缺 點,第一是當問題不具備單調性時,可能會將最佳解修剪掉。第二是因截枝法必須對所 有未修剪的區域做全域搜尋,所以當特徵數增多時,截枝法依然要花費很多的時間去搜 尋最佳解,尤其是當特徵數大於二十時,截枝法將變成難以實行。有鑒於此,Siedlecki 與 Sklansky[11]將基因演算法應用於特徵選取上,以解決非單調性或大型的特徵選取問 題。. 基因演算法是一種有效的最佳解搜尋方法,於六零年代由 John Holland 所提出,因 其擁有從區域性最佳解跳脫,並快速找到近似最佳解,甚至是全域最佳解的可能性,所 以被廣泛的應用於各種科學領域上。基因演算法最早於 1989 年,被 Siedlecki 及 Sklansky 使用於特徵選取後,直到最近,基因演算法於特徵選取之應用的相關研究,依然持續著。 Kuncheva[8]在 1999 年將基因演算法應用於樣本及輸入屬性的選取上,以提高最近距離 分類器的運算速度。Chen[3]於 2003 年發表 GKMT(GA based k-means-type algorithm)演 算法,處理樣本選取及輸入屬性之權重調整的問題。. 當使用基因演算法進行特徵選取時,通常利用染色體(chromosome)表示可能的特徵 組合,而如何評定染色體,亦即各種特徵組合之間的優劣,是攸關特徵選取成敗的關鍵 步驟之一。一般考慮的因素包括特徵組合之分類準確率及所使用之特徵數兩者,但此兩 者在某種程度上經常是相衝突的,當使用越少之特徵進行分類,通常分類準確率亦會隨 之下降,要如何在兩者之間取得平衡,是一個難題。Emmanouilidis[4]等人應用 Horn[5] 所提出之利基柏拉圖基因演算法(Niched Pareto GA)於特徵組合之評選上,透過搜尋柏拉 2.
(12) 圖最佳邊際(pareto optimal frontier),找出多種不同的準確率與使用特徵數之間的折衷方 案。. 使用基因演算法搜尋各種使用特徵數與分類準確率之間的折衷解時,染色體會均勻 散佈到柏拉圖邊際上,某些無法互相取代的不同聚落裡,而實驗過程中所設定的聚落半 徑(σs),將決定這些聚落的影響範圍及聚落個數。當原始資料所考慮的總特徵數非常龐 大時,如果設定的聚落半徑較小,可能導致所找到之柏拉圖邊際不完整,偏向某一方向; 此狀況常常會使基因演算法的搜尋方向偏向減少特徵數的方向,導致因特徵數刪減過 多,而使得分類準確率降低。但若加大聚落半徑,可能會使染色體收斂到少數幾個點上, 導致很多折衷解無法顯現。針對此一問題,增加染色體數是一項解決辦法,但隨著使用 的染色體數增多,將使基因演算法之運算時間,也隨之大幅的增加。如何使用適當的染 色體數,搜尋到符合使用者需求的柏拉圖最佳邊際,是一值得研究的議題。. 1.2 研究目的 圖 1.2 顯示使用之染色體多寡,與所搜尋到的柏拉圖邊際之關係。為了解決當需考 慮的特徵項目過多,需要大量的染色體去搜尋柏拉圖邊際的問題。本研究透過設定分類 準確率限制的方法,以限制基因演算法的搜尋方向,使基因演算法能針對使用者感興趣 的方向搜尋,希望能以較少的染色體數,依然能求得分類準確率較高之特徵組合。. 本研究首先採用三筆 UCI 資料庫的資料,說明不同的準確率限制將如何影響基因演 算法所搜尋到之柏拉圖最佳邊際。在各種不同的準確率限制條件下,基因演算法隨著世 代交替數的增加,透過所搜尋到之染色體族群重心的移動趨勢,我們將比較在各種不同 世代交替數下,於各種不同使用特徵數所能找到之最佳分類準確率。此外,本研究所提 之方法將被應用到一個製程參數最佳化之實際問題。本研究以所提之限制型利基柏拉圖 演算法來找出製程參數中之重要因子,並利用類神經網路,建構這些重要因子與產品品. 3.
(13) 質之間的模型,最後再利用基因演算法針對此模型找出製程之最佳參數設定。 分 類 準 確 率. 使 用 染 色 數 多. 柏拉圖邊際. 特徵使用率. 使 用 染 色 數 少. 分 類 準 確 率. 分 類 準 確 率. 特徵使用率. 分類準確率限制. 特徵使用率. 圖 1.2 使用染色體多寡與準確率限制對柏拉圖邊際之影響. 1.3 研究架構 本篇論文餘後的部分,將於第二章介紹前人所提出的以基因演算法為基礎之特徵選 取相關方法。第三章為本研究所提之方法,此方法之有效性將以測試資料比較於第四 章。第五章為實例應用,第六章則為結論。. 4.
(14) 第二章 相關研究 2.1 基因演算法 基因演算法(Genetic Algorithm)是一種有效的最佳解搜尋方法,於六零年代由 John Holland 所提出,用以處理較複雜、非線性時間可解決之問題。在原始的基因演算法中, 解空間的所有可能解將被編碼成以 0 和 1 組成的字串,而一個解所代表的字串則稱之為 一條染色體(chromosome)。GA 搜尋最佳解的方法是先依據編碼規則隨機產生一組包含 大量染色體的初始解集合,稱之為親代集合(parent set),然後針對此集合中的所有染色 體一一評分,分數高低由設計者所設定之適應函數(fitness function)所決定,染色體的分 數越高,表示該染色體所代表的解,越符合設計者的要求。染色體將依據分數高低,決 定其獲選產生子代的機會。常見的染色體挑選方法有適應分數比例選取法 (fitness-proportionate selection)、順位選取法(rank selection)及競爭式選取法(tournament selection)。被選中產生子代的染色體可透過與其他染色體進行交配(crossover)以及突變 (mutation)兩種操作方法,將本身好的基因傳承下去。. 交配與突變是基因演算法最重要的操作元之二。所謂的交配,代表染色體間資訊的 交換,藉由此一步驟,不同染色體之間的優秀基因塊(building block),將有機會被組合, 以產生更好的染色體。染色體之交配機率,一般設定在 0.75 至 0.95 之間。所謂的突變, 可幫助基因演算法從區域性最佳解裡跳脫,求得更佳的解。染色體之突變機率,一般設 定在 0.01 至 0.005 之間。而這些由親代染色體透過交配、突變所產生的子代染色體將成 為下一世代的新親代染色體,繼續進行生存的競爭。透過一個世代一個世代的競爭與淘 汰,染色體將愈來愈符合設計者的要求,此即為基因演算法搜尋最佳解的方法。圖 2.1 為基因演算法之操作流程圖。. 5.
(15) 產生第一代的親代染色體(可能解), P={a1,a2,…,an}. 對於親代集合 P 中的染色體,根據適應函數,一一進行評分。. 從 P 中依據染色體的分數高低,挑選染色體進入交配池 中,直到交配池中的染色體數與親代染色體數相同。. 從交配池中挑選兩條染色體,根據交配率pc,決定是否交配, 並產生子代。. 根據突變機率pm,決定子代染色體是否進行突變. 重複交配與突變的動作,直到子代染色體總數與親代 染色體數 n 相同。. 將子代染色體集合轉成新的親代集合 P. 否. 滿足 條件 是. 實驗解答 (區域最佳解或全域最佳解). 圖 2.1 基因演算法流程圖. 6.
(16) 2.2 基因演算法於特徵選取上的應用 2.2.1 染色體編碼方式 當基因演算法應用於特徵選取上,每一條染色體將代表一種特徵選取的子集合,染 色體第 k 位置的數字代表相對應位置的第 k 項特徵是否被選取到,如果是 1 代表選取, 若為 0 則代表不選取[11]。Pei 等人[10]進一步考慮資料轉換,讓染色體的編碼不再是單 純的選取或不選取。在線性轉換的部分: Y = WX. (1). ⎡ w1 ⎢0 W =⎢ ⎢. ⎢ ⎣0. 0 w2 . 0. 0⎤ ... 0 ⎥⎥ ... . ⎥ ⎥ ... wn ⎦ .... (2). X = [x1 , x 2 ,..., x n ]. (3). X 代表原本的 n 項資料特徵,W 為轉換矩陣,此舉使得原本資料的每一項特徵有權重的 變化,Y 則為轉換後之新的 n 項資料特徵。此處,每一條染色體代表一種轉換矩陣 W, GA 於此的目的在找出每一項特徵的適當權重。在非線性轉換的部分: Y = WX ,. (4). ⎡ w1 ⎢0 ⎢ ⎢. ⎢ W =⎢ . ⎢. ⎢ ⎢. ⎢0 ⎣. [. 0 w2. .. .. .. .. .. .. .. .. .. .. .. .. .. .. . wn. .. .. . .. . .. . .. 0. .. .. wn+1 . . . .. .. 0 ⎤ 0 ⎥⎥ . ⎥ ⎥ . ⎥ . ⎥ ⎥ 0 ⎥ wn+ k ⎥⎦. (5). ]. (6). X = x1 , x 2 ,..., x n , ( xi x j )1 ,..., ( x r x s ) k. 非線性轉換與線性轉換的不同點在於,所考慮的 X 不僅包括原本的 n 項資料特徵, 還包含了 k 項使用者有興趣的特徵交互作用項。使得轉換矩陣 W 更為複雜。在原始論文 中,此方法被應用於增進 KNN 分類器之分類準確率。實驗結果說明透過基因演算法找. 7.
(17) 出適當的轉換矩陣 W,將可降低 KNN 分類器的分類錯誤率,而非線性轉換的效果會比 線性轉換的效果更好。. 2.2.2 染色體評分及選取 利用基因演算法進行特徵選取時,要如何評量一組特徵選取組合的好壞,通常包括 兩種考量因素,第一種是分類錯誤率越低越好,第二種是使用的特徵數越少越好。如果 能找到同時使這兩種條件皆為最佳的特徵組合,當然最為理想。但這兩者通常是互相衝 突的,當特徵數降低到某個層度時,常常伴隨著分類錯誤率的升高。如何在分類錯誤率 及特徵數之間進行協調,實在難以決定。. Siedlecki 與 Sklansky[11]在評分方法上,設計了一種懲罰函數,以解決此問題,懲 罰函數如下:. p (e) =. exp((e − t ) / m) − 1 exp(1) − 1. (7). 上式中,e 代表分類錯誤率、t 代表使用者自訂的可行性門檻(feasibility threshold)、m 代 表容忍界線(tolerance margin)。當 e > t 時,懲罰函數將為正值,當 e < t 時,懲罰函數將 為負值,代表當分類錯誤率高於門檻值時,將會受到懲罰,且差距越多,懲罰越重。反 之,當錯誤率低於門檻值時,染色體不但不會受到懲罰,反而會受到獎勵,至於懲罰及 獎勵的程度,將由容忍界限 m 決定。將此懲罰函數加進評分方法中,每條染色體將各自 有一個分數:. J (a) = l (a ) + p(e(a)). (8). 上式中的 a = {α 1 ,...,α d } 代表著某一條染色體的字串, α i = 1 代表第 i 位置的特徵需選 取,α i = 0 則代表第 i 位置的特徵不選取。e(a)為染色體 a 所代表之特徵組合的分類錯誤 d. 率。 l (a) = ∑ α i 代表著此染色體選取到的總特徵數。根據此式,分類錯誤率愈高及特徵 i =1. 數愈多,將獲得愈高的分數。針對此分數,基因演算法的適應函數設定如下: f (ai ) = (1 + ε ) max J (a j ) − J (a i ) a j∈p. 8. (9).
(18) 上式中, ε 為一常數,以確保每條染色體的選取的可能性均能大於零。. 除了使用懲罰函數外,亦可利用權重的方式對特徵使用個數及分類錯誤率做綜合評 價。如 Bhanu 及 Lin[2]所設計的 MDLP(minimum description length principle)方法中,其 適應函數設定為:. F (ci ) = −(k log( f ) + ne log(n)). (10). 式(10)中 f 代表總特徵數,k 代表染色體 ci 中,字元為 1 的數目,亦即被選取到的特徵數。. n 代表訓練資料的總筆數, ne 則代表訓練資料中,分類錯誤的筆數。透過此式,特徵使 用個數及分類錯誤率的相對重要性,將由總特徵數及總樣本數決定。. Horn[5]等人,針對此等多目標最佳化的問題,提出了使用柏拉圖選取(pareto selection)配合 niched-GA 的方法,以求得各種不同目標值之間的最佳折衷方案。柏拉圖 選取方法在判斷兩筆資料間的優劣時,除非其中一筆資料在所有考量的目標上皆優於另 一筆資料,否則兩者將被視為同樣的好。當一筆資料並不劣於任何一筆其他資料時,即 稱此筆資料為不可取代(non-dominated)的解。基因演算法於此的目的即是試圖找到所有 不可被取代的最佳解集合,此集合又稱為柏拉圖最佳邊際(pareto optimal frontier)。為了 避免基因演算法的染色體收斂到單一個不可取代解上,Horn[5]加入了適應分數共享. (fitness-sharing)的概念,所謂的適應分數共享的原始做法是,對於所有的染色體 i,算出 在染色體族群中與其相似的染色體當量數(niche count) mi ,再將其適應分數除以 mi ,成 為該染色體的真正得分。透過這種手段,可避免所有染色體收斂到某一個最佳解上,以 確保染色體之間的多樣性。但 Horn 於此不計算適應分數,直接以相似染色體當量數決 定應選取的染色體。. 2.2.3 分類錯誤率計算方法 當要計算某一筆屬性組合的分類錯誤率時,毫無疑問需要某種分類規則或分類器的 幫助。雖然基因演算法亦可自行發展出一套分類方法,例如 Bandyopadhyay 等人[1]透過 9.
(19) 多條直線,將樣本所處的多維空間分割成許多小區域,測試樣本將依據落於每條直線的 正端或副端,而被分類到某一個小區域中。基因演算法被其使用於尋找能使分類錯誤率 最低的最佳直線組合。此外,Nakashima 等人[9]使用模糊邏輯分類器,讓基因演算法找 尋適當的模糊規則。然而,要讓基因演算法同時進行發展分類規則及尋找最佳特徵組合 兩種工作,將會使得基因演算法的搜尋過程變得極為複雜,而且會大幅拉長基因演算法 的搜尋時間。. 另外一種方法,是直接使用某種現成的分類器,如 KNN 最近距離分類器、貝氏分 類器、邏輯式迴歸分類器等。在 Huang 等人[6]的研究中,其比較了 KNN、貝式及線性 迴歸三種不同的分類器,在特徵選取上的成效。結果說明不論是何種分類器,透過基因 演算法進行特徵選取後,皆能有效降低其分類錯誤率。不過因為每組資料所適合的分類 器不同,故在不同資料上,每種分類器的效果亦不相同。. 10.
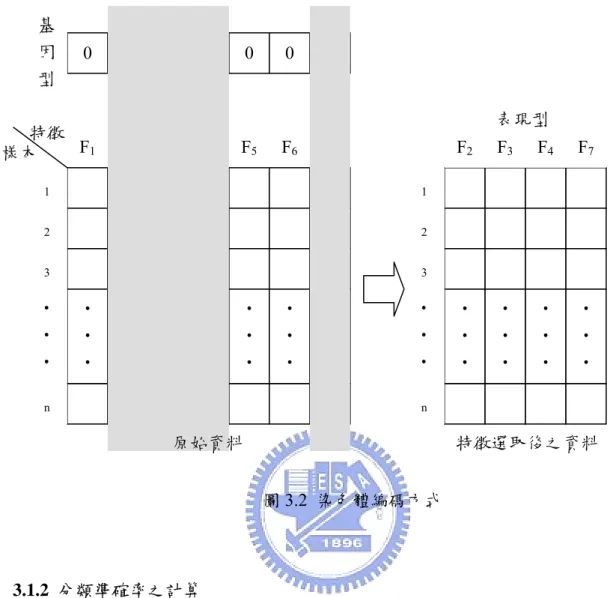
(20) 第三章 研究方法 3.1 限制型利基柏拉圖基因演算法 本研究利用基因演算法進行特徵選取的工作,希望在滿足一定分類準確率之下,找 出各種不同的特徵數,使具有最佳之分類準確率。圖 3.1 顯示本研究所提方法的基本架 構,其中重要的步驟包括如何進行染色體編碼與計算染色體相對應特徵組合的分類錯誤 率,如何決定準確率限制,如何進行柏拉圖及競爭式選取,以及如何選擇所需解等。以 下將進行更詳盡的說明。 染色體編碼 產生親代染色體集合 計算染色體相對應的使用屬性個數及 分類錯誤率 選取適當的染色體,進行交配及突變產 生子代 否. 滿足條件 是 挑選適合的特徵組合. 圖 3.1 特徵選取基本流程 3.1.1 染色體編碼 本研究使用最簡單的編碼規則,每種特徵組合將被編碼成由 0 與 1 所構成的字串, 一條染色體即代表一種特種組合。每條染色體的字串長度將與總特徵數相同,字串中第. k 位置的數字若為 0,代表進行資料分析時,第 k 項特徵將不會被考慮,反之若為 1,則 代表第 k 項特徵將納入考量。其基因型(genotype)與表現型(phenotype)之間的關係如圖. 3.2 所示。 11.
(21) 基 因 型. 0. 特徵 F1 樣本. 1. 1. 1. 0. 0. 1. 表現型. F2. F3. F4. F5. F6. F7. 1. 1. 2. 2. 3. 3. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. n. F2. F3. F4. F7. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. ‧ ‧ ‧. n. 原始資料. 特徵選取後之資料 圖 3.2 染色體編碼方式. 3.1.2 分類準確率之計算 根據染色體所代表的特徵組合將原始資料進行特徵選取後,為了判斷此特徵組合的 優劣,計算其分類準確率是必須的。在本研究中,使用最近歐氏距離分類器(1-NN)作為 評斷分類準確率的工具。於計算訓練資料(training data set)的分類準確率時,訓練資料中 的每一樣本,其預測類別將由在訓練資料中,在歐氏距離上與其最為相近的訓練樣本之 真實類別決定。在計算測試資料(testing data set)之分類準確率時,測試資料中的每一樣 本,其預測類別亦由在訓練資料中之歐氏距離最近樣本決定。. 3.1.3 染色體挑選方法 因為特徵選取問題在判斷特徵組合的優劣時,必須同時考慮特徵數及分類錯誤率, 與其只提供一組符合適應函數的最佳解,倒不如提供使用者多組特徵數及分類錯誤率之. 12.
(22) 間的折衷解,讓使用者自行決定要採取哪一種設定。有鑑於此,本研究採用 Horn 所提 出的利基柏拉圖基因演算法(niched pareto genetic algorithm)來處理基因演算法中,染色 體評分及選取的部分。不過當考慮大量的特徵數時,為了避免基因演算法持續朝減少特 徵數的方向搜尋,而忽略了分類錯誤率,故此處加入分類錯誤率的限制,以減少基因演 算法往錯誤方向搜尋的機會。其詳細步驟如下: 步驟 1. 從親代樣本中隨機挑選兩條染色體作為可產生子代的候選人。. 步驟 2 從親代樣本中隨機挑選 k 條染色體作為比較基準,k 為使用者自訂的競爭集合 染色體數,當 k 越大時,將給予候選人較大的競爭壓力,亦會導致染色體集合 以較快的速度收斂。 步驟 3 將兩條候選染色體逐一與競爭染色體集合比較,紀錄此兩條候選染色體是否有 被任一條競爭染色體擊倒(dominate)。擊倒之意義為:染色體所代表的特徵組 合,其使用於分類之特徵數較多,但分類準確率卻較低。 步驟 4 計算兩條候選染色體之利基數(niche count) mi , mi 代表染色體 i 於樣本集合 中,與其相似之染色體當量數,其計算方法為:. mi =. ∑ sh[d [i, j ]]. (11). j∈Pop. ⎧ ⎛ d ⎪1 − ⎜ sh[d ] = ⎨ ⎜⎝ σ s ⎪ 0 ⎩. ⎞ ⎟⎟ ⎠. if. d <σs. (12). otherwise. 上式中, d [i, j ] 代表染色體 i 與染色體 j 之距離。sh[d]為計算兩染色體之間相似 程度的方程式。 σ s 為使用者自訂的族群半徑。 步驟 5 比較兩染色體候選人是否被競爭染色體集合擊敗,選取未被擊敗的染色體候選 人。若兩者皆被擊敗,或皆未被擊敗,則檢查是否滿足最小準確率限制 C min 。 如果兩條候選染色體其一滿足 C min ,則選取該滿足 C min 之染色體。若兩者皆滿 足或皆不滿足 C min ,則選取利基數 mi 較小的染色體候選人。 步驟 6. 重複步驟 1 到 5,以選出另一條染色體進行交配。. 13.
(23) 3.1.4 交配及突變 在基因演算法產生子代的過程中,所謂的交配,代表染色體間資訊的交換。本研究 採用單點交配法,其操作方法為在染色體上隨機選取一點為交配點(crossover point),然 後根據此交配點,將第一條染色體的後半段,接上第二條染色體的前半段;第二條染色 體的後半段,接上第一條染色體的前半段。例如有兩條親代染色體 100100 與 010110, 若選擇第二個位置為交配點,則產生的兩條子代染色體分別為 100110 與 010100。. 基因演算法的突變操作,可使染色體本身,具有更多樣的發展空間。本方法採用單 點突變法,其操作方法為在染色體上隨機選取一點為突變點(mutation point),如果該位 置的字元為 0,則將之變為 1,反之,若該位置的字元為 1,則將之變為 0,例如現有一 染色體為 100100,若選擇第三點為突變點,所產生的新染色體將為 101100。. 3.1.5 分類準確率限制之決定 利基柏拉圖基因演算法的優點在於透過利基數的限制,使染色體能保持多樣性,藉 由不同的染色體之間的交配,使後代染色體更有機會產生更優秀的解。當加上分類準確 率之限制後,等於限制了基因演算法的搜尋方向,變相的使染色體的多樣性降低了。故 若是分類準確率設得太高,一種可能是因無法搜尋到準確率如此高的解,而使得此限制 如同虛設。另一種可能是使染色體收斂到極少數幾個特定的族群上,使得染色體之間多 樣性大幅的降低,而限制了染色體透過交配以找到更優秀解的能力。. 在本研究中,最小準確率之限制採用每一世代計算一次的方法。於每次世代交替產 生新的親代集合後,取染色體群集中,分類準確率較差的染色體之平均數為下一世代挑 選時的最小準確率限制值。如此將可使準確率限制依當時族群中的分類準確率表現狀 況,進行調整。然而要選取後百分之幾的染色體作為計算準確率限制的基準,在本研究 的嘗試中,建議採用 5%~10%之間。 圖 3.3 為限制型利基基因演算法之詳細操作流程圖。 14.
(24) 染色體編碼. 原始資料. 產生第一代染色體. 根據染色體,決定特徵組合. 訓練資料. 計算特徵組合的使用屬性個數及分類. 測試資料. 錯誤率(訓練組). 計算準確率限制(Cm). 根據每條染色體相對應的屬性數及錯誤 率,利用柏拉圖及競爭式選擇法,選取適 當的染色體. 染色體經過交配及突變操作後,產 生子代. 是. 子代數與親. 否. 代數相同 否. 達到預定的 世代交替數 是 為每條染色體計算使用特徵 數及分類錯誤率(訓練組). 為每條染色體計算分類錯 誤率(測試組). 綜合使用特徵個數及分類 錯誤率等因素,挑選適合 的特徵組合. 圖 3.3 限制型利基柏拉圖基因演算法 15.
(25) 3.2 混合類神經網路及基因演算法之參數最佳化方法 要將一個如製造流程般複雜的系統,用數學模型描述其設定參數及產品輸出值之間 的關係,是一件很困難的事。類神經網路是一種能透過不斷的學習,建構輸入值與輸出 值之間關聯的工具,其已被廣泛應用於此等建模問題上。本研究針對製程參數最佳化之 問題,提供一混合類神經網路及基因演算法之解決方案。. 首先所有的製程參數特徵將透過限制型利基柏拉圖基因演算法,挑選出對產品特性 有重大影響的特徵。接著,透過倒傳遞式(Back Propagation)類神經網路,建構此等重要 特徵與產品特性之間的關聯模型。最後,針對類神經網路所建構出模型,再次利用利基 柏拉圖基因演算法,挑選出最佳的特徵參數設定組合。. 在利用利基柏拉圖基因演算法最佳化製程參數之組合時,每一染色體將代表一種製 程的參數設定。染色體採用實數型的編碼方式,如參數甲的設定為 1、參數乙的設定為. 1.2、參數丙的設定為 0.7,則染色體紀錄為(1, 1.2, 0.7)。將染色體所代表的參數設定套 入訓練好的類神經網路模型後,所得到的產品特性值,即為該染色體的適應分數。此分 數將被利用於染色體的挑選上。. 16.
(26) 第四章 實驗與結果比較 4.1 實驗方法說明 限制型利基柏拉圖基因演算法乃希望透過設定分類準確率閥值,調整利基型柏拉圖 基因演算法的挑選機制,刺激基因演算法往提高分類準確率的方向努力,避免因屬性刪 減過快,導致準確率偏低的問題。此處使用三筆資料測試本研究所提方法的有效性。. 4.1.1 實驗資料 本研究所使用之三筆資料,威斯康辛乳癌及聲納金屬探測來自 UCI 資料庫,肝病診 斷為台灣國泰醫院之實際病歷資料。以下為此三筆資料進行詳細的介紹。. 肝病診斷 此資料為肝病診斷資料,總共包含了 168 筆樣本,其中有 89 筆為良性,79 筆為惡 性。每筆樣本包含 15 項肝病相關診斷屬性,1 項診斷結果。在進行實驗時,本研究將全 部 168 筆資料,隨機挑選出 54 筆類別為良性及 47 筆類別為惡性之樣本作為訓練組資料。 再從剩下的資料中,隨機挑選出 35 筆良性及 32 筆惡性樣本作為測試組資料。經此處理 後之資料使用 1-NN 分類器進行分類時,訓練組之分類準確率為 86.1%,測試組之分類 準確率為 80.1%。. 威斯康辛乳癌 此資料為美國威斯康辛州的乳癌診斷資料,總共包含了 569 筆樣本,其中有 357 筆 為良性腫塊樣本,212 筆為惡性腫塊樣本。每筆樣本包含 30 項乳癌相關診斷屬性,1 項 診斷結果。本研究在進行實驗時,將全部 569 筆資料,隨機挑選出 140 筆類別為良性及. 140 筆類別為惡性之樣本作為訓練組資料。再從剩下的資料中,隨機挑選出 70 筆良性及 70 筆惡性樣本作為測試組資料。經過處理後之資料使用 1-NN 分類器進行分類時,訓練. 17.
(27) 組之分類準確率為 0.946,測試組之分類準確率為 0.957。. 聲納金屬探測 此資料為利用聲納分辨金屬及石頭之資料。總共包含了 208 筆樣本,其中有 111 筆 為金屬,97 筆為石頭。每筆樣本包含 60 項屬性,1 項類別屬性。本研究將將全部 208 筆資料,隨機挑選出 65 筆類別為金屬及 65 筆石頭為惡性之樣本作為訓練組資料。再從 剩下的資料中,隨機挑選出 32 筆金屬及 32 筆石頭樣本作為測試組資料。經過處理後之 資料使用 1-NN 分類器進行分類時,訓練組之分類準確率為 0.846,測試組之分類準確率 為 0.703。. 4.1.2 實驗進行 為了說明設定準確率閥值對所搜尋到的結果之影響,本研究設計了三種實驗加以說 明。 實驗一 此實驗的目的在比較設定不同的分類準確率限制,將對基因演算法搜尋最佳解的過 程產生何種影響。在此實驗中,本研究使用染色體族群重心隨著世代交替的移動情況, 比擬基因演算法的搜尋方向;使用特徵使用率標準差,顯示染色體族群於特徵使用率上 的分布廣度;使用分類準確率標準差,顯示染色體族群於分類準確率上的分布廣度。染 色體族群重心將以(平均特徵使用率 f , 平均分類準確率 c )代表,計算方法如下:. ∑ f (a ). f =. ai ∈P. c=. ai ∈P. i. num( P). (13). ∑ c( a ) i. num( P). (14). 上式中,f(ai)為染色體ai所對應的特徵使用率,c(ai)為染色體ai所對應的分類準確率。特 徵使用率標準差STD(f)及分類準確率標準差STD(c)之計算方法如下:. 18.
(28) ∑ ( f (a ) − f ). ai ∈P. STD ( f ) =. STD(c) =. 2. i. (15). num( P). ∑ (c ( a ) − c ). 2. i. ai ∈P. (16). num( P). 實驗一之相關參數設定如表 4.1。 表 4.1 實驗一參數設定 共同. 突變率. 交配率. 染色體數. 競爭集合染色體數. 族群半徑. 參數. 0.1. 0.8. 50. 3. 0.05. 組別. 代號. 描述. 對照. 0%. 不設定準確率限制。. 5%. 以族群中,分類準確率較低之 5%染色體的分類準確率平均值為閥. 組 實驗 組一. 值。. 10%. 實驗 組二. 以族群中,分類準確率較低之 10%染色體的分類準確率平均值為閥 值。. 20%. 實驗 組三. 以族群中,分類準確率較低之 20%染色體的分類準確率平均值為閥 值。. 實驗二 此實驗的目的,在比較設立準確率限制後,是否能幫助基因演算法搜尋到分類準確 率更高的特徵組合。本研究共選取三種不同的準確率閥值,於實驗開始後,每隔一定的 世代交替數,紀錄當時染色體族群於各種不同使用特徵數下,所能找到分類準確率最高 的特徵組合,並觀察準確率閥值的高低,與所找到的最佳解之間的關係。每種準確率閥 值設定,於同一測試資料上,皆會重複進行三次實驗,比較時將會把此三次實驗之結果 19.
(29) 整合,以加強實驗結果的說服力。實驗二之相關參數設定如表 4.2。 表 4.2 實驗二參數設定 共同. 突變率. 交配率. 染色體數. 競爭集合染色體數. 族群半徑. 參數. 0.1. 0.8. 50. 3. 0.05. 組別. 代號. 描述. 對照. 0%. 不設定準確率限制。. 5%. 以族群中,分類準確率較低之 5%染色體的分類準確率平均值為閥. 組 實驗 組一. 值。. 10%. 實驗 組二. 以族群中,分類準確率較低之 10%染色體的分類準確率平均值為閥 值。. 實驗三 此實驗的目的在比較相同的實驗設定下,不設定準確率閥值,但使用多一倍的染色 體進行搜尋工作,能否使基因演算法找到分類準確率更高的特徵組合。又其效果與限制 型利基柏拉圖演算法相比,兩種方法之間的差異。實驗三之相關參數設定如表 4.3。. 20.
(30) 表 4.3 實驗三參數設定 共同. 突變率. 交配率. 競爭集合染色體數. 族群半徑. 參數. 0.1. 0.8. 3. 0.05. 組別. 代號. 描述. 對照. 0%. 不設定準確率限制,使用染色體數為 100。. 5%. 以族群中,分類準確率較低之 5%染色體的分類準確率平均值為閥. 組 實驗. 值。使用染色體數為 50。. 組一. 以族群中,分類準確率較低之 10%染色體的分類準確率平均值為閥. 10%. 實驗. 值。使用染色體數為 50。. 組二. 4.2 實驗結果 4.2.1 肝病診斷 實驗一 圖 4.1 至圖 4.3 為實驗過程中,染色體族群重心在實驗開始後 20 代、50 代及 100 代內之移動趨勢。圖 4.4 及圖 4.5 則為特徵使用率標準差及分類準確率標準差之紀錄圖。 訓練資料 20代 1. 分類準確率. 0.9. 0% 5%. 0.8. 10% 20%. 0.7 0.6 0. 0.1. 0.2. 0.3. 0.4. 0.5. 特徵使用率. 圖 4.1 前 20 代趨勢圖(肝病) 21. 0.6.
(31) 訓練資料 50代 1. 分類準確率. 0.9. 0% 5%. 0.8. 10% 20%. 0.7 0.6 0. 0.1. 0.2. 0.3. 0.4. 0.5. 0.6. 特徵使用率. 圖 4.2 前 50 代趨勢圖(肝病). 訓練資料 100代 1. 分類準確率. 0.9. 0% 5% 10% 20%. 0.8 0.7 0.6 0. 0.1. 0.2. 0.3. 0.4. 0.5. 0.6. 特徵使用率. 圖 4.3 前 100 代趨勢圖(肝病). 特徵使用率標準差. 0.2. 0% 5% 10% 20%. 0.1. 0 0. 50. 100. 150. 世代交替數. 圖 4.4 特徵使用率變異趨勢圖(肝病) 22. 200.
(32) 分類準確率標準差. 0.2. 0% 5% 10% 20%. 0.1. 0 0. 50. 100. 150. 200. 世代交替數. 圖 4.5 分類準確率變異趨勢圖(肝病) 觀察圖 4.1 至圖 4.3,雖然一開始族群的特徵使用率及分類準確率之重心相差不大, 但隨著世代交替數的增加,重心的移動方向卻不盡相同。在不設定準確率限制(0%)的狀 況下,族群的重心快速的朝向左下方前進。在短短的二十代內,染色體族群的平均特徵 使用率已經從 50%減少到 12%,不過在分類準確率上,卻沒有上升的趨勢。而設定準確 率閥值後,隨著限制的高低不同,基因演算法的搜尋方向也隨之調整,限制越高,搜尋 方向越往上調整。但此一現象,有可能只是因設定準確率限制,會將分類準確率較差之 染色體踢除,導致重心會有向上偏移的假象,事實上並沒有搜尋到分類準確率較高的特 徵組合。圖 4.4.及圖 4.5 顯示準確率閥值越高,分類準確率標準差越低,而於特徵使用 率上則無明顯差別。此點顯示,設置準確率限制,並不會使得染色體族群於特徵使用率 上的分布廣度降低,但會使得染色體族群於分類準確率上的分布廣度降低。. 實驗二 在實驗二中,本研究分別針對 0%、5%、10%三種不同的準確率限制條件,每種條 件皆重複進行三次演化計算。比較其在經過 50 代、100 代、200 代演化後,於各種不同 使用特徵數下,綜合訓練組分類準確率及測試組分類準確率兩種指標,所能找到之最佳 解(圖 4.6 至圖 4.8)。. 23.
(33) 訓練資料 50代. 測試資料 50代. 100.0%. 100.0%. 90.0%. 90.0% 分類準確率. 分類準確率. 0% 5% 10% 80.0%. 0% 5% 10% 80.0%. 70.0%. 70.0% 0. 1. 2. 3. 4. 5. 6. 7. 8. 9 10 11 12 13 14 15. 0. 1. 2. 3. 4. 5. 6. 使用特徵數. 7. 8. 9 10 11 12 13 14 15. 使用特徵數. 圖 4.6 50 代之最佳解(肝病 實驗二) 訓練資料 100代. 測試資料 100代. 100.0%. 100.0%. 90.0%. 90.0% 分類準確率. 分類準確率. 0% 5% 10% 80.0%. 0% 5% 10% 80.0%. 70.0% 0. 1. 2. 3. 4. 5. 6. 7. 8. 70.0%. 9 10 11 12 13 14 15. 0. 使用特徵數. 1. 2. 3. 4. 5. 6. 7. 8. 9 10 11 12 13 14 15. 使用特徵數. 圖 4.7 100 代之最佳解(肝病 實驗二) 訓練資料 200代. 測試資料 200代. 100.0%. 100.0%. 90.0%. 90.0% 0%. 分類準確率. 分類準確率. 0% 5% 10% 80.0%. 5% 10% 80.0%. 70.0%. 70.0% 0. 1. 2. 3. 4. 5. 6. 7. 8. 9 10 11 12 13 14 15. 0. 使用特徵數. 1. 2. 3. 4. 5. 6. 7. 8. 9 10 11 12 13 14 15. 使用特徵數. 圖 4.8 200 代之最佳解(肝病 實驗二) 24.
(34) 在不加準確率限制(0%)的情況下,可以明顯的觀察到,使用特徵數大於四的解,其 分類準確率不管在訓練組或在測試組皆表現較差。且隨著世代交替數的增加,此情況會 愈形嚴重,到了 200 代時,甚至根本沒有使用特徵數高於四的解。而加上準確率限制後, 隨著限制的升高,將可幫助基因演算法往提升分類準確率的方向搜尋,而非以減少特徵 使用數為優先考量。而在準確率限制為 10%的狀況下,其所搜尋到使用特徵數為 1 及 2 的特徵組合,分類準確率似乎越來越差,但使用特徵數為 5 及 6 的特徵組合,分類準確 率有稍微的提升。不過在訓練組擁有較高的分類準確率,並不一定在測試組也能有同樣 的表現。. 實驗三 實驗三將不設定準確率閥值之實驗,增加其染色體使用數量一倍,其實驗結果如圖. 4.9 至圖 4.11。 訓練資料 50代. 測試資料 50代. 100.0%. 100.0%. 90.0%. 90.0% 分類準確率. 分類準確率. 0% 5% 10% 80.0%. 0% 5% 10% 80.0%. 70.0% 0. 1. 2. 3. 4. 5. 6. 7. 8. 70.0%. 9 10 11 12 13 14 15. 0. 1. 2. 3. 4. 5. 使用特徵數. 6. 7. 8. 9 10 11 12 13 14 15. 使用特徵數. 圖 4.9 50 代之最佳解(肝病 實驗三). 25.
(35) 訓練資料 100代. 測試資料 100代. 100.0%. 100.0%. 90.0%. 90.0% 分類準確率. 分類準確率. 0% 5% 10% 80.0%. 0% 5% 10% 80.0%. 70.0% 0. 1. 2. 3. 4. 5. 6. 7. 8. 70.0%. 9 10 11 12 13 14 15. 0. 使用特徵數. 1. 2. 3. 4. 5. 6. 7. 8. 9 10 11 12 13 14 15. 使用特徵數. 圖 4.10 100 代之最佳解(肝病 實驗三) 訓練資料 200代. 測試資料 200代. 100.0%. 100.0%. 90.0% 分類準確率. 90.0% 0%. 分類準確率. 0% 5% 10% 80.0%. 5% 10% 80.0%. 70.0% 70.0%. 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15. 0 1. 使用特徵數. 2 3. 4 5 6. 7 8. 9 10 11 12 13 14 15. 使用特徵數. 圖 4.11 200 代之最佳解(肝病 實驗三) 將族群之染色體數增加一倍,的確可搜尋到分類準確率較高的解。比較在相同世代 交替數下,設定準確率限制與讓染色體增加一倍兩種方法,兩者所找到的解互有高低, 不過將染色體數增加一倍,將可找到較多的可行解,但代價是需要較多的運算時間。. 4.2.2 威斯康辛乳癌 實驗一 圖 4.12 至圖 4.14 為染色體族群重心在實驗開始後 50 代、100 代及 200 代內之移動 趨勢。圖 4.15 及圖 4.16 則為特徵使用率標準差及分類準確率標準差之紀錄圖。 26.
(36) 訓練資料 50代. 分類準確率. 1. 0%. 0.9. 5% 10% 0.8. 20%. 0.7 0. 0.1. 0.2. 0.3. 0.4. 0.5. 0.6. 特徵使用率. 圖 4.12 前 50 代趨勢圖(乳癌). 訓練資料 100代. 分類準確率. 1. 0%. 0.9. 5% 10% 0.8. 20%. 0.7 0. 0.1. 0.2. 0.3. 0.4. 0.5. 0.6. 特徵使用率. 圖 4.13 前 100 代趨勢圖(乳癌). 訓練資料 200代. 分類準確率. 1. 0. 0.9. 0.05 0.1 0.8. 0.2. 0.7 0. 0.1. 0.2. 0.3. 0.4. 0.5. 特徵使用率. 圖 4.14 前 200 代趨勢圖(乳癌) 27. 0.6.
(37) 特徵使用率標準差. 0.2. 0% 5% 10% 20%. 0.1. 0 0. 50. 100. 150. 200. 世代交替數. 圖 4.15 特徵使用率變異趨勢圖(乳癌). 分類準確率標準差. 0.2. 0% 5% 10% 20%. 0.1. 0 0. 50. 100. 150. 200. 世代交替數. 圖 4.16 分類準確率變異趨勢圖(乳癌) 在前 50 代時,族群的特徵使用率及分類準確率之重心相差不大,皆是水平的朝左 方移動,到了 100 代後,不設定閥值之重心才稍微領先往左下方移動。而隨著世代交替 數的增加,閥值設定為 5%之族群重心,亦開始下降,但閥值設定為 10%及 20%之族群 重心,卻不再有明顯的變化。由圖 4.16,可推斷,不設定閥值之族群重心,約在 70 代 左右開始大幅下降,而閥值設定為 5%之族群重心,約在 120 代時開始下降。原因應為 此資料所使用之原始屬性,包含太多交互作用項的緣故,故雖大量的刪減屬性,依然可 以維持很高的分類準確率。. 28.
(38) 實驗二 實驗二分別針對 0%、5%、10%三種不同的準確率限制條件,每種條件皆重複進行 三次演化計算。比較其在經過 50 代、100 代、200 代演化後,於各種不同使用特徵數下, 綜合訓練組分類準確率及測試組分類準確率兩種指標,所能找到之最佳解(圖 4.17 至圖. 4.19)。 訓練資料 50代. 測試資料 50代. 1. 0%. 分類準確率. 分類準確率. 1. 5%. 0.9. 10%. 0% 5%. 0.9. 10%. 0.8. 0.8 0. 2. 4. 6. 0. 8 10 12 14 16 18 20 22 24 26 28 30. 2. 4. 6. 8 10 12 14 16 18 20 22 24 26 28 30 使用特徵數. 使用特徵數. 圖 4.17 50 代之最佳解(乳癌 實驗二) 訓練資料 100代. 測試資料 100代. 100.0%. 0%. 分類準確率. 分類準確率. 100.0%. 5%. 90.0%. 10%. 80.0% 0. 2. 4. 6. 0% 5%. 90.0%. 10%. 80.0%. 8 10 12 14 16 18 20 22 24 26 28 30. 0. 使用特徵數. 2. 4. 6. 8 10 12 14 16 18 20 22 24 26 28 30 使用特徵數. 圖 4.18 100 代之最佳解(乳癌 實驗二). 29.
(39) 訓練資料 200代. 測試資料 200代. 100.0%. 分類準確率. 分類準確率. 100.0%. 0% 5%. 90.0%. 10%. 80.0%. 0% 5%. 90.0%. 10%. 80.0% 0 2 4 6 8 10 12 14 16 18 20 22 24 26 28 30. 0 2 4 6 8 10 12 14 16 18 20 22 24 26 28 30. 使用特徵數. 使用特徵數. 圖 4.19 200 代之最佳解(乳癌-實驗二) 在不加準確率限制(0%)的情況下,在經過 50 次世代交替時,其表現似乎較有準確 率限制的狀況要好。隨著世代交替數的增加,其所搜尋到使用特徵數為 4 至 7 之特徵組 合,分類準確率表現漸漸升高,不過使用特徵數較多的特徵組合,其分類準確率卻漸漸 的降低,到了 200 代後,其所搜尋到的最佳解已經集中在使用特徵數為 8 以下的特徵組 合了。在準確率限制為 10%的狀況下,不管經過幾次世代交替,其所找到之特徵組合, 改變並不大,不管在分類準確率或使用特徵數量上,皆非最佳。原因應出在此資料的分 類準確率很高,使得準確率限制為 10%的條件過於嚴苛,造成染色體族群之變異性大幅 降低,同時也阻礙了基因演算法搜尋更佳解的能力。而在準確率限制為 5%的狀況下, 雖然在世代交替為 50 時,表現並不亮眼,但在世代交替數為 100 及 200 時,其所搜尋 到的柏拉圖邊際,卻有很好的表現。原因應在此狀況下,於前五十代之染色體變異不大, 而在七十代左右,因為染色體之間的變異開始拉大,使得基因演算法有了更好的表現。. 實驗三 實驗三比較增加染色體數量與設定準確率閥值兩種方法效果上之差異,其實驗結果 如圖 4.20 至圖 4.22。. 30.
(40) 訓練資料 50代. 測試資料 50代. 1. 0%. 分類準確率. 分類準確率. 1. 5%. 0.9. 10%. 0.8. 0% 0.9. 5% 10%. 0.8 0. 2. 4. 6. 8 10 12 14 16 18 20 22 24 26 28 30. 0. 2. 4. 6. 8. 使用特徵數. 10 12 14 16 18 20 22 24 26 28 30 使用特徵數. 圖 4.20 50 代之最佳解(乳癌 實驗三) 訓練資料 100代. 測試資料 100代. 100.0%. 0% 90.0%. 分類準確率. 分類準確率. 100.0%. 5% 10%. 0% 5%. 90.0%. 10%. 80.0% 0. 2 4. 6 8 10 12 14 16 18 20 22 24 26 28 30. 80.0% 0. 使用特徵數. 2. 4. 6. 8 10 12 14 16 18 20 22 24 26 28 30 使用特徵數. 圖 4.21 100 代之最佳解(乳癌 實驗三) 訓練資料 200代. 測試資料 200代. 100.0%. 分類準確率. 分類準確率. 100.0%. 0% 5%. 90.0%. 10%. 80.0%. 0% 5%. 90.0%. 10%. 80.0% 0 2 4 6 8 10 12 14 16 18 20 22 24 26 28 30. 0 2 4 6 8 10 12 14 16 18 20 22 24 26 28 30. 使用特徵數. 使用特徵數. 圖 4.22 200 代之最佳解(乳癌 實驗三) 31.
(41) 在前 50 代時,不設定準確率限制而將族群染色體數增加一倍的方法,所找到的最 佳特徵組合,比其他兩者為佳,此差距在使用特徵數小於 8 的解上尤其明顯。不過當世 代交替數增加時,其所搜尋到的最佳解將會迅速的集中到使用特整數較少的區域。雖然 使用較多的染色體進行蒐尋,的確可以幫助基因演算法找到分類準確類較高的特徵組 合,但隨著世代交替數的增加,此方法依然會因解集中在特徵數較少的部分,而失去找 尋分類準確率更高的特徵組合之能力。. 4.2.3 聲納金屬探測 實驗一 圖 4.23 至圖 4.26 為染色體族群重心在實驗開始後 50 代、100 代、200 代及 300 代 內之移動趨勢。圖 4.27 及圖 4.28 為特徵使用率標準差及分類準確率標準差之紀錄圖。 訓練資料 50代. 分類準確率. 1. 0%. 0.9. 5% 10% 0.8. 20%. 0.7 0. 0.1. 0.2. 0.3. 0.4. 特徵使用率. 圖 4.23 前 50 代趨勢圖(聲納). 32. 0.5. 0.6.
(42) 訓練資料 100代. 分類準確率. 1. 0%. 0.9. 5% 10% 0.8. 20%. 0.7 0. 0.1. 0.2. 0.3. 0.4. 0.5. 0.6. 特徵使用率. 圖 4.24 前 100 代趨勢圖(聲納). 訓練資料 200代. 分類準確率. 1. 0%. 0.9. 5% 10% 0.8. 20%. 0.7 0. 0.1. 0.2. 0.3. 0.4. 0.5. 0.6. 特徵使用率. 圖 4.25 前 200 代趨勢圖(聲納). 訓練資料 300代. 分類準確率. 1. 0% 5% 10% 20%. 0.9. 0.8. 0.7 0. 0.1. 0.2. 0.3. 0.4. 0.5. 特徵使用率. 圖 4.26 前 300 代趨勢圖(聲納) 33. 0.6.
(43) 特徵使用率標準差. 0.2. 0% 5% 10% 20%. 0.1. 0 0. 50. 100. 150. 200. 250. 300. 世代交替數. 圖 4.27 特徵使用率變異趨勢圖(聲納). 分類準確率標準差. 0.2. 0% 5% 10% 20%. 0.1. 0 0. 50. 100. 150. 200. 250. 300. 世代交替數. 圖 4.28 分類準確率變異趨勢圖(聲納) 觀察圖 4.23 至圖 4.26,準確率限制為 10%及 20%之重心皆是朝左上方移動,且移 動路徑差異不大。在不設定準確率限制(0%)的狀況下,族群的重心朝向左方前進,不過 在約 150 代左右,重心開始明顯的下降。而準確率限制為 5%之重心移動路徑,與不設 定準確率限制之狀況差不多,不過重心下降較為和緩。而在分類準確率標準差及特徵使 用率標準差的部分,情況與前兩筆資料類似,因設立準確率限制所造成的染色體變異縮 減,在分類準確率較為明顯,而在特徵使用率部分則不明顯。. 34.
(44) 實驗二 實驗二針對 0%、5%、10%三種不同的準確率限制條件,每種條件皆重複進行三次 演化計算。比較其在經過 100 代、200 代、300 代演化後,於各種不同使用特徵數下, 綜合訓練組分類準確率及測試組分類準確率兩種指標,所能找到之最佳解(圖 4.29 至圖. 4.31)。 測試資料 100代. 100.0%. 100.0%. 90.0%. 90.0% 0%. 分類準確率. 分類準確率. 訓練資料 100代. 5%. 80.0%. 10%. 70.0%. 0% 80.0%. 5% 10%. 70.0%. 60.0% 0. 5. 60.0%. 10 15 20 25 30 35 40 45 50 55 60. 0. 使用特徵數. 5. 10. 15. 20. 25. 30. 35. 40. 45. 50. 55. 60. 使用特徵數. 圖 4.29 100 代之最佳解(聲納-實驗二) 測試資料 200代. 100.0%. 100.0%. 90.0%. 90.0% 0%. 分類準確率. 分類準確率. 訓練資料 200代. 5%. 80.0%. 10%. 70.0%. 0% 5%. 80.0%. 10%. 70.0%. 60.0%. 60.0% 0. 5. 10 15 20 25 30 35 40 45 50 55 60. 0. 使用特徵數. 5. 10 15 20 25 30 35 40 45 50 55 60 使用特徵數. 圖 4.30 200 代之最佳解(聲納-實驗二). 35.
(45) 測試資料 300代. 100.0%. 100.0%. 90.0%. 90.0% 0%. 分類準確率. 分類準確率. 訓練資料 300代. 5%. 80.0%. 10%. 70.0%. 0% 80.0%. 5% 10%. 70.0%. 60.0%. 60.0% 0. 5. 10 15 20 25 30. 35 40 45 50 55 60. 0. 使用特徵數. 5. 10. 15. 20. 25. 30. 35. 40. 45. 50. 55. 60. 使用特徵數. 圖 4.31 300 代之最佳解(聲納-實驗二) 在訓練資料的部份,隨著世代交替數的增加,三種不同的分類準確率限制條件,使 得基因演算法所求得之最佳解漸漸的形成三個部分。在減少特徵數上,不設定準確率限 制,擁有最好的表現。而準確率限制為 5%之狀況,可找到使用特徵數在 5 至 15 之間的 最高分類準確率特徵組合。但準確率限制為 10%之狀況,其所找到特徵數大於 15 之最 佳特徵組合,擁有比其他兩種條件所能找到之特徵組合更高之分類準確率。. 實驗三 實驗三比較增加染色體數量與設定不同的準確率閥值之差異,其實驗結果如圖 4.32 至圖 4.34。. 36.
(46) 測試資料 100代. 100.0%. 100.0%. 90.0%. 90.0% 0% 5%. 80.0%. 10%. 分類準確率. 分類準確率. 訓練資料 100代. 70.0%. 0% 80.0%. 5% 10%. 70.0%. 60.0% 0. 5. 60.0%. 10 15 20 25 30 35 40 45 50 55 60. 0. 使用特徵數. 5. 10. 15. 20. 25. 30. 35. 40. 45. 50. 55. 60. 使用特徵數. 圖 4.32 100 代之最佳解(聲納-實驗三) 測試資料 200代. 100.0%. 100.0%. 90.0%. 90.0% 0% 5%. 80.0%. 10%. 分類準確率. 分類準確率. 訓練資料 200代. 70.0%. 0% 80.0%. 5% 10%. 70.0%. 60.0% 0. 5. 60.0%. 10 15 20 25 30 35 40 45 50 55 60. 0. 使用特徵數. 5. 10. 15. 20. 25. 30. 35. 40. 45. 50. 55. 60. 使用特徵數. 圖 4.33 200 代之最佳解(聲納-實驗三) 測試資料 300代. 100.0%. 100.0%. 90.0%. 90.0% 0% 5%. 80.0%. 10%. 分類準確率. 分類準確率. 訓練資料 300代. 70.0%. 0% 80.0%. 5% 10%. 70.0%. 60.0% 0. 5. 10. 15 20 25 30 35 40. 60.0%. 45 50 55 60. 0. 使用特徵數. 5. 10. 15. 20. 25. 30. 35. 使用特徵數. 圖 4.34 300 代之最佳解(聲納-實驗三) 37. 40. 45. 50. 55. 60.
(47) 將染色體數增加一倍的方法,的確大幅的增進利基柏拉圖基因演算法所求得解的表 現,尤其在使用特徵數為 10 個以下的特徵組合,其分類準確率大幅的領先限制型利基 柏拉圖基因演算法所求得的解。不過在使用特徵數較多的部分,其表現與限制型利基柏 拉圖基因演算法就相差不大。而隨著世代交替數的增加,即使使用多一倍的染色體數搜 尋,依然會發生所搜尋到的特徵組合,較容易偏向減少使用特徵數的方向。. 38.
(48) 第五章. DVD 製程參數之最佳化. 5.1 背景簡介. DVD(Digital Versatile Disk)是數位多功能光碟的簡稱,因其早期僅被使用於視聽方 面,故又稱為數位影音光碟(Digital Video Disk)。DVD 是繼 CD-R (Compact. Disk-Recordable)後,另一數位儲存裝置的重大突破,與傳統的數位儲存媒體相比,其在 容量及讀取速度上皆有大幅的提升。. 觀察 CD-R 的發展歷史,當記錄倍速一直被提高,讀取光碟片上之資料時,雜訊將 會隨之放大,所以也導致高倍速光碟片的良率一直無法有效的提高。在 DVD 導入 8 倍 速產品時,逐漸有此問題的浮現,隨著目前 DVD 在市場上逐漸將 16 倍速的成品導入量 產,此問題的嚴重性亦日漸提升。根據光碟機廠商的要求,循軌(Tracking)的雜訊必須被 控制在一定的數值以下,其記錄過程會較順暢,當超出規格時,就容易導致降速、中斷 或寫後訊號超出管控規格的現象。. 本研究中的個案公司是一座落於新竹工業區的高科技公司,當該公司將 16 倍速. DVD 逐步導入量產時,發現循軌誤差(Tracking Error,TE)及聚焦誤差(Focus Error,FE) 為光碟片不良的最大問題,佔產品不良原因之 27%,其中又以循軌誤差較為嚴重。TE 代表光學拾訊器(Optical Pickup Units)雷射的聚焦點與數據所在軌道之偏離程度,FE 代 表光學拾訊器雷射的聚焦點與光碟片所在平面之偏離程度。當 DVD 光碟片於此兩個指 標表現越高時,代表燒錄碟片的動作越容易失敗。在本研究中,選用 Plextor 8x 的 DVD 燒錄機進行非破壞性檢測,並將 TE 的品檢規格設定為「低於 37」 ,FE 的品檢規格設定 為「低於 20」,預估該公司改善前的 TE 平均值約為 40,FE 平均值約為 23,整體良率 約為 30%。. DVD 光碟片的製造流程主要包括刻板、基板成型、塗佈與膠合等四大項,簡扼敘 39.
(49) 述如下:. 1. 刻版:透過雷射刻印、電鑄、研磨清洗等步驟製造模板(stamper)。 2. 基板成型:使用刻板流程所製造之模板為模型,以射出成型的方式製造 DVD 光碟片 的資料儲存用基板,及空白(dummy)基板。. 3. 塗佈:在資料儲存用基板上塗佈染料及濺鍍金屬。 4. 膠合:將塗佈完成之資料儲存用基板及空白基板以膠黏合。. 本研究希望利用限制型利基柏拉圖基因演算法,從上述四個製程中,找出對 DVD 光碟片成品之 TE 及 FE 兩指標具有重大影響的製程因子。接著,透過類神經網路建構 重要製程因子與光碟片成品 TE、FE 之間的關係模型,最後利用利基柏拉圖基因演算法 針對此關係模型進行製程參數最佳化之工作,期找出能使 TE 及 FE 最小化之製程參數 設定,以改善產品品質。. 5.2 製程重要製程參數之選擇 本研究從 DVD 製造的四個製程中,總共選出 27 個製程參數作為重要製程參數的候 選人,並將類別型製程參數中的每一類別獨立為一項特徵,使得資料的總輸入特徵擴展 為 49 個。實驗之總收集樣本數為 46 筆,其中於 TE 超過品檢標準者有 28 筆,於 FE 超 過品檢標準者有 9 筆。進行特徵選取時,此 46 筆樣本將被拆為訓練組 32 筆與測試組 14 筆。. 5.2.1 循軌誤差之特徵選取 有關影響 TE 之特徵選取,本研究以 TE 高於 37 者為不良品,低於或等於 37 者為 良品。訓練組之不良品樣本共有 19 筆,測試組之不良品樣本共有 9 筆。使用 1-NN 分類 器進行分類時,訓練組之分類準確率為 65.6%,測試組之分類準確率為 85.7%。. 使用限制型利基基因演算法進行實驗時,其參數設定如表 5.1。在準確率限制的設 40.
(50) 定上,本研究同樣使用三種不同的限制以作為對照,每種限制皆重複進行三次實驗,實 驗結果如表 5.2 所示。 表 5.1 限制型利基柏拉圖基因演算法之參數設定 突變率 交配率 染色體數 世代交替數 競爭集合染色體數 族群半徑 準確率限制. 0.1. 0.8. 100. 200. 4. 0.05. 0, 0.05, 0.1. 表 5.2 循軌誤差之特徵選取結果 準確率限制 = 0. 準確率限制 = 0.05. 準確率限制 = 0.1. 編. 分類準確率. 使用特. 分類準確率. 使用特. 分類準確率. 使用特. 號. 訓練. 測試. 徵數. 訓練. 測試. 徵數. 訓練. 測試. 徵數. 組. 組. 組. 組. 組. 組. 1. 75.0%. 71.4%. 1. 78.1%. 85.7%. 2. 81.3%. 85.7%. 3. 2. 78.1%. 92.9%. 2. 78.1%. 92.9%. 3. 75.0%. 85.7%. 4. 3. 81.3%. 85.7%. 3. 78.1%. 92.9%. 4. 78.1%. 78.6%. 5. 4. 75.0%. 78.6%. 4. 75.0%. 78.6%. 5. 78.1%. 85.7%. 6. 5. 75.0%. 92.9%. 5. 87.5%. 71.4%. 5. 81.3%. 71.4%. 8. 6. 78.1%. 92.9%. 6. 78.1%. 85.7%. 6. 78.1%. 78.6%. 9. 7. 75.0%. 92.9%. 7. 81.3%. 85.7%. 7. 78.1%. 71.4%. 11. 8. 81.3%. 85.7%. 7. 81.3%. 85.7%. 8. 81.3%. 71.4%. 12. 9. 78.1%. 85.7%. 8. 81.3%. 71.4%. 13. 最後決定要選擇哪些製程參數,進行 TE 部分之建模工作時,本研究以準確率限制 為 0.05 所找到的八個最佳特徵組合為基礎,累計每一原始的製程參數於這些最佳特徵組 合中的出現次數,選擇其中累計出現次數高於一次者。本研究共選出了九個製程參數, 分別為顯影時間、空白基板模具、夾具壓力、成型線、資料儲存用基板模具、模具溫度. 41.
(51) 最小值、塗佈線、染料一平均濃度、染料二平均濃度。. 5.2.2 聚焦誤差之特徵選取 有關影響 FE 之特整選取,本研究以 FE 高於 20 者為不良品,低於或等於 20 者為 良品。訓練組之不良品樣本共有 6 筆,測試組之不良品樣本共有 3 筆。使用 1-NN 分類 器進行分類時,訓練組之分類準確率為 68.8%,測試組之分類準確率為 64.3%。. 使用限制型利基基因演算法進行實驗時,其參數設定如表 5.1 與 TE 部分相同。其 實驗結果如表 5.3 所示。 表 5.3 聚焦誤差之特徵選取結果 準確率限制 = 0. 準確率限制 = 0.05. 準確率限制 = 0.1. 編. 分類準確率. 使用特. 分類準確率. 使用特. 分類準確率. 使用特. 號. 訓練. 測試. 徵數. 訓練. 測試. 徵數. 訓練. 測試. 徵數. 組. 組. 組. 組. 組. 組. 1. 68.8%. 78.6%. 1. 81.3%. 71.4%. 1. 78.1%. 85.7%. 2. 2. 81.3%. 71.4%. 1. 78.1%. 78.6%. 2. 81.3%. 71.4%. 2. 3. 84.4%. 78.6%. 2. 81.3%. 78.6%. 3. 81.3%. 71.4%. 3. 4. 75.0%. 85.7%. 3. 87.5%. 64.3%. 4. 87.5%. 71.4%. 4. 5. 84.4%. 78.6%. 3. 87.5%. 71.4%. 5. 84.4%. 78.6%. 5. 6. 84.4%. 71.4%. 6. 87.5%. 71.4%. 6. 7. 87.5%. 64.3%. 6. 87.5%. 71.4%. 7. 最後決定要選擇哪些製程參數,進行 FE 部分之建模工作時,本研究以準確率限制 為 0.1 所找到的七個最佳特徵組合為基礎,累計每一原始的製程參數於這些最佳特徵組 合中的出現次數,選擇其中累計出現次數高於一次者,本研究共選出了六個製程參數, 42.
(52) 分別為電鑄機槽別、輸入厚度、空白基板模具、成型線、資料儲存用基板模具、塗佈線、 染料一平均濃度。. 5.3 類神經網路模型之構建 於建構類神經網路模型時,本研究將原始的 46 筆樣本分為訓練組 36 筆與測試組 10 筆,採用倒傳遞式(back propagation)的學習方法,使用的軟體為 Qnet,並使用其內建之 學習率控制方法。類神經網路之隱藏層神經元數使用試誤法,學習率(learning rate)與動 量(momentum)的設定則如表 5.4 所示。 表格 5.4 學習率與動量之設定 學習率初始值 學習率最小值 學習率最大值 動量. 0.1. 0.001. 0.1. 0.8. 9-9-1 模式 此模式為 TE 指標之類神經網路模型,輸入層使用的神經元數目為 9 個,代表 5.2.1 節中所選出之影響 TE 的九項製程參數,隱藏層使用的神經元數目為 9 個,輸出層使用 的神經元數目為 1 個,以 TE 為學習目標。經過 12000 次的訓練後,此網路之 RMSE(root. mean square error)於訓練組為 0.067,於測試組為 0.120。使用 TE 之品檢標準,依類神經 網路輸出值將樣本分為兩類後,訓練組之分類準確率為 86.1%,測試組之分類準確率為. 90%。. 6-7-1 模式 此模式為 FE 指標之類神經網路模型,輸入層使用的神經元數目為 6 個,代表 5.5.2 節中所選出之影響 FE 的六項製程參數,隱藏層使用的神經元數目為 7 個,輸出層使用 的神經元數目為 1 個,以 FE 為學習目標。經過 17000 次的訓練後,此網路之 RMSE 於 訓練組為 0.074,於測試組為 0.138。使用 FE 之品檢標準,依類神經網路輸出值將樣本. 43.
數據


Outline
相關文件
介面最佳化之資料探勘模組是利用 Apriori 演算法探勘出操作者操作介面之 關聯式法則,而後以法則的型態儲存於介面最佳化知識庫中。當有
Keywords: light guide plate, stamper, etching process, Taguchi orthogonal array, back-propagation neural networks, genetic algorithms, analysis of variance, particle
Thus, the proposed approach is a feasible and effective method for process parameter optimization in MIMO plastic injection molding and can result in significant quality and
我們提出利用讀取器與讀取器間的距離為參數來優化利用分層移除讀取器之方法的最佳 化技術 Distance Based Optimization for Eliminating Redundant Readers (DBO) ,此方法完全
實習老師 教 甄試教者 實 習老師 輔導 老師 正式教 師 教授,研 究員 同事..
譚志忠 (1999)利用 DEA 模式研究投資組合效率指數-應用
中華大學應用數學研究所 研 究 生:黃仁性 敬啟 指導教授:楊錦章 教授 中華民國九十六年..
aaacggctcatcgtceaaaggcgtaetgccatgctaaatctggtacccggcaagcagea tgtgaaacgctggaacatctgaecgtgagaaggatgeccaggaatagaaaaatacatc