分類蛋白質質譜資料變數選取的探討 - 政大學術集成
57
0
0
全文
(2) 謝辭 至今,論文的完成最感謝的就是我的指導老師-郭訓志博士。記得第一次與老師會 面聊天時,老師對我說道「我覺得你選擇跟我很有勇氣。」,而當下我的心中其實也早 已決定要為我所作的選擇負責到底。在撰寫論文的過程中,感謝老師先將分析的流程講 解的很詳細,利於發展我之後的研究方向。此外除了感謝老師不時的在過程中提供在學 術界常用的統計方法、相關期刊、書籍以及其專業知識外,尤其感謝老師訓練我研究生 該有的研究精神、遇到困難的解決方法以及作事情該具備的程序和態度,相信對於之後 出社會的我會有很大的幫助。. 政 治 大 一開始還擔心同學們會因為我是私校畢業的而疏遠我或不想跟我作朋友,不過還好這些 立. 兩年半的碩士求學生涯終於要完成了。想當初從私立大學考進國立的統研所就讀,. ‧ 國. 學. 情形後來都沒有發生。感謝同學們在我撰寫論文的期間提供我程式上問題的協助、對於 期刊內容的看法以及翻譯的幫忙,此外大家一起為彼此打氣、加油的感覺真的很好。. ‧. 最後,要感謝我的爸爸和媽媽。在撰寫論文的過程中,對於你們我常常出現情緒化. sit. y. Nat. 的現象,如不耐煩等等…,但你們卻都能給予無限的包容和忍耐,仍然會在我睡前或是. n. al. er. io. 去學校前表達對我的關心和鼓勵。此外也感謝你們體諒我常常拒絕在假日時與你們一起. v. 出去散步的邀約。這段期間你們真的辛苦了,而未來我一定會用我的孝心好好的報答你 們對於我的寬容和體諒。. Ch. engchi. i Un. 林婷婷 2012 年 6 月. I.
(3) 摘要 本研究所利用的資料是來自美國東維吉尼亞醫學院所提供的攝護腺癌蛋白質質譜 資料,其資料有原始資料和另一筆經過事前處理過的資料,而本研究是利用事前處理過 的資料來作實証分析。由於此種資料通常都是屬於高維度資料,故變數間具有高度相關 的現象也很常見,因此從大量的特徵變數中選取到重要的特徵變數來準確的判斷攝護腺 的病變程度成為一個非常普遍且重要的課題。那麼本研究的目的是欲探討各(具有懲罰 項)迴歸模型對於分類蛋白質質譜資料之變數選取結果,藉由 LARS、Stagewise、 LASSO、Group LASSO 和 Elastic Net 各(具有懲罰項)迴歸模型將變數選入的先後順序. 政 治 大 Kruskal-Wallis 檢定)以及 SVM「分錯率排序」的判別結果相比較。而分析的結果顯示, 立 當作其排序所產生的判別結果與利用「統計量排序」(t 檢定、ANOVA F 檢定以及. ‧ 國. 學. Group LASSO 對於六種兩兩分類的分錯率,其分錯率趨勢的表現都較其他方法穩定,並 不會有大起大落的現象發生,且最小分錯率也幾乎較其他方法理想。此外 Group LASSO. ‧. 在四分類的判別結果在與其他方法相較下也顯出此法可得出最低的分錯率,亦表示若須. n. al. er. io. sit. y. Nat. 同時判別四種類別時,相較於其他方法之下 Group LASSO 的判別準確度最優。. v. 關鍵詞:LARS、Forward Stagewise、LASSO、Group LASSO、Elastic Net、支持向量機。. Ch. engchi. II. i Un.
(4) Summary Our research uses the prostate proteomic spectra data which is offered by Eastern Virginia Medical School. The materials have raw data and preprocessed data. Our research uses the preprocessed data to do the analysis of real example. Because this kind of materials usually have high dimension, so it maybe has highly correlation between variables very common, therefore choose from a large number of characteristic variables to accurately determine the pathological change degree of the Prostate is become a very general and important subject. Then the purpose of our research wants to discuss every (penalized). 政 治 大 LARS, Stagewise, LASSO, Group LASSO and Elastic Net, each variable is chosen 立. regression model in variable selection results for classifying the proteomic spectra data. With. ‧ 國. 學. successively by each (penalized) regression model, and it is regarded as each variable’s order then produce discrimination results. After that, we use their results to compare with using. ‧. statistic order (t-test, ANOVA F-test and Kruskal-Wallis test) and SVM fault rate order. And. sit. y. Nat. the result of analyzing reveals Group LASSO to two by two of six kinds of rate by mistake. n. al. er. io. that classify, the mistake rate behavior of trend is more stable than other ways, it doesn’t. i Un. v. appear big rise or big fall phenomenon. Furthermore, this way’s mistake rate is almostly more. Ch. engchi. ideal than other ways. Moreover, using Group LASSO to get the discrimination result of four classifications has the lowest mistake rate under comparing with other methods. In other words, when must distinguish four classifications in the same time, Group LASSO’s discrimination accuracy is optimum.. Key words: LARS, Forward Stagewise, LASSO, Group LASSO, Elastic Net, SVM.. III.
(5) 目錄 第一章 緒論………………………………………………………………………………….1 第一節 研究背景………………………………………………………………………...1 第二節 研究動機與目的………………………………………………………………...2 第三節 研究架構………………………………………………………………………...3 第二章 蛋白質質譜資料介紹……………………………………………………………….4 第一節 表面強化雷射解析電離飛行質譜技術………………………………………...4 第二節 攝護腺癌蛋白質質譜資料……………………………………………………...5. 政 治 大 分析方法…………………………………………………………………………...10 立. 第三章 文獻回顧…………………………………………………………………………….7 第四章. ‧ 國. 學. 第一節 分析流程……………………………………………………………………….10 第二節 統計量排序…………………………………………………………………….14. ‧. 第三節 LARS、Stagewise、LASSO 迴歸模型…………………………………………15. sit. y. Nat. 第四節 Group LASSO 迴歸模型………………………………………………………20. n. al. er. io. 第五節 Elastic Net 迴歸模型…………………………………………………………...21. v. 第六節 支持向量機 SVM………………………………………………………………24. Ch. engchi. i Un. 第五章 實証分析…………………………………………………………………………...26 第一節 R 函數之設定…………………………………………………………………..27 第二節 探討兩兩分類之分錯率結果………………………………………………….28 第三節 探討四分類之分錯率結果…………………………………………………….42 第六章 分析結果討論與建議……………………………………………………………...45 參考文獻……………………………………………………………………………………...47 附錄一………………………………………………………………………………………...50. IV.
(6) 表目錄 表 2.1. 四種類別之受測人數和樣本筆數………………………………………………….5. 表 2.2. 事前處理的部份資料……………………………………………………………….6. 表 3.1. AUC 搭配決策樹之兩兩分類的敏感度、特異度以及分錯率……………………..9. 表 4.1. 對於每個特徵變數產生的統計量之值取平均過程……………………………...11. 表 4.2. 對於每個特徵變數產生的等級取平均之過程…………………………………...12. 表 4.3. 對於每個特徵變數產生的 SVM 平均分錯率之流程…………………………….13. 表 4.4. 對於前兩百名特徵變數依序代入 SVM 建模之流程…………………………….13. 表 5.1. 訓練資料和測試資料筆數………………………………………………………...26. 表 5.2. 政 治 大 七種合併資料刪除零後之特徵變數個數………………………………………...27 立 各特徵選取方法於兩兩分類上之最小分錯率與組合數………………………...35. 表 5.4. 對於一組訓練資料 LARS、Stagewise 以及 LASSO 配適迴歸模型過程之時間…36. 表 5.5. LARS、Stagewise、LASSO 於各兩兩分類中之每組訓練資料配適迴歸模型過程. ‧. ‧ 國. 學. 表 5.3. sit. y. Nat. 的平均步驟數………………………………………………………………………………...37. 表 5.7. 各兩兩分類中 Elastic Net 以及 AUC 決策樹的分錯率結果……………………...41. 表 5.8. Elastic Net 特徵選取方法與判定係數萃取 SVM 串聯法之分錯率比較………...42. 表 5.9. 各特徵選取方法於四分類上之最小分錯率與組合數…………………………...44. n. al. er. 各變數在 LARS、Stagewise 以及 LASSO 中迴歸係數的變化……………………37. io. 表 5.6. Ch. engchi. V. i Un. v.
(7) 圖目錄 圖 2.1. 「表面強化雷射解析電飛行質譜技術」流程圖…………………………………...4. 圖 3.1. 兩兩分類之決策樹………………………………………………………………….9. 圖 4.1. 訓練資料和測試資料之抽樣方法………………………………………………...10. 圖 4.2. 二維自變數下的 LARS……………………………………………………………16. 圖 4.3. SVM 原理示意圖…………………………………………………………………..24. 圖 5.1. 各特徵選取方法下判別正常與良性腫瘤之分錯率趨勢圖……………………...28. 圖 5.2. 各特徵選取方法下判別正常與癌症早期之分錯率趨勢圖……………………...29. 圖 5.3. 各特徵選取方法下判別正常與癌症晚期之分錯率趨勢圖……………………...30. 圖 5.4. 政 治 大 各特徵選取方法下判別良性腫瘤與癌症早期之分錯率趨勢圖………………...31 立 各特徵選取方法下判別良性腫瘤與癌症晚期之分錯率趨勢圖………………...32. 圖 5.6. 各特徵選取方法下判別癌症早期與癌症晚期之分錯率趨勢圖………………...33. 圖 5.7. LARS、Stagewise 以及 LASSO 在 NO vs. BPH 中之 X 6 的迴歸係數估計值之變. ‧. ‧ 國. 學. 圖 5.5. y. sit. n. al. er. 各特徵選取方法下四分類之分錯率趨勢圖……………………………………...43. io. 圖 5.9. Nat. 化……………………………………………………………………………………………...40. Ch. engchi. VI. i Un. v.
(8) 第一章 緒論 第一節 研究背景 攝護腺,又稱為前列腺。雖然攝護腺從被發現至今已超過 2300 年,但一直到近代, 醫學專家才開始研究其構造、生理作用以及病理變化。 攝護腺是只有男生才有的生殖器官,它位於膀胱下方、直腸前面,外形像核桃,且 圍繞著尿道。而攝護腺是由三葉組成,外層覆有被膜,兩側為精囊,精囊為一對囊狀腺 體。那麼發育完成的成人之攝護腺腺體可分成邊緣、中央與過渡區三個區塊,其中邊緣 區占體積的 70%、中央區占 25%而過渡區在這兩區之間,占 5%的體積。那麼「攝護腺. 政 治 大. 肥大」以及「攝護腺癌」是最常見的攝護腺疾病,其中「攝護腺肥大」其實就是攝護腺. 立. 增生(良性腫瘤),最主要是發生在過渡區的腺體,因此攝護腺肥大容易產生連道阻塞的. ‧ 國. 學. 現象。另外,「攝護腺癌」主要發生在邊緣區的腺體,所以攝護腺肥大和攝護腺癌發生 病變的的位置是在不同的區塊,兩者的病理變化也完全獨立發展,故這兩者容易同時存. ‧. 在。(蒲永孝和黃昌淵,1997). y. Nat. sit. 雖然亞洲國家的攝護腺癌盛行率低於西方,可是近 20 年來大部分的亞洲國家的發. n. al. er. io. 生率和死亡率也都逐年上升。台灣行政院衛生署統計,攝護腺癌新診斷人數從 1993 年. i Un. v. 的 801 人逐年增加,2007 年更增加到了 3367 人。統計每 10 萬人口死於攝護腺癌的比率,. Ch. engchi. 在 1993 年是 2.5 人 , 2008 年則升高至 7.7 人。在男性癌症的死亡率排行中,攝護腺癌 慢慢往前竄升,從 1995 年的 10 名外到 2001 年也升至第 7 名,且維持至今。之所以有 這樣的趨勢是因為”老年人口增加(老年人口越多,診斷出攝護腺癌的機會就越 高)”、”診斷率大幅提升(由於攝護腺特異性抗原(Prostate-specific antigen,PSA)的運 用)”以及”生活型態改變(西化飲食製造更多肥胖者)”。(簡邦平,2006) 早期在診斷攝護腺癌時,就是驗血清中的 PSA。它是一種由攝護腺產生的蛋白質, 當攝護腺發生病變時,PSA 就會升高;數值越高的話,癌的機率也就越高,擴散的程度 也越大。若 PSA>20 時,則幾乎就確定是癌症。若 PSA>100,則癌細胞應已擴散至骨骼 了。雖然 PSA 的敏感度有達到 90%以上,但特異性卻只有 25%,表示這項指標還是有 1.
(9) 缺陷。不過,還好有「表面強化雷射解吸電離飛行質譜技術」(Surface-Enhanced Laser Desorption Ionization- Time of Flight,SELDI)的問世。這種診斷方法,不僅有高的敏感 度也有高的特異性。若在未來這種技術能夠被有效推廣的話,勢必能降低台灣攝護腺癌 症的發生率和死亡率了。所以我們欲利用這種技術所得出的數據來發覺攝護腺在不同時 期中蛋白質的變化,並找出精確的生物標誌。(潘荔錞等人,2003;賴基銘,2004). 第二節 研究動機與目的 本研究是利用美國東維吉尼亞醫學院所提供的攝護腺癌蛋白質質譜資料來作分 析。而此資料的自變數是為多個質量電荷比的蛋白質(我們往後將稱它為特徵變數),此. 政 治 大 因為這筆資料的應變數是屬於類別型的,因此會讓我們聯想到分類準確度的問題,故在 立 外,樣本數也小於這些特徵變數個數,故我們會將其視為一種「高維度資料」。另外,. ‧ 國. 學. 節省人力和時間花費的前提下,我們會希望由這如此大量且具有高度相關的高維度資料 中選取對分類結果有幫助的特徵變數來判別分類結果即可,所以很常見又普遍的作法是. ‧. 先藉由特徵選取這步驟再代入分類器得出其分類結果。(Guyon 等人,2002;Degroeve 等. y. sit. Nat. 人,2002;Weston 等人,2003;Ma 和 Huang,2005). n. al. er. io. 那麼所謂的特徵選取其實就是由訓練集中盡可能的發現那些對分類結果沒有用處. v. 的變數,並將其刪除的一種過程。而最後剩餘下來的這些變數集合,不僅可降低原資料. Ch. engchi. i Un. 的維度,且對於我們的分類結果也有所幫助。其實在過去十年中,將特徵選取的技術應 用至生物資訊學中對於高維度資料的建模、序列分析、微陣列分析和質譜分析已相當普 遍。(Efron 等人,2001;Somorjai 等人,2003;Jiang 等人,2004;Fox 和 Dimmic 等 人,2006) 在以往特徵選取的步驟中,利用各特徵變數之統計量的顯著性來排序是很常見的作 法。那麼除了此排序方法外,我們其實也可以考慮各特徵變數被選入迴歸模型中的順序 當作其排序,舉例來說若以向前選取法來選取變數的話,第一步就被選入模型的變數, 由於它與應變數最相關,故我們就可以將它排序為第一;到了第二步被選入模型的變數 就將它排序為第二…。然而在這麼多種迴歸模型選取變數的方法中,本研究考慮了近期 2.
(10) 在學界很著名的縮減維度的懲罰性迴歸方法-最小絕對值壓縮和選取(Least Absolute Shrinkage and Selection Operator,LASSO),因為這方法也可同時達成估計迴歸係數和縮 減維度的目的,另外由於 LASSO 可以算是屬於最小角度迴歸(Least Angle Regression, LARS)的變形,而向前逐段迴歸(Forward Stagewsie Regression)也可由 LARS 變形而成, 所以我們除了想探討上述三種迴歸模型之特徵選取情形之外,另外也將 Zou 和 Hastie (2004)提出在高維度的情況下,選取變數的表現上比 LASSO 更令人滿意的彈性網路 (Elastic Net)迴歸模型以及 Yuan 和 Lin (2007)為了改善 LASSO 在高維度資料中的缺點 而提出的 Group LASSO 迴歸模型一併加入本研究的探討。. 第三節 研究架構 政 治 大 本文一共分為六個章節。第二章為蛋白質質譜資料介紹,其中第一節簡述, 立. ‧ 國. 學. 表面強化雷射解析電離飛行質譜技術、第二節說明攝護腺癌蛋白質質譜資料之內容。接 著第三章是文獻回顧,然後第四章分析方法,共分六節,第一節說明分析流程,第二節. ‧. 說明統計量排序方法、第三節說明 LARS、Stagewise 以及 LASSO 的迴歸模型及其演算. sit. y. Nat. 法、第四節說明 Group LASSO、第五節是 Elastic Net 及其演算法以及第六節支持向量機. n. al. er. io. SVM 的原理。再來第五章為實証分析,其中第一節為 R 函數之設定、第二節是探討兩. v. 兩分類之分錯率結果以及第三節探討四分類之分錯率結果,而第六章為結論與建議。. Ch. engchi. 3. i Un.
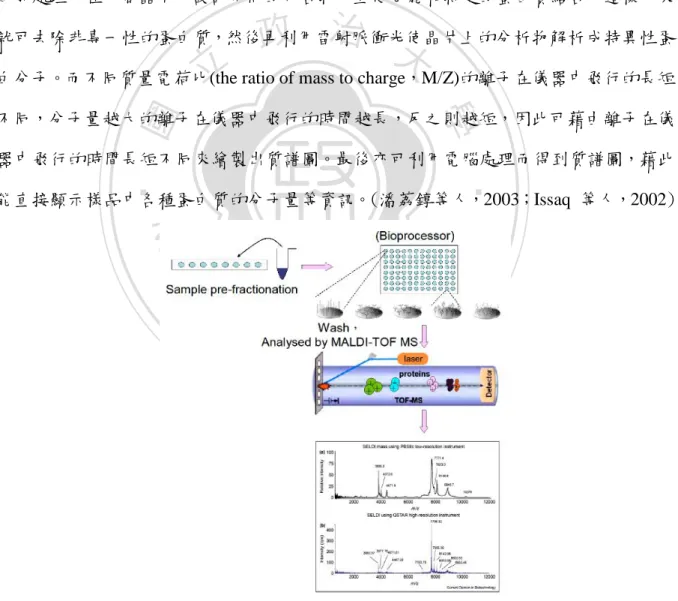
(11) 第二章 蛋白質質譜資料介紹 第一節 表面強化雷射解析電離飛行質譜技術 「表面強化雷射解吸電離飛行質譜技術」英文名稱為Surface-Enhanced Laser Desorption Ionization- Time of Flight(SELDI-TOF)是美國Ciphergen Biosystems 公司根 據基質輔助雷射脫附游離飛行時間質譜儀(Matrix-Assisted Laser Desorption/Ionization Time of Flight Mass Spectrometer,MALDI-TOF MS)為基礎所改良出來的產品。它結合 了生物晶片的概念,而SELDI-TOF技術的流程如圖2.1,首先先在蛋白質晶片上進行樣 本前處理,在每個晶片中設計不同的化合物,並使它能和特定的蛋白質結合,這樣一來. 政 治 大. 就可去除非專一性的蛋白質,然後再利用雷射脈衝光使晶片上的分析物解析成特異性蛋. 立. 白分子。而不同質量電荷比(the ratio of mass to charge,M/Z)的離子在儀器中飛行的長短. ‧ 國. 學. 不同,分子量越大的離子在儀器中飛行的時間越長,反之則越短,因此可藉由離子在儀 器中飛行的時間長短不同來繪製出質譜圖。最後亦可利用電腦處理而得到質譜圖,藉此. ‧. 能直接顯示樣品中各種蛋白質的分子量等資訊。(潘荔錞等人,2003;Issaq 等人,2002). n. er. io. sit. y. Nat. al. Ch. engchi. i Un. v. 圖 2.1 「表面強化雷射解析電飛行質譜技術」流程圖 資料來源:“The SELDI-TOF MS Approach to Proteomics: Protein Profiling and Biomarker Identification” by Issaq, H. L., Veenstra, T. D., Conrads, T. P. and Felschow, D., 2002, Biochemical and Biophysical Research Communications, 589. 4.
(12) 第二節 攝護腺癌蛋白質質譜資料 本研究所採用的資料是由美國東維吉尼亞醫學院(Eastern Virginia Medical School) 利用「表面強化雷射解析電離飛行質譜技術」所建立的攝護腺癌蛋白質質譜資料。而此 資料其實有原始資料(raw data)以及另一組事前處理的資料(preprocessed data)。在此, 我們是以人工處理的這組資料來作實証分析,那麼所謂的人工處理,就是將原始質譜資 料進行扣除基線、正規化和校準等動作。故根據專家的意見,只保留原始資料中其蛋白 質的質量電荷比落在 2000 至 40000M/Z 之間的資料,然後進行人工處理後的資料只會擁 有 779 個特徵變數。. 政 治 大 分成四種類別,分別為正常人(Normal,NO)、良性腫瘤(Begin Prostate Hyperplasia,BPH)、 立 而這組人工處理的攝護腺癌蛋白質質譜資料收集了 326 位受測者的觀測資料,而且. ‧ 國. 學. 癌症早期(Prostate Cancer Stage A and B,CAB)以及癌症晚期(Prostate Cancer Stage C and D,CCD),此外,每一位受測者都進行兩次的重複實驗,故每位受測者皆會有兩筆樣本. ‧. 筆數,如表 2.1,總計共有 652 筆資料。. sit. y. Nat. al. n. 類別. 正常. 受測人數. 82. 樣本筆數. 164. er. io. 表 2.1 四種類別之受測人數和樣本筆數. i 癌症早期 n C良性腫瘤 U hengchi 77 84 154. 168. v. 癌症晚期. 總計. 83. 326. 166. 652. 此外,表 2.2 為事前處理的部份資料。其中每一行為受測者編號,在此我們定義以 下表格中的 i-1 是表示第 i 位受測者的第一筆資料,i-2 是表示第 i 位受測者的第二筆資 料,而第 i 位受測者前頭也會同時標示著所屬類別。此外,每一列則是表示特徵變數 (M/Z),總共會有 779 個,而下表僅列出四種類別的部份資料。在每個細格中的數值是 表示某受測者對應至某特徵變數(蛋白質或縮氨酸)所偵測到的強度(intensity)。. 5.
(13) 表 2.2 事前處理的部份資料 特徵變數. M/Z. NO1-1. NO1-2. NO2-1. NO2-2. NO3-1. NO3-2. 1. 2009.089071. 0. 0. 0. 0. 0. 0. 2. 2013.865841. 0. 0. 2.131. 0.823. 0.813. 1.609. 3 2013.8658 4 2020.5369 41 5 2021.5333 25 2021.9094 66 779. 2020.536925. 0. 0. 0. 0. 0. 0. 2021.533366. 0. 0. 0. 0. 0. 0. 2021.909411. 0. 0. 0. 0. 0. 0. . . . . . . . 39965.84865. 0.026. 0.038. 0.031. 0. 0.106. 0.067. 11 特徵變數. M/Z. BPH1-1. BPH1-2. BPH2-1. BPH2-2. BPH3-1. BPH3-2. 1. 2009.089071. 0. 0. 0. 0. 0. 0. 2. 2013.865841. 0. 7.198. 0. 0. 3. 2020.536925. 0. 0. 0. 4. 2021.533366. 立0. 0. 5. 2021.909411. . 政 00 治 6.72 大 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. . . . . . . . 779. 39965.84865. 0. 0. 0. 0. 0.045. 0.076. 特徵變數. M/Z. CAB1-1. CAB1-2. CAB2-1. CAB2-2. CAB3-1. CAB3-2. 1. 2009.089071. 0. 0. 0. 0. 0. 0. 2. 2013.865841. 0. 0. 0. 0. 0. 0. 3. 2020.536925. 0. 0. 0. 0. 0. 0. 4. 2021.533366. 0. 0. 0. 0. 0. 5. 2021.909411. 0. 0. 0. . . 779. 39965.84865. 特徵變數. y. sit. io. n. . e n g0 c h i U0. er. Nat. al 0 0C h. ‧. ‧ 國. 0. 學. 0. v ni. . . . . . 0.057. 0.051. 0. 0. 0.047. 0.053. M/Z. CCD1-1. CCD1-2. CCD2-1. CCD2-2. CCD3-1. CCD3-2. 1. 2009.089071. 0. 0. 0. 0. 0. 0. 2. 2013.865841. 0. 14.987. 9.076. 4.143. 0. 0. 3. 2020.536925. 0. 0. 0. 0. 0. 0. 4. 2021.533366. 0. 0. 0. 0. 0. 0. 5. 2021.909411. 0. 0. 0. 0. 0. 0. . . . . . . . . 779. 39965.84865. 0. 0. 0. 0. 0. 0. 6.
(14) 第三章 文獻回顧 黃仁澤(2005)提出一篇名為「對於高維度資料進行特徵選取-應用於分類蛋白質質譜 儀資料」的論文。作者同時採用原始質譜資料以及事前處理的質譜資料,不過以原始資 料為主、事前處理資料為輔來作實証分析。其目的為藉由原始質譜資料來進行特徵選 取,除了找出有利於判別兩兩分類以及四分類的特徵選取方法外,亦可得知對於判別分 類病況時所組成的特徵變數個數。詳細作法即先將資料分為一百組的訓練集和測試集, 然後以最小分錯率特徵選取法和最小p值特徵選取法將所有特徵變數依其對應到的p值 由小到大排序,再以遞增選取的方式選取前200個特徵變數,並依序代入支持向量機. 政 治 大 因作者發現特徵變數間存在共線性的問題,故又進而發展三種特徵萃取的方法,分別為 立. (Support vector machine,SVM)中建模,藉此找出分錯率最好的特徵變數組合數。此外,. ‧ 國. 學. k-mean分群萃取法、最大相關係數萃取法以及判定係數萃取法。其分析的結果顯示對於 分類原始資料時,利用判定係數萃取法搭配最小p值特徵選取法可得最佳的分類結果。. ‧. 而在本研究中,我們將會與該作者所使用之最小分錯率特徵選取方法和最小p值特徵選. sit. y. Nat. 取方法加上遞增選取方式的分類結果來進行比較。. n. al. er. io. 另外,陳詩佳(2007)也提出將後設學習(Meta-Learning)應用至蛋白質質譜資料的特. v. 徵選取方法。其目的為利用後設學習結合分類器搭配逐步選取特徵變數的方法,希望找. Ch. engchi. i Un. 出能夠利用較少的特徵變數來將資料分類並達到較高正確率的特徵選取方式。其中,後 設學習就是把每個分類器融合成一個多元分類器,文章中作者運用到三種分類器,分別 為線性判別分析(Linear Discriminant Analysis,LDA)、第K位最接近鄰居(K-th Nearest Neighbor,KNN)以及SVM,並將其結合為一個多元分類器,而作者結合的方法又可分 為多數表決法(Majority Vote)、權重投票法(Weighted Vote)以及串聯法(Cascading)。那麼 此篇作者先將資料分為一百組的訓練集以及測試集,然後作者利用投票法來分類樣本。 其中多數表決法是計算某一個特徵變數在LDA、KNN和SVM下的平均分類正確率,權 重投票法來預測每個樣本最後會被預測到的類別。之後,作者又考慮兩種串聯法來結合 多種分類器,其中所謂的串聯法就是利用反覆地將分類器結合的過程,串接所有分類器 7.
(15) 的預測結果,而每次都會用到前一次之預測結果,然後不斷的更新。一種是多個分類器 的串聯方法,而另一種是單一分類器的串聯方法,此處作者是用支持向量機SVM來作單 一分類器的串聯。而作者將多個分類器串聯的分類結果、串聯SVM的分類結果與只有用 SVM的分類結果比較時,發現就算增加特徵變數也無法提升正確率,故作者就利用 Elastic Net加上SVM單一分類器的串聯方法以及判定係數萃取法加上SVM串聯來試圖改 善此現象,最後與只用SVM的分類結果比較時,可得出Elastic Net加上SVM單一分類器 的串聯方法可稍微改善上述情形,但正確率卻有些許的降低,而判定係數萃取方法確實 能夠達到僅用較少的變數來提升正確率。那麼在本研究中,我們將會採用作者所提供的 判定係數萃取法搭配SVM串聯法之分類結果來進行比較分析。. 政 治 大. 而在外國文獻中,Adam 等人(2002)提出藉由計算每個特徵變數在ROC曲線下的面. 立. 積,並找出合乎其所設門檻值的變數以便加入決策樹中產生分類結果。作者欲利用多種. ‧ 國. 學. 蛋白質來找出更好的生物標誌改善以往攝護腺特異性抗原(PSA)診斷敏感度高、特異性. ‧. 低的缺點, 於是利用到事前處理的攝護腺癌蛋白質質譜資料,且將資料分為正常人、良. y. Nat. 性腫瘤、癌症早期和癌症晚期,然後將癌症早期和晚期合併為癌症病患,接著將正常人、. er. io. sit. 良性腫瘤和癌症病患的所有樣本資料設為訓練集,藉由盲眼測試(blinded test)將15位正 常人、15位良性腫瘤和30位癌症病患的資料設為測試集。而其分析方法就是將事前處理. al. n. iv n C 的攝護腺癌蛋白質質譜資料中的779個特徵變數先計算其ROC曲線以下的面積(Area hengchi U. under ROC Curve,AUC),其中ROC曲線的產生方式就是在一個縱軸為敏感度、橫軸為 1-特異度的二維平面中,利用診斷工具或診斷方式不斷變動的情形下,所畫出的的一種 凹向橫軸的曲線。因此,一旦產生ROC曲線後即可算出AUC,於是作者將AUC 0.62的 那124個特徵變數放入決策樹中來建立分類模型。而兩兩分類之分析結果如圖3.1,一共 找出了9個重要的特徵個數,分別是4475、5074、5382、7024、7820、8141、9149、9507 和9656這9個特徵變數,然後共產生10個節點,並利用這10個節點來判別受測者是否為 正常人(Normal)、良性腫瘤(BPH)和癌症病患(Prostate Cancer,PCA)。. 8.
(16) 圖3.1 兩兩分類之決策樹 資料來源:“Serum Protein Fingerprinting Coupled with a Pattern-matching Algorithm. 政 治 大. Distinguishes Prostate Cancer from Benign Prostate Hyperplasia and Healthy Men” by Adam, B. L., Qu, Y., Davis, J. W., Ward, M. D., Clements, M. A., Cazares, L. H., Semmes, O. J.,Schellhammer, P. F., Yasui, Y., Feng, Z. and Wright, G. L. Jr., 2002, Cancer Research, 62(13), 3611.. 立. ‧. ‧ 國. 學. 最後表3.1列出AUC搭配決策樹之兩兩分類的判別結果,其中包含敏感度和特異度. sit. n. al. er. io. 表3.1. y. Nat. 以及分錯率數值。. Ch. i Un. v. AUC搭配決策樹之兩兩分類的敏感度、特異度以及分錯率. engchi. 兩兩分類. 敏感度. 特異度. 分錯率. 正常人vs.良性腫瘤. 93%. 100%. 3.3%. 良性腫瘤vs.癌症早期. 93%. 80%. 13.3%. 癌症早期vs.癌症晚期. 80%. 87%. 16.6%. 正常人vs.癌症. 83%. 100%. 11%. 良性腫瘤vs.癌症. 83%. 93%. 13.3%. 正常與良性腫瘤vs.癌症. 83%. 97%. 10%. 9.
(17) 第四章 分析方法 第一節 分析流程 在事前處理的攝護腺癌蛋白質質譜資料中,我們有四種類別,分別為正常、良性腫 瘤、癌症早期以及癌症晚期病人之資料。若想比較兩兩類別或四種類別在各種特徵選取 方法下之分類效果,其過程大致可分為三個部份。首先,先將資料分為訓練資料和測試 資料,再來排序特徵變數,最後將排名前兩百名的特徵變數依序放入 SVM 中建模,並 得出最低的分錯率結果以及其所對應的特徵變數組合數。詳述如下: 第一部份:將欲分析的資料分為一百組的訓練資料和測試資料. 政 治 大. 如圖 4.1 所示,假使我們想比較正常人和良性腫瘤病人在各特徵選取方法下之分類. 立. 效果為何,則我們必須先在正常人和良性腫瘤的資料中分別抽百之分六十六點七的樣本. ‧ 國. 學. 資料作為訓練資料以及百分之三十三點三的樣本作為測試資料。此外每次選取樣本(某 受測者)時須同時選入(某人)兩次重複的觀測值:. ‧. 訓練資料. y. Nat. BPH. n. al. er. io. sit. NO. 66.7%. Ch. i Un. v. 66.7%. engchi. 33.3%. 33.3%. 測試資料. 圖 4.1 訓練資和測試資料之抽樣方法 重複上述的程序一百次,即可產生一百組的訓練資料以及一百組的測試資料。. 10.
(18) 第二部份:排序特徵變數 在排序這個部份,我們對於每組訓練資料中的特徵變數利用其「統計量排序」和被 「選入迴歸模型之順序排序」及其個別的「分錯率排序」。其中「統計量排序」我們是 利用 t 檢定、ANOVA F 檢定以及 Kruskal-Wallis 檢定來得之變數的統計量。而「選入迴 歸模型之順序排序」我們所採用的迴歸估計式分別有 Least Angel Regression、Forward Stagewise regression、LASSO、Group LASSO 以及 Elastic Net,藉此來得到各變數被選 入模型的順序。在「分錯率排序」的部份我們是採用支持向量機 SVM 來得出各變數之 分錯率。而以上講到的這些方法的理論部份將在此章的第二、三、四、五、六節呈現給 大家。. 政 治 大. 我們先概述如何利用「統計量排序」 ,如表 4.1 所示,我們以 t 檢定為例來說明,對. 立. 於每組訓練資料中的每一個特徵變數進行 t 檢定時,由於每個特徵變數在一百組訓練資. ‧ 國. 學. 料中皆可得到一個 t 統計量的值,最後將此一百個統計值取平均可得 ti ,表示第 i 個特. ‧. 徵變數之平均統計值,並利用它來排序每個特徵變數。而平均統計值最小的特徵變數排. y. sit. io. n. al. er. 檢定也依此作法。. Nat. 第一個,而平均統計值最大的特徵變數排最後一個。故 ANOVA F 檢定和 Kruskal-Wallis. Ch. 表 4.1 對於每個特徵變數產生的統計量之值取平均過程. engchi. i Un. v. 訓練資料組別. 平均統計值. 特徵變數 1. 2. . 100. ti. X1. t1,1. t1, 2. . t1,100. t1. X2. t 2,1. t 2, 2. . t 2,100. t2. . . . . . . X 779. t779,1. t779, 2. . t779,100. t779. 11.
(19) 再來我們說明「選入迴歸模型之順序排序」的方法。如表 4.2 所示,對於每組訓練 資料中的每個特徵變數配適 Least Angel Regression、Forward Stagewise Regression、 LASSO、Group LASSO 以及 Elastic Net 模型時,即可得到一個被選入迴歸模型的「等 級」(Rank),這裡的等級意思即若某個特徵變數的等級為 1,就表示其特徵變數是第一 個被選入迴歸模型的;若某個特徵變數的等級為 779,則表示其特徵變數是最後一個被 選入迴歸模型的。因為每個特徵變數在一百組訓練資料中皆可得到一個等級,再將這一 百個等級取平均即 Ri ,最後利用它來排序每個特徵變數。平均等級最小的特徵變數排第 一個、平均等級最大的特徵變數排最後一個。. 政 治 大. 表 4.2 對於每個特徵變數產生的等級取平均之過程. 訓練資料組別. . R1,1. R1, 2. . R 2,1. R 2, 2. . . . engchi. . X 779. R 779,1. R1,100. R1. R 2,100. R2. n. al. . . R 779,100. R 779. sit. . Ri. er. . io. X2. 100. y. 2. 平均等級. ‧. 1. Nat. X1. 學. 特徵變數. ‧ 國. 立. Ch. R 779, 2. i Un. v. 而「分錯率排序」就是利用支持向量機 SVM 來得出每個特徵變數的分錯率,並利 用分錯率由低至高來排序這些特徵變數。如表 4.3,將每組訓練資料中的每個特徵變數 分別代入支持向量機中配適模型,然後再計算所建構出來的模型在測試資料下的分錯 率。因每個特徵變數在一百組的訓練資料下皆可產生一個分錯率,最後即利用每個特徵 變數的平均分錯率( SV M i )來作為排序這些特徵變數的依據。. 12.
(20) 表 4.3 對於每個特徵變數產生的 SVM 平均分錯率之流程 訓練資料組別. 平均分錯率. 特徵變數 1. 2. . 100. SV M i. X1. SVM 1,1. SVM 1, 2. . SVM 1,100. SV M 1. X2. SVM 2,1. SVM 2, 2. . SVM 2,100. SV M 2. . . . . . . X 779. SVM 779,1. SVM 779, 2. . SVM 779,100. SV M 779. 政 治 大. 立. 第三部分:將排序在前兩百名的特徵變數依序代入 SVM 中建模. ‧ 國. 學. 由第二個部份所得出的 779 個特徵變數的排名順序後,我們就取前兩百名特徵變數依序 代入 SVM 中建模,而所謂依序代入的意思就如表 4.4 中所示,先將順位第一的特徵變. ‧. 數帶入 SVM 中建模得出模型 M 1 ,然後由第一和第二順位的變數代入 SVM 中建模組成. y. Nat. io. sit. 模型 M 2 ,將前兩百名變數依循此方式建模即可得兩百組的模型。藉由預測一百組測試. n. al. er. 資料的結果來分別得知分錯率,最後對其取平均而得各模型之平均分錯率。表中 M i 即. Ch. 表示第 i 個模型的平均分錯率。 表 4.4. engchi. i Un. v. 對於前兩百名特徵變數依序代入 SVM 建模之流程 模型的平 均分錯率. 測試資料組別 特徵變數. 模型 1. 2. . 100. Mi. X (1). M1. M 1,1. M 1, 2. . M 1,100. M1. X (1) , X ( 2). M2. M 2,1. M 2, 2. . M 2,100. M2. . . . . . . . X (1) X ( 200). M 200. M 200,1. M 200, 2. . M 200,100. M 200. 13.
(21) 第二節 統計量排序 一、兩獨立母體期望值差 1 2 之假設檢定 在比較攝護腺癌蛋白質質譜資料中的兩兩分類時,我們對於每個 特徵變數先作兩母體之平均數是否有差異之檢定,因此對於每個特徵變數都要作一次 t 檢定才可得出 T 統計量。 首先令 1 為第一類病人的母體平均數, 2 為第二類病人的母體平均數,可得一組. H : 2 0 假設檢定 0 1 ,其中虛無假設是表示兩類別的病人於此特徵變數的平均上是 H 1 : 1 2 0. 政 治 大 在此我們假設母體為常態分配且母體變異數未知不相等的情況下,其檢定統計量為 立. 沒有差異的,而對立假設為兩類別的病人於此特徵變數的平均上是有差異的。. X1 X 2 。因雙尾檢定故我們將每個特徵變數的 T 統計量皆取絕對值以方便特徵變 S1 S 2 n1 n2. ‧ 國. ‧. 數的排序。. 學. T. sit. y. Nat. 二、一因子變異數分析完全隨機化設計檢定. n. al. 比較時,對於每個特徵變數亦可用此方法檢定。. Ch. engchi. er. io. 變異數分析可以檢定兩個母體以上的平均數檢定,故我們在作兩兩分類和四分類的. i Un. v. 一因子變異數分析就是在一因子實驗設計中,隨機分配 n 個實驗單位(總樣本數為 n ) k. 至 k 個不同類別的病患,故將各類別的樣本數記為 n1 , n2 ,, nk ,因此 n n j 。而一因 j 1. 子變異數分析完全隨機化設計模型如下: X jm j jm j jm. j 1,2,, k、m 1,2,n j. X jm:第j類病患中之第m個樣本觀測值 :k個類別的混合總平均 : 此處 j 第j類病患之母體平均 j j :第j類病患之處理效應 jm:第j類病患中第m個樣本之個別誤差效應. 14.
(22) H 0 : 1 2 k 其假設檢定為 ,其中虛無假設是 k 個類別的母體平均無差異,對 H1 : j不全相等 立假設是 k 個類別的母體平均不全相等。最後將處理平方和( SSTR )除以其自由度 k 1 的 MSTR 與誤差平方和( SSE )除以其自由度 n k 的 MSE 相除即產生檢定統計量 F. MSTR 。 MSE. 三、多個獨立母體之無母數檢定 Kruskal-Wallis 檢定(KW 檢定) 此法不像上述的一因子變異數分析須要同質變異數或常態分配等假設,只須將 n 筆 資料混合排序其值的大小,並利用等級來運算即可。它可以檢定母體的中位數或是母體. 政 治 大. 分配的位置。而在本研究中我們對於各特徵變數分別進行 KW 檢定如下:. 立. ‧ 國. 之平均不全相等。而檢定統計量為 H . 學. H 0 : 1 2 k 其中虛無假設為 k 個類別的平均皆相等,對立假設則為 k 個類別之 H1 : j不全相等. ‧. k Rk2 12 3(n 1) , n(n 1) j 1 nk. sit. n. al. er. io. 為 k 1 之分配。. y. Nat. n n1 n2 n: k k組獨立樣本之樣本個數總和 此處 n: ,且此統計量會服從卡方自由度 j 第j組獨立樣本之樣本個數, j 1,2, , k R:第j組獨立樣本所對應觀測值之等級和 j . i Un. Ch. v. engchi 第三節 LARS、Stagewise、LASSO 迴歸模型 此節將 LARS、Stagewise、LASSO 一起討論的原因在於三種方法皆可由 LARS 演 算法求得其迴歸係數估計值。此外,在演算法中如何修改使我們得知 Stagewise 和 LASSO 的估計值結果以及各方法運用至高維度資料時的優缺點也會在此節一併說明。 n. n. n. i 1. i 1. i 1. 首先,將資料標準化後( yi 0 、 xij 0 和 xij2 1 ),我們先定義一個迴歸模 型為 y X nm β ε ,. 15.
(23) y1 y 其中 y 2 , X nm x 1 yn . x2. 1 1 x m , β 2 , ε 2 且假設 ε ~ N (0, I n 2 ) 。 m n . 然後藉由最小平方法在最小化殘差平方和(亦即 S ( βˆ ) y μˆ. 2. n. ( yi ˆ i ) 2 )之目標下 i 1. n. 求解迴歸係數後,可得樣本估計式為 μˆ x j ˆ j Xβˆ 。 i 1. 一、Least Angle Regression(LARS) 我們先由圖 4.2 來簡單解釋 LARS 的概念。若現在只考慮兩個變量 x 1 、 x 2 對 y 的 影響,先將 y 投影至由 x 1 和 x 2 生成的線性空間中之一個向量 y 2 後,計算個變量與 y 的. 政 治 大. 相關係數哪個較大,由圖中可知 y 2 與 x 1 的夾角較小亦即 x 1 與 y 的相關係數較大,則. 立. 我們就沿著 x 1 的方向前進,前進到 μˆ1 處確定可產生一條 x 1 和 x 2 的角平方線向量(就是. ‧ 國. 學. 此向量與 x 1 和 x 2 相關程度一樣)且最後能與 y 2 相交,然後再沿著 u 2 前進,最終可達到. ‧. y 2 即配適完 LARS 迴歸估計式。. n. er. io. sit. y. Nat. al. Ch. engchi. i Un. v. 圖 4.2 二維自變數下的 LARS 資料來源:“Least Angle Regression” by Efron, B., Hastie, T., Johnstone, I. and Tibshirani R., 2003, Annals of Statistics, 32(2), 412.. 當 m 2 時,我們即以數學式來表示。假設有 m 個變數, n 個觀察值, 令 為 1,2,, m的一個子集合,又可稱為活動集合(active set),則可定義. . . 2 X s j x j j,其中s j不是 1即為 1 、 A (1' G11 ) 1/ , 其中G1 X ' X 和. 16.
(24) u X w為角平方向量,其中w AG11 。 若有個當前的 LARS 估計式 μˆ ,則可得到一個當前相關(current correlations)向量. cˆ X ' ( y μˆ ) ,那麼 這個集合就是收集與當前相關取絕對值後最大的第 j 個變數,. . . . 也就是 Cˆ max cˆ j 而 j : cˆ j Cˆ ,此外 s j sgn cˆ j 。則 LARS 下一步的估計式就是 j. 將 μˆ 調整為 μˆ μˆ ˆu ,在此定義一個內積向量 a X 'u ,並由. Cˆ cˆ j Cˆ cˆ j min jc , 來決定 ˆ 值,最後新的 就是 ˆj 。 A a A a j j . . 此種迴歸模型選取變數的方法是由 Efron 等人(2003)提出,其優點除了可產生一個. 政 治 大. 完整分段的線性模型並得出各迴歸係數之變化路徑外,也可以很容易的將其修改為其他. 立. 迴歸模型(如:Stagewise、LASSO),此外若同時有兩個變數與應變數的相關程度差不多. ‧ 國. 學. 時,則它們的迴歸係數應該會以約略相同的速度增加。不過這方法的缺點是因為利用殘 差來疊代,所以若面對高維度資料中變數間容易不為獨立時,會容易影響其模型的選取. ‧. 結果,導致對於變數的排序可能也不太可靠;另外在高維度的資料中,對於模型的配適. y. Nat. sit. 易受訓練集中樣本數的多寡而受限,也就是說若變數個數(m)大於樣本數(n)的話,. n. al. er. io. LARS 迴歸模型中最多就會有 n 個迴歸係數不為零。(Efron 等人,2003;Leng 等人, 2006;Hastie 等人,2009). Ch. engchi. 二、Forward Stagewise Regression. i Un. v. 與向前選取方法類似,差別是在於此方法每一步的移動幅度很小,且較謹慎。簡單 來說由第一個模型 μˆ 0 開始出發,假設 x j 與 μˆ 0 的當前相關最大,亦即有最大的. c j x Tj ( y μˆ 0 ) ,接著取一個 (0 c j ) 可得第二個模型為 μˆ1 μˆ 0 sgn( c j )x j ,然 後重複上述步驟直到模型中所產生的當前殘差與剩餘未被選入模型的變數都不相關即 停止。而修正 LARS 演算法來求解 Stagewise 估計值的方法是先考慮 Stagewise 選取變數 的每個步驟大小 趨近於零,假設已作了 N 個 Stagewise 的步驟,就會產生一些估計式 μˆ 且將 N j 定義成將第 j 個變數選入模型中所須之步驟數,其中 j 1,2,, m ,當 j 時, 17.
(25) N j 0 。定義 P 為 ( N1 , N 2 ,, N m ) / N ,則對於 j 可產生一個新估計式為. μ μˆ N X P ,此外我們也已經知道 LARS 演算法是沿著 μ X w 前進。比較 和可以發現因為 P 是非負的,故 LARS 估計式在 w 中有負值時會與 Stagewise 不吻. 合,這時藉由 C v s j x j Pj , Pj 0 集合使得 μ C 則可以去除這裡的矛盾。 j 此方法的優點是比向前選取法更謹慎的將變數選入模型中,而且可視為單調版的 LASSO,因為當 LARS 或是 LASSO 對於某個變數之迴歸係數有向零變化之趨勢時,此 時的 Stagewise 反而會將其變數之迴歸係數膨脹,並盡可能的將其變數之迴歸係數維持. 政 治 大 縮減變數之效果且在高維度資料中,迴歸模型最多會選入與樣本數(n)相同之變數個 立. 非遞增或非遞減的變化,而此特性會在實証分析的部份有所驗証。而缺點就是比較沒有. ‧. ‧ 國. 三、LASSO. 學. 數。(Efron 等人,2003;Hastie 等人,2009;Leng 等人,2006). Tibshirani(1996)提出關於 LASSO 的文章,LASSO 是一種具有懲罰項的迴歸估計 m. y. sit. j 1. Nat. 式,即限制在 ˆ j t 條件下,能夠最小化殘差平方和的迴歸係數值即為 LASSO 估計. n. al. er. io. 值。由於這種限制條件是不等號的情況,因此必須藉由凸函數最佳化問題中的 KKT 條. i Un. v. 件來求解。另外,LASSO 估計式具有縮減迴歸係數值和變數選取兩種功能,當 t 值小到. Ch. engchi. 一定程度的時候,LASSO 估計式能夠使得某些迴歸係數的估計值為零,因此的確可以 達到選取變數的作用。當 t 不斷增加時,被選入迴歸模型中的變數也會逐漸增多,且當 t 增大到某個值時,所有的變數都會被選入迴歸模型,而這時迴歸模型中的各迴歸係數 值會與利用最小平方法所求出來的迴歸係數值相等。另一方面,也可視它為一種縮減維 度的迴歸方法,因為它可以將不顯著的迴歸係數自動估計為零,因此估計迴歸係數和縮 減維度可同時被完成。 對於 LASSO 估計式,LARS 演算法之修正方式是先假設已經完成若干 LARS 的步驟, 所以這時已經存在一個活動集合 ,且可得知一個估計式 μˆ 。假設 LASSO 的迴歸係數 估計值向量為 βˆ ,而 X w 為在 LARS 中的角平分向量。此處定義一個向量 dˆ ,向量中 18.
(26) 的元素是 j 時其值為 s j w j ,反之為零。因此 μˆ Xβ ( ) ,其中 j ( ) ˆ j dˆ j 。此 時若將 LARS 的角平分向量對應至 LASSO 估計值的路徑上會發現 j ( ) 在 j ˆ j / dˆ j 時變號那麼對於我們已經有的 LASSO 估計值 β ( ) 中的元素會在最小的的那個大於零 的 j 處變號且將其記為 ~ 表示 min ( j ) ,若沒有 j 大於零的話,則將 ~ 記為無窮大。另 j 0. 外,若 ~ 小於 ˆ ,因 j ( ) 之正負號需與 c j ( ) 一致,則對應至 LARS 估計的 j ( ) 就不 會成為一個 LASSO 估計值,所以在此狀況下就不能繼續在 LARS 的步驟上繼續前進, ~ 而解決辦法就是將與 j 相等的 ~ 所對應之 j 從 中刪掉後再繼續進行 LARS 步驟以得. 政 治 大. 出 LASSO 迴歸係數估計值。 (Efron 等人,2003;Leng 等人,2006). 立. ‧ 國. 學. LASSO 這種方法的優點是可同時達到將迴歸係數縮減和變數選取的效果。不過缺 點是面對高維度資料變數個數大於樣本數時,除了在對於兩個具有高度相關的變數選入. ‧. 模型的方式是採「任意」的方式將某個變數加入其模型中,因而影響變數的排序;此外. sit. y. Nat. 最後模型中之非零迴歸係數個數最多並不會超過樣本數(n)。(Hastie 等人,2009). n. al. er. io. 當面對高維度資料時,以上三種迴歸選模方法相同之處是除了都可產生一連串選取. Ch. i Un. v. 變數模型的過程外,每種方法的步驟數雖不相同,而實際上最後會被選入迴歸模型中的. engchi. 變數個數最多都不會超過樣本數(n)。而在高維度資料中,LARS 選取變數的步驟數目 會由樣本數(n)來控制,而 Stagewise 是會根據一個很接近零的 來控制其步驟數目, LASSO 則是依據 t 值的變化來更改其迴歸模型。此外,在選取變數的過程中只有 LARS 必會於每個步驟中將某個變數加入活動集合中(從此再也不會將此變數從活動集合中刪 除),但 Stagewise 以及 LASSO 就可能會出現將某個變數先加入活動集合中,然後經過 若干步驟後又將此變數從活動集合中刪除的情形發生。. 19.
(27) 第四節 Group LASSO 迴歸模型 Yuan 和 Lin(2006)提出 Group LASSO 這種迴歸模型選取方法來改善 LASSO 在面 對高維度資料時,由於變數間可能存在著高度相關而導致變數的選取結果不太可性 的缺點。其想法是來自眾多的自變數中,也許可分成若干群組,而且在變數選取時, 通常是選擇一個群組,而不是一個個別的變數。 接著我們利用數學式來說明此方法,首先假設一基本的迴歸模型如下: Yn1 X nm β m1 n1. (1). 再來若我們欲將 m 個變數分成 J 個組,則可將(1)式變為. 政 治 大. J. Yn1 X j β j n1 j 1. 立. (2). 其中 β j 的向量長度用 p j 來表示,也就是對應至第 j 組中的變數個數。接著對於一個向. ‧ 國. 學. 量 R d ( d 1)且定義一 d d 的對稱正定矩陣為 ,並產生一個矩陣為. ( ')1/ 2 ,在給定 1 ,, J 後即可得到 Group LASSO 的拉格朗日方程式(Lagrange. 2. y. Nat. equation):. sit. . ‧. . n. al. er. io. J J 1 Y X j β j β j , 0 j 2 j 1 j 1. Ch. engchi. i Un. v. (3). 最後只要我們找出能夠使(3)式得到最小值的 βˆ j 即為 Group LASSO 的迴歸係數估計值。 因此 Group LASSO 之迴歸係數估計式如下:. ˆ j (1 . pj Sj. ) S j. 其中 S j X 'j (Y X j )且 j (1' ,, 'j 1 ,0, 'j 1 ,, J' ) 。 而在本研究中,我們的作法是將每個變數都視為一個群組,也就是說若共有 417 個變數, 則我們就分為 417 個群組,然後來得出各分類之判別結果。(Yuan 和 Lin,2006;Friedman 等人,2010). 20.
(28) 第五節 Elastic Net 迴歸模型 這方法的限制式是脊迴歸(Ridge regression)和 LASSO 的融合,由此產生以下 Elastic Net 的拉格朗日方程式: L(1 , 2 , β ) Y Xβ 2 β 2 1 β 1 2. 2. 所以若 1 0 ,則為脊迴歸的拉格朗日方程式;若 2 0 ,則形成 LASSO 的拉格朗日方 程式。(Zou 和 Hastie,2004) 對於此方法的迴歸係數估計值,Park 和 Hastie(2006)提出利用預測-校正 (Predictor-corrector)演算法來求得 Elastic Net 之所有迴歸係數的路徑發展。先假設一組具. 政 治 大. p 有 n 筆樣本和 p 個變數的資料為 (x i , yi : ) x i R、 yi R,i 1,, n,且 Y 為服從指數家. 立. ‧ 國. 學. 族之分配( E (Y ) 、 V Var (Y ) )。設 g ( ) 0 x ' 而 Y 的密度函數為. L( y; , ) exp ( y b( )) / a( ) c( y, ),其中 a(.) 、b(.) 和 c(.) 會依 Y 分配不同而改變。. i 1. ‧. n. l ( , ) yi ( ) i b( ( ) i ) . 1. (4). Nat. sit. y. 在此若已知散佈變數 為已知,欲找出能夠極大化概似函數的 也就是相當於尋求使第. n. al. er. io. (4)式最小的 β ( 0 , ' )' 。假設 β 不為零,在此我們令一函數 H 為:. Ch. 0 l ' H (β , ) X W (y μ ) sgn β μ β . engchi. i Un. v. 其中. 1 x11 1 x 21 X 1 xn1. . 1 l 2 V1 ( )1 0 0 x1 p 1 l 2 x2 p 0 V2 ( ) 2 ,W , 0 xnp l n( p 1) 0 0 Vn1 ( ) 2n nn . l ( y1 1 )( μ )1 l (y μ ) 。 μ l ( yn n )( ) n μ n1 21. (5).
(29) 每給定一個不同的 值,則第(5)式就需重新計算迴歸係數值。因此預測-校正演算法的 目標就是在 從最大值( max )往零移動的過程中記錄迴歸係數估計值的變化路徑。而預 測校正演算法大致可分為以下四個部份: 1.預測步驟(Predictor step) 假設在第 k 個預測步驟,則 β (k 1 ) 可藉由以下公式概略計算得來:. βˆ k βˆ k (k 1 k ). 0 β βˆ k (k 1 k )(X ' W k X ) ' sgn ˆ k β . 其中W k 和 X 分別表示當前的權重矩陣以及在當前活動集合中的共變量矩陣,且此處 β 是只有包含當前迴歸係數被估計成非零的迴歸係數矩陣。 2.校正步驟(Corrector step). 立. 政 治 大. 利用 βˆ k 1 為初始值,來找尋可以最小化 l ( β , k 1 ) l(,k 1) 的 β 。基本上, βˆ k 通常. ‧ 國. 學. 會很接近 βˆ k 1 。在此步驟中不僅可找給定一個 下的精確解,也提供了之後 β 在預測步. ‧. 驟中的方向。. y. Nat. 3.活動集合(Active set). er. io. sit. 一開始的活動集合中只有截距項,但之後在作每一個修正步驟時,若某個原本不在 活動集合中的變數滿足以下第(6)式時,就放入活動集合中。且一直重複校正步驟直到活. al. n. iv n C 動集合不再擴大,然後我們再將原本在活動集合中的變數代入第(7)式,若發現某變數之 hengchi U 迴歸估計值為零,則再將此變數從活動集合中剔除。. x 'jW ( y μ ). ,j c j . ˆ j 0,j \ j. (6) (7). 4.步驟長度(Step length) 就是估計前一個步驟與後一個步驟的距離(長度),也就是 k k 1 。假設由一個修 正步驟中可得到 y 的估計 μˆ 而第(8)式即表示 μˆ 所對應的權重相關係數矩陣。 ' ˆ cˆ X W ( y μˆ ). . (8). 22.
(30) 然後因在下一個預測步驟中會擴展 βˆ ,故當前的相關係數矩陣也會改變。而第(9)式為下 降一單位的 時其相關矩陣的變動情形。. 0. ' ˆ c (h) cˆ ha cˆ hX W X ( X ' WˆX ) 1 sgn ˆ β . (9). 再來我們利用第(10)式可解得一個估計步驟長度的估計值即第(11)式。 c j (h) cˆ j ha j h , j c. (10). cˆ j cˆ j h minc , j 1 a 1 aj j . (11). 那麼將 Elastic Net 運用至預測-校正運算法中來求取迴歸係數的路徑方式就是將第. 政 治 大. (12)式中的 2 固定成某個很小的正數且 1 (0, ) 。. 立. ‧ 國. β. 2. . 2 2 2. (12). 學. . βˆ (1 ) arg min log L( y ; β ) 1 1 . ' 假設 β 皆不為零且 X 沒有行獨立時,則 H (β , ) / β X WX 將會形成不可逆矩陣,然. ‧. 而再藉由增加一個二次懲罰項的話,則我們可將 H 重新定義為第(13)式。. er. io. sit. y. Nat. 0 0 ~ ' H ( β , 1 , 2 ) X W (y μ ) 1 sgn 2 (13) β β μ ~ H 此時對於任何的 2 0 , 就是個可逆矩陣。因此對於自變數間有高度相關的資料之 β. al. n. iv n C 處理方式即固定 為一個小的值, h然後將 e n g c由它的最大值往零移動來得出迴歸係數的整 hi U 2. 1. 個估計路徑。因此,不同於 LASSO 迴歸模型。(Park 和 Hastie,2006). 此迴歸方法除了考慮到變數的縮減之外也考慮到群組效果(grouping effect),也就是 說它具有群組選取(group selection)的能力。也就是說遇到有兩個具有高度相關的變數 時,此方法會同時將這兩個變數選入模型中,這樣一來不僅沒有使其模型的預測準確度 消失同時也達到縮減變數的效果。因此在處理高維度資料時,Elastic Net 所選取到的變 數與 LASSO 相比之下也較可靠,不過其演算法確實也複雜許多。(Zou 和 Hastie,2004). 23.
(31) 第六節 支持向量機 SVM 支持向量機的英文全名為 Support Vector Machine,簡稱為 SVM,是一種基於統計 學習理論(statistical learning theory)基礎的學習機器。其概念為對於一群資料而言,我們 會希望依據資料的某些特性將這群資料分為兩群。以二維的例子來說,如圖 4.3,我們 希望能找出一條線將黑點和白點分開,且這條線距離這兩個類別的邊際(margin)越大越 好,才能夠很明確的分辦某個點是屬於那個類別,否則在計算上容易因精確度的問題而 產生誤差。. H1. H H2. 立. 政 治 大. ‧. ‧ 國. 學 margin . n. al. er. io. sit. y. Nat 圖 4.3 SVM 原理示意圖. 2 w. Ch. engchi. i Un. v. 因此以兩種分類的 SVM 來說,假設訓練資料為 x i , yi 其中 i 1,, l、xi R d、yi 1,1且 l 為訓練資料個數、d 是維度。而在此分類超平面上的 x. 需滿足 w T x b 0 ,其中 w Rl 是訓練樣本中係數向量, b 是截距常數。若我們令 f (x ) w T x b 為決策函數,那麼將測試資料代入決策函數後即可根據決策函數之值來. 加以分類,也就是希望找到一條線 f (x ) w T x b 使所有 yi 1 的點落在 f (x ) 0 的這 一邊,且使所有 yi 1 的點落在 f (x ) 0 這一邊,這樣就可根據 f ( x ) 的正負號來區分 點是屬於這兩個分類的哪一類,故此超平面稱為分類超平面(separating hyperplane),而 24.
(32) 距離兩邊邊界最大的距離且到各類最近點的距離相等時稱為最佳分類超平面,而此處的 距離相等是指此分類超平面到兩邊界的距離皆為 1 / w ,因此為了求取最佳分類超平面 就是要求 w 之最小值。 此外,由於一般的分類平面方程式為 w T (x ) b ,而 是指將樣本空間轉換到另一 個高維度空間的對應函數。當 ( x i ) x i 時,即表示在原本空間中可找到一個平面將資 料分類並獲得最小的分類錯誤以及不同類別之間的最大間隔,我們就稱此為「線性可分 問題」。 若遇到線性不可分的問題時,則必須將原始向量空間對應到較高維度的空間,來尋. 政 治 大 l. 找分類的超平面,此時分類平面方程式會變成 i yi ( x i ) ( x i ) b ,其中的轉換核心. 立. i 1. l. (x i ) (x i ) 以 K ( x i , x ) 取代後,分類平面方程式即成為 i yi K ( x i , x ) b ,通常使用的. ‧ 國. 學. i 1. 轉換核心有以下三種: 線性(Linear): K (x i , x j ) x iT x j. . 半徑基底函數(Radial basis function, RBF): e. . 多項式(Polynomial): ( (x iT x j ) ) d. io. sit. 2. er. Nat. x i x. y. ‧. . al. n. iv n C 其中 , d , R 是轉換核心的參數,其分類規則也可以表示成 sgn( y K ( x , x ) b) 。 hengchi U l. i 1. 25. i. i. i.
(33) 第五章 實証分析 本研究是利用事前處理的攝護腺癌之蛋白質質譜資料來進行判別兩兩分類以及四 分類的分析,而我們的目的最主要是探討各(具有懲罰項)迴歸模型在各分類之判別結果 的表現情形。此部份我們會先利用五種「選入迴歸模型之順序排序」的特徵變數選取結 果與三種「統計量排序」和「分錯率排序」的特徵變數選取結果作比較,然後再將五種 迴歸模型中表現最突出者與 Adam 等人(2002)以及陳詩佳(2007)之判別結果作比較。 首先分別將四種類別,正常、良性腫瘤、癌症早期以及癌症晚期分為訓練資料和測 試資料兩個部份,而表 5.1 是各分類之訓練資料和測試資料的筆數。. 立. 受測者. 正常. NO. 82. 164. 110. 54. 良性腫瘤. BPH. 77. 154. 102. 52. 84. 168. 112. 83. 166. CCD. n. al. Ch. engchi. sit. io. 癌症晚期. CAB. er. 癌症早期. 資料筆數 訓練資料 測試資料. y. 簡稱. Nat. 類別. ‧. 學. ‧ 國. 表 5.1 訓練資料和測試資料筆數. 政 治 大. i Un. v. 110. 56 56. 再來由於我們要比較兩兩分類以及四分類的判別結果,故我們將正常、良性腫瘤、 癌症早期以及癌症晚期四種類別的資料合併為「正常 vs.良性腫瘤」(NO vs. BPH)、「正 常 vs.癌症早期」(NO vs. CAB)、「正常 vs.癌症晚期」(NO vs. CCD)、「良性腫瘤 vs.癌症 早期」(BPH vs. CAB)、「良性腫瘤 vs.癌症晚期」(BPH vs. CCD)、「癌症早期 vs.癌症晚 期」(CAB vs. CCD)以及、 「正常 vs.良性腫瘤 vs.癌症早期 vs.癌症晚期」(四分類)這七個 類別資料檔。其實我們從這七個資料中會發現某些特徵變數在其資料中的觀測值皆為 零,我們會認為此特徵變數在其資料中是無意義的,於是將它刪除再對資料進行分析, 因此表 5.2 是分別在七個類別資料檔中,刪除所有受測者測得強度為零的那些特徵變數 後所剩餘的特徵變數個數。 26.
(34) 表 5.2 七種合併資料刪除零後之特徵變數個數 合併資料. 刪除零後之特徵變數個數. NO vs. BPH. 470. NO vs. CAB. 555. NO vs. CCD. 634. BPH vs. CAB. 555. BPH vs. CCD. 629. CAB vs. CCD. 678. 四分類. 立. 政 治 740 大. ‧ 國. 學. 第一節 R 函數之設定. ‧. 本篇研究中當我們在分析兩兩分類以及四分類時,是利用 R package 中的“LARS” 函數來配適 LARS、Stagewise 以及 LASSO 迴歸模型,因此需在”LARS”函數中作設定,. y. Nat. io. sit. 而以下為我們設定的內容:. n. al. er. 1. 有截矩項的存在(intercept=TRUE)。. Ch. 2. 資料為未標準化資料(normalize=FALSE)。. engchi. i Un. v. 3. 型態的部份分別設定成 type=lar、forward.stagewise 和 lasso。 另外,我們也利用 R package “glmpath”函數來配適 Elastic Net 迴歸模型,其設定內容 如下: 1. 將 y 的分配設為二項分配(family=binomial(link = "logit")) 2. 資料為未標準化資料(standardize=FALSE) 上述兩點是對於每組訓練集都採用的設定。但此函數較特別的是,有兩個設定值 “min.lambda“和“relax.lambda“必須依據各組訓練資料而作改變。所謂的“min.lambda“表 示我們可以決定第四章第五節中所提到的 1 之下界,由於每一組訓練資料的變數個數皆 比觀測個數還要多,因此將此值設為 1e 6 或是其他不為零的值較適當。而“relax.lambda 27.
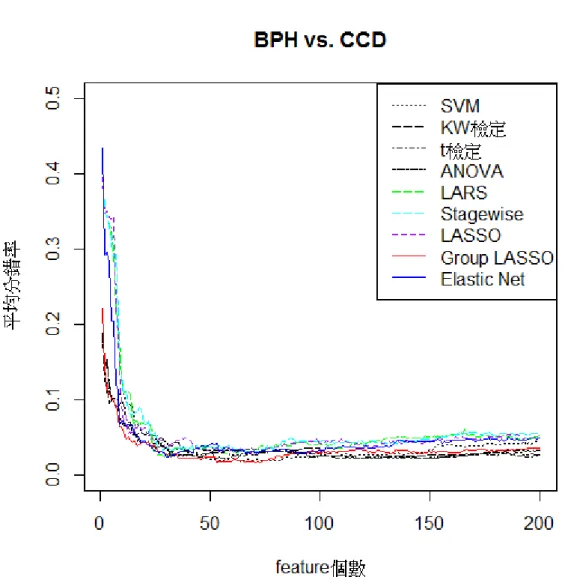
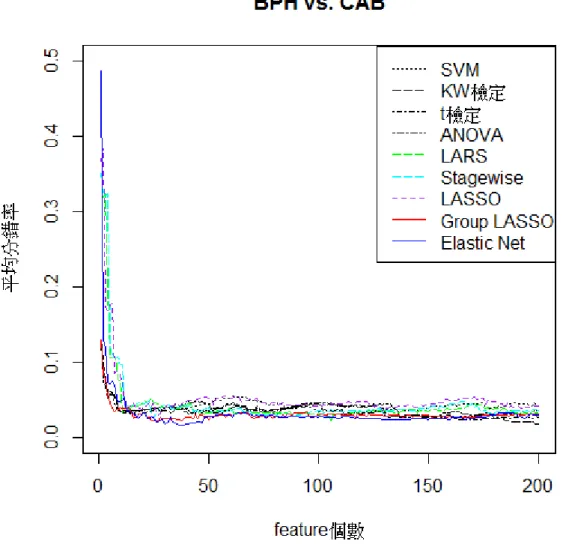
(35) “的設定會與某個變數在哪一步被選入迴歸模型有相關,因此若某個變數滿足 l ' (β ) 1 (1 ralax.lamb da ) 就將其加入模型,此處 n. l ( β ) yi ( β )i b( ( β )i ) 1 β 1 。而“relax.lambda“的預設是為 1e 8 。若超過 20 個 i 1. 步驟仍然無變數可加入活動集合的話,則可將其值增加到 1e 7 或 1e 6 。其實有時藉由對 於這個值的調整,才可免除在執行 R 軟體時會遇到一些的問題,如:出現無法收斂的警 告標語或是 R 軟體呈現無法回應的狀態。. 第二節 探討兩兩分類之分錯率結果 在此我們先藉由圖 5.1、圖 5.2、圖 5.3、圖 5.4、圖 5.5 以及圖 5.6 來討論九種特徵. 政 治 大. 選取方法於各兩兩分類下的分錯率表現。. 立. 圖 5.1 為判別正常和良性腫瘤的分錯率趨勢圖,可以發現 LARS 和 Stagewise 的分. ‧ 國. 學. 錯率的走向很一致,有同時上升同時下降的現象。另外,當特徵變數組合數為 1 或 2 個. ‧. 時,Elastic Net 所得之分錯率比其他特徵選取方法高很多。此外利用 Group LASSO 所選. n. al. er. io. sit. y. Nat. 取的前兩百名特徵變數不論其組合數為何所得之分錯率皆比其他特徵選取方法來得低。. Ch. engchi. i Un. v. 圖 5.1 各特徵選取方法下判別正常與良性腫瘤之分錯率趨勢圖 28.
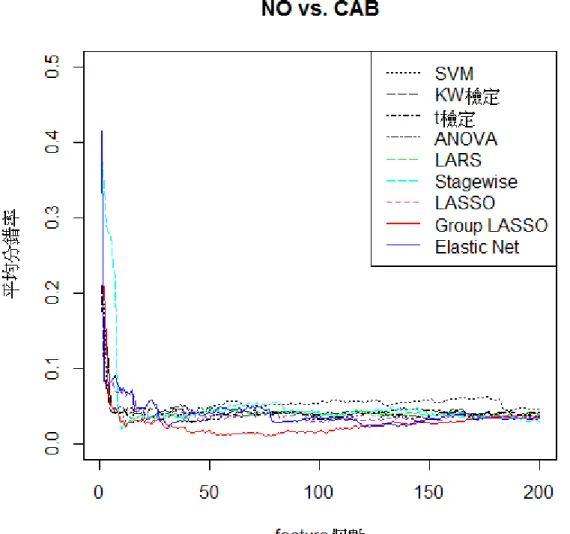
(36) 圖 5.2 為判別正常和癌症早期的分錯率趨勢圖,發現 SVM 之「分錯率排序」方法 在組合數超過 84 個以後其分錯率的表現與其他相較之下最不理想。 而 LARS 和 Stagewise 在組合數 1 至 15 個時,其分錯率完全相同。而 Group LASSO 在組合數 21 至 120 個時之分錯率比其他者更優。. 立. 政 治 大. ‧. ‧ 國. 學. n. er. io. sit. y. Nat. al. Ch. engchi. i Un. v. 圖 5.2 各特徵選取方法下判別正常與癌症早期之分錯率趨勢圖. 29.
(37) 圖 5.3 為判別正常和癌症晚期的分錯率趨勢圖,發現在組合數大約介於 1 至 60 個之 間時,不論何種特徵選取方法其分錯率有很明顯的波動,其中利用 LARS 和 Stagewise 之特徵選取方法在組合數由 10 增加為 11 個時的分錯率差異極大。另外,藉由 KW 檢定 的「統計量排序」方法在組合數介於 39 至 70 個間的分錯率表現最不理想。. 立. 政 治 大. ‧. ‧ 國. 學. n. er. io. sit. y. Nat. al. Ch. engchi. i Un. v. 圖 5.3 各特徵選取方法下判別正常與癌症晚期之分錯率趨勢圖. 30.
(38) 圖 5.4 為判別良性腫瘤和癌症早期的分錯率趨勢圖,可發現當組合數為 1 個時, Elastic Net 之分錯率將近百分之五十,但組合數增為 2 個時其分錯率馬上降為百分之十 三,此外 LARS、 Stagewise 和 LASSO 在組合數為 1 至 12 個間其分錯率快速下降,而 LASSO 在組合數為 33 至 60 個之間和組合數超過 108 個時其分錯率較其他方法高。. 立. 政 治 大. ‧. ‧ 國. 學. n. er. io. sit. y. Nat. al. Ch. engchi. i Un. v. 圖 5.4 各特徵選取方法下判別良性腫瘤與癌症早期之分錯率趨勢圖. 31.
(39) 圖 5.5 為判別良性腫瘤和癌症晚期的分錯率趨勢圖,當組合數為 1 個時,LARS、 Stagewise、LASSO 以及 Elastic Net 的分錯率最高,不過當組合數到達 13 個時其分錯率 已與其他方法相近,然而上述方法在組合數大約到達 140 個以後,其分錯率有明顯上升 的跡象。. 立. 政 治 大. ‧. ‧ 國. 學. n. er. io. sit. y. Nat. al. Ch. engchi. i Un. v. 圖 5.5 各特徵選取方法下判別良性腫瘤與癌症晚期之分錯率趨勢圖. 32.
(40) 圖 5.6 判別癌症早期和癌症晚期的分錯率趨勢圖。普遍來看,五種「迴歸模型選取 變數排序之方法」對於此分類的分錯率結果似乎都較「統計量排序」以及「分錯率排序」 還要理想,而且可以很顯然的可以發現當組合數到達 13 個以後,Group LASSO 的分錯 率的表現明顯的比其他特徵選取方法來得好。. 立. 政 治 大. ‧. ‧ 國. 學. n. er. io. sit. y. Nat. al. Ch. engchi. i Un. v. 圖 5.6 各特徵選取方法下判別癌症早期與癌症晚期之分錯率趨勢圖. 33.
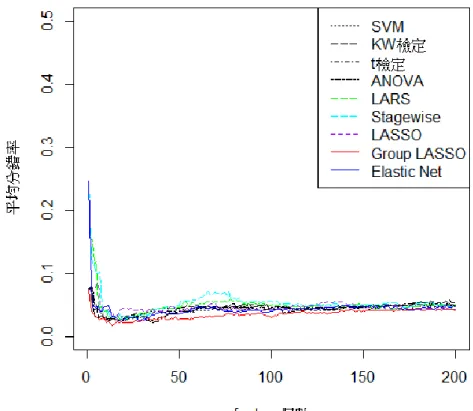
(41) 再來我們探討各特徵變數選取方式在六種兩兩分類上的結果。由於黃仁澤(2005)在 其文章中提及利用 p 值排序的特徵選取方法,於是在本研究的附錄一中也附上在兩兩分 類中利用 p 值排序的分錯率結果。而本研究的目的最終是要探討利用各迴歸模型選取變 數的方法是否能比其他排序變數的方法在分錯率的表現上還要好。 表 5.3 中所呈現的是九種特徵選取方法在六個兩兩分類之最小分錯率以及組合數, 其中我們最小分錯率的計算單位是百分比,而括號內的數值是為分錯率的標準差。那麼 我們由表中可知 Group LASSO 特徵變數選取方法在分類「NO vs. BPH」、「NO vs. CAB」、「BPH vs. CCD」以及「CAB vs. CCD」時效果最好,分別可得到最小的分錯率 為 1.82%、1.10%、1.65%和 9.09%,而其組合數分別為 14、76、53 和 47 個特徵變數。. 政 治 大. 而 Elastic Net 是在「NO vs. CCD」以及「BPH vs. CAB」的分類中表現最佳,分別可得. 立. 最小的分錯率為 1.45%以及 1.68%,而組合數分別為 30 和 37 個特徵變數。. ‧ 國. 學. 若對於各兩兩分類的判別結果僅比較五種迴歸模型選取變數的方法時,LASSO 的. ‧. 表現在各兩兩分類中之分錯率幾乎都不盡理想。而 Group LASSO 在「NO vs. BPH」 、 「NO. y. Nat. vs. CAB」 、 「BPH vs. CCD」以及「CAB vs. CCD」這四種兩兩分類之判別結果表現最好,. er. io. sit. 而 Elastic Net 則在「NO vs. CCD」以及「BPH vs. CAB」中之分錯率表現最佳,那 Elastic Net 之所以能夠在上述兩個分類中勝過 Group LASSO 我認為是因為由圖 5.3 和圖 5.4 中. al. n. iv n C 我們可以發現此兩個圖形中之紅線(表示 LASSO)和藍線(表示 Elastic Net)的分錯 h e n gGroup chi U 率趨勢一路糾纏,有時紅線勝過藍線,有時藍線勝過紅線。不像其他分類,會在到達某 一組合數後,表示 Group LASSO 的紅線就會開始明顯的維持在藍線下方。不過我們也 可發現這兩種方法最大的共同點就是因為具備群集選擇的功能,故將其運用至變數間具 有高度相關的資料中時其變數選取的結果可能較能夠讓人信任。. 34.
數據
+2
相關文件
從思維的基本成分方面對數學思維進行分類, 有數學形象思維; 數學邏輯思維; 數學直覺 思維三大類。 在認識數學規律、 解決數學問題的過程中,
對任意連續函數,每個小區間上的取樣點 x 都選擇在函數最 大值與最小值發生的點。如下圖,淺色方塊的高度都挑選小
甄選類別 甄選名額 缺額性質 聘期
※概略說明與被保險人開始縮減工資工時之實施日期,及其所採取
甄選類別 甄選名額 缺額性質 聘期
中學中國文學課程分為必修和選修兩部分。必修部分的學習材料
分類法,以此分類法評價高中數學教師的數學教學知識,探討其所展現的 SOTO 認知層次及其 發展的主要特徵。本研究採用質為主、量為輔的個案研究法,並參照自 Learning
學校管理層有責任了解和監察 教師選取或編訂 的 教材的內容和質素 ,並要考慮到教材是否切